近段时间,老王在研究RNA与蛋白相互作用的过程中,发现了一个近两年来颇受关注的实验技术——RIP(RNA Binding Protein Immunoprecipitation,RNA结合蛋白免疫沉淀),顾名思义就是研究细胞内RNA与蛋白结合情况的技术。
RIP实验的目的是分析与目标蛋白结合的RNA,原理则是运用针对目标蛋白的抗体把相应的RNA-蛋白复合物沉淀下来,经过分离纯化并对结合在复合物上的RNA进行测序或PCR分析。
2016年9月23日,意大利Maria R. Matarazzo教授在Methods in Molecular Biology杂志介绍了RIP实验的经典操作方法【1】(觉得英文原文阅读起来比较麻烦的可以到吉赛生物的circRNA公众号搜“RIP实验方法”,即可获得超详细版中文解读)。文中介绍的RIP实验操作流程如下图所示:

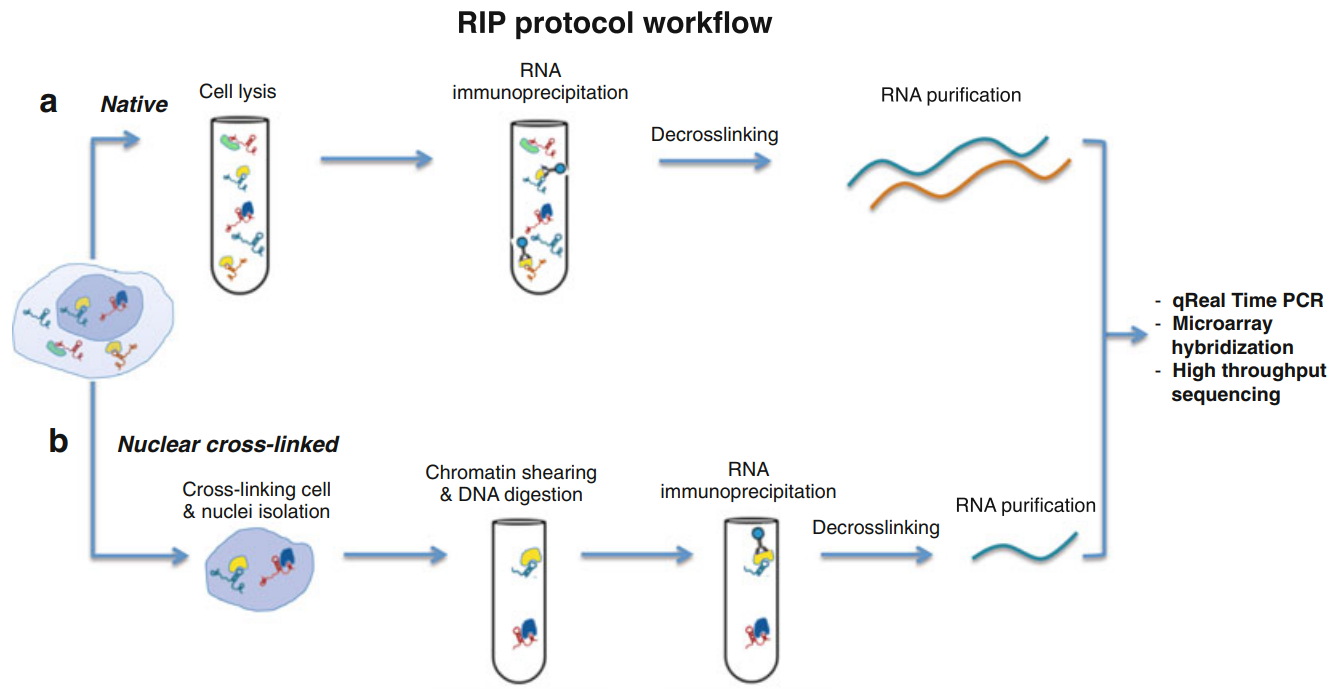
图1 经典RIP实验操作流程(来自【1】)
对于完整的RIP实验流程,老王也画了个中文版简图可以帮助大家更好的理解RIP实验操作。
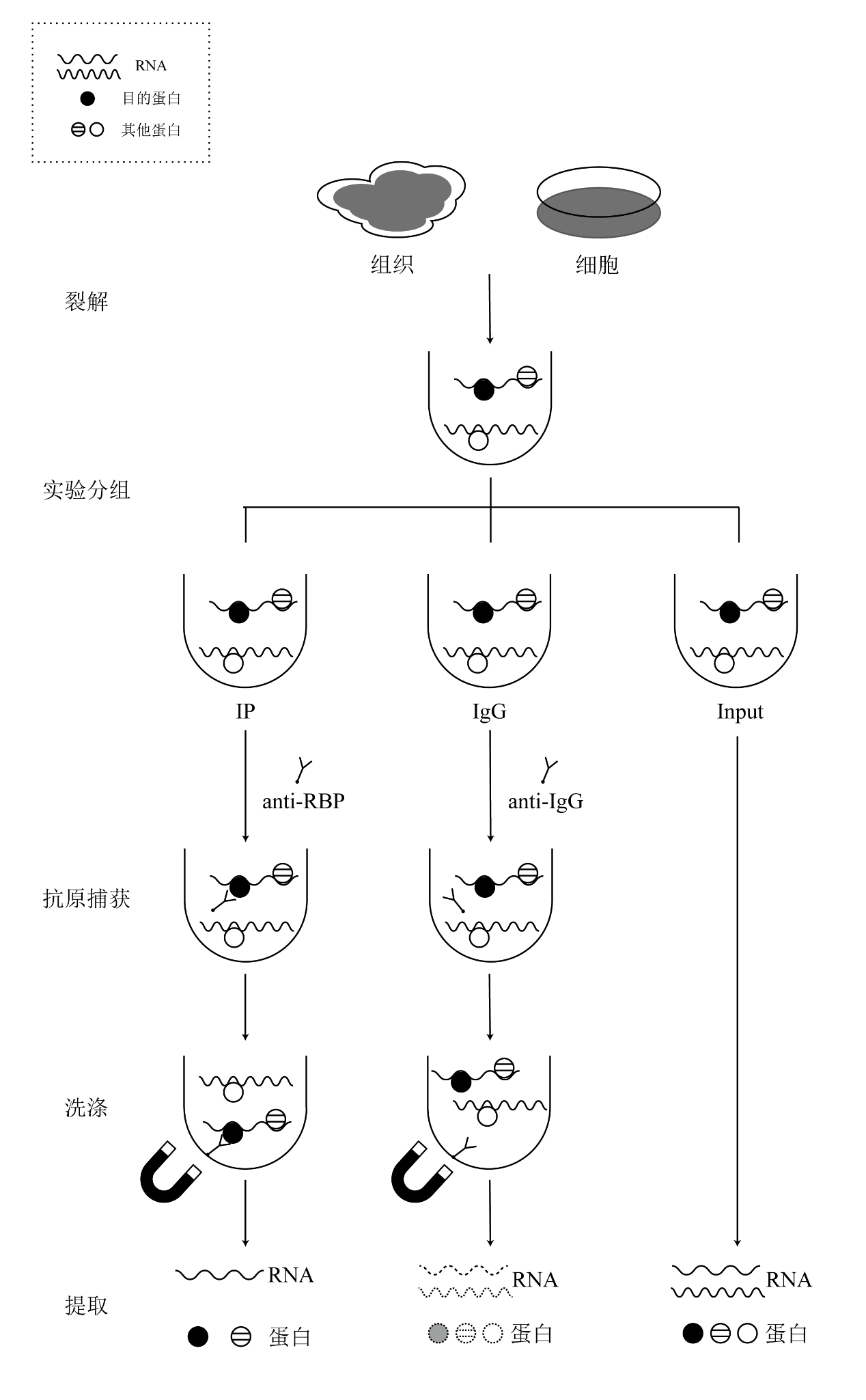
图2 RIP原理流程图
而RIP实验有哪些应用呢?大家都知道RNA是一种不稳定的生物大分子,绝大多数RNA都需要与特定的RNA结合蛋白形成RNA-蛋白复合物才能稳定存在于细胞中。RNA-蛋白复合物驱动了几乎所有细胞过程的基因表达的转录后调控,包括剪接(splicing)、出核转运(nuclear export)、mRNA稳定性以及蛋白转译过程。因此,对基因调控的了解就有赖于确定这些过程中RNA的结合的变化,RIP技术应运而生,可应用于研究miRNA调节靶点、RNA与RNP(RNA结合蛋白)的互作关系等。
案例1. RIP用于研究circRNA与蛋白互作

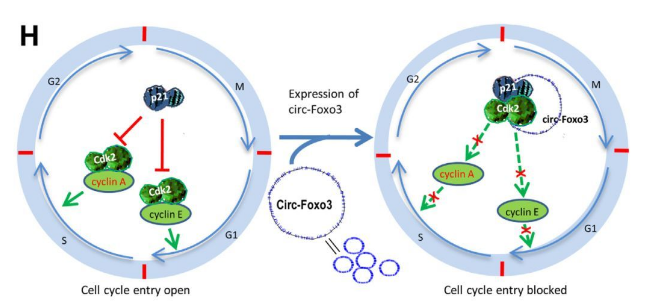
图3 circ-Foxo3在细胞周期进程中的作用(来自【2】)
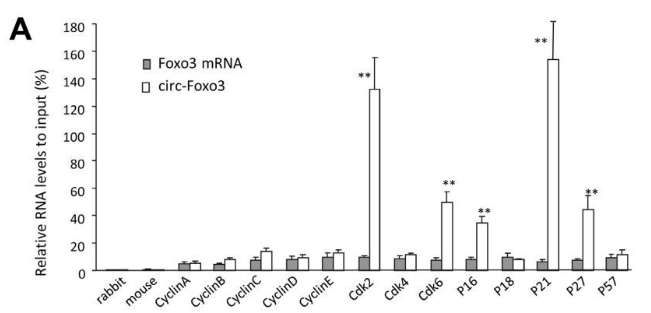
图4 RIP和qPCR检测,揭示了circ-Foxo3与蛋白p21和CDK2的关系(来自【2】)
在本研究中,通过p21和CDK2等多种蛋白抗体的RIP实验验证了circ-Foxo3通过与蛋白p21和CDK2的结合【2】。
案例2. RIP用于研究lncRNA与蛋白互作

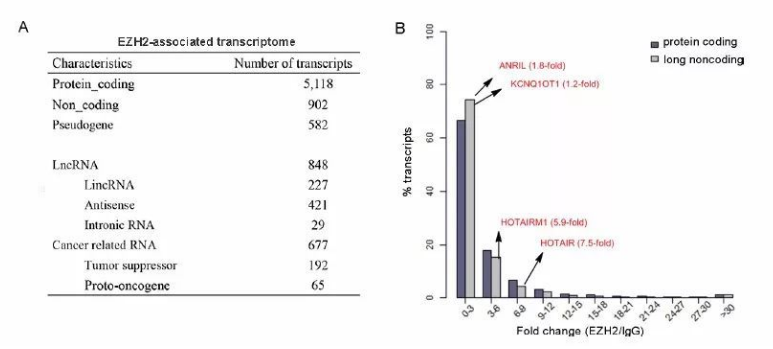
图5 lncRNA和编码RNA与EZH2结合能力的比较(来自【3】)
在本研究中,使用EZH2特异性抗体对人胃癌细胞系MKN45和AGS,以及对照细胞系GES-1进行RIP-seq,分析了8256个与EZH2相关的RNAs,包括编码和非编码RNAs,其中MALAT1在MKN45细胞系中高表达。并且发现MALAT1通过和EZH2相互作用,抑制原钙黏蛋白因子10(PCDH10)的表达,从而促进胃癌细胞的侵袭和转移【3】。
案例3. RIP用于研究miRNA与蛋白互作

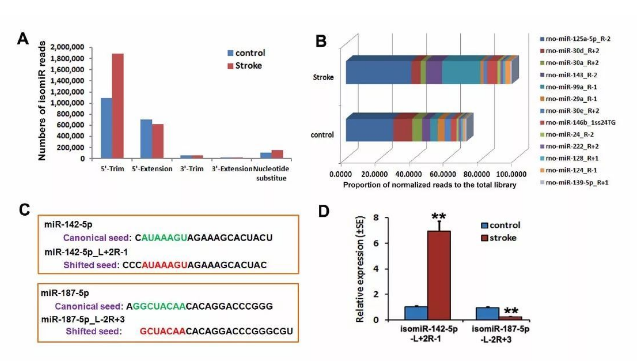
图6 缺铁性和非缺血性NPCs中isomiRs分析(来自【4】)
在本研究中,利用Ago2 RIP-seq,分析非缺血性和缺血性成年大鼠侧脑室下区(SVZ)的神经祖细胞(NPCs)miRNAs的表达情况【4】。
结果显示,相较于非缺血性NPCs,中风改变了NPCs中Ago2相关的miRNAs表达,缺血性NPCs中的isomiRsreads数增加,其中5’末端添加核苷酸的isomiR-142-5p_L+2R-1显著上调,5’末端缺失核苷酸的isomiR-187-5p_L-2R+3显著下调【4】。
说到RIP,就不得不提一下它的搭档RNA pulldown技术。我们知道,RIP是从蛋白出发研究蛋白RNA相互作用的技术,通过已知蛋白去验证可能有相互作用RNA分子;而RNA pulldown则是从RNA出发研究蛋白与RNA相互作用的技术,通过已知RNA去拉蛋白,推测可能与已知RNA结合的蛋白分子,后续可通过WB验证相互作用。所以RIP和RNA pulldown两种技术可以从正反双向进行验证,能更准确的反映RNA与蛋白的相互作用。
案例:综合利用RNA pulldown和RIP用于研究lncRNA与蛋白相互作用


本研究首先通过RNA Pulldown和后续质谱实验找到了与lncRNA MEG3结合的蛋白PTBP1,然后再通过PTBP1的抗体的RIP实验反向验证了lncRNA MEG3与PTBP1蛋白的结合【5】。
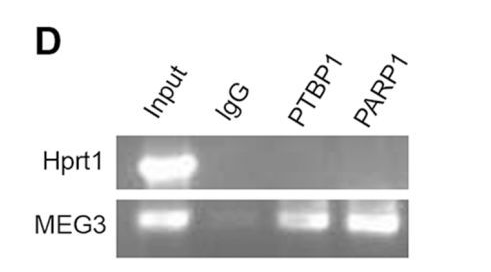
图7 RIP揭示HEK293T细胞中PTBP1蛋白与MEG3 RNA的相互作用(来自【5】)
上图中MEG3和PTBP1是要研究的分子,Hprt1是阴性对照。Input是未经IP的对照,IgG是用来判断非特异性扩增的对照,PART1是阳性对照(已知与MEG3结合的蛋白)【5】。
脑洞:
有意思的是,我们在研究中发现,同一个RNA,无论是microRNA还是lncRNA或者circRNA,他们的靶蛋白很可能不仅仅只有一个,运用RIP实验技术,得到RNA-蛋白复合物,分离复合物中的RNA,经过逆转录或构建cDNA 文库,最后利用基因特异性分析技术(PCR、qRT-PCR)或高通量分析技术(高通量测序、基因芯片),分析复合物中RNA的类型及多少;而分离RNA-蛋白复合物中的蛋白,通过质谱或蛋白组测序等方法,还可以用来研究与目标蛋白结合的RNA的其他互作蛋白(蛋白研究非RIP的初始目的,感兴趣的可以充分发挥想象力,惊和喜随时在等你)。
老王看了市场上现在的各种RIP试剂盒,发现绝大部分都只是提供提取纯化RNA,提取蛋白的很少有涉及,这可能是大家对RIP用抗体特异性比较信任,默认实验拉下来的蛋白就是需要的目的蛋白。但从科研严谨性上来说,不提取蛋白做WB等验证,怎么就敢确认拉下来的是你的目标蛋白呢?
吉赛生物强力推出新款RIP试剂盒,能同时分离提取RNA-蛋白复合物中的RNA和蛋白,高质量的RNA可以用于下游的qRT-PCR、数字PCR以及高通量测序(NGS)等实验;提取的蛋白,可广泛用于PAGE凝胶电泳、Western Blot、ELISA检测、质谱或蛋白组测序等实验,一方面验证了RIP拉下来的目标蛋白,另一方面可以研究与目标蛋白结合的RNA的其他相互作用蛋白,满足大家的脑洞需求。
研发已经成功,即将投产,敬请期待。
参考文献:
1. RIP: RNA Immunoprecipitation. Methods Mol Biol, 2016. 1480: p. 73-86.
2. Foxo3 circular RNA retards cell cycle progression via forming ternary complexes with p21 and CDK2.Nucleic Acids Res. 2016 Apr 07;44(6)
3. MALAT1 long ncRNA promotes gastric cancer metastasis by suppressing PCDH10.Oncotarget. 2016 Mar 15; 7(11): 12693–12703.
4. Identification of miRNomes associated with adult neurogenesis after stroke using Argonaute 2-based RNA sequencing.RNA Biol. 2017; 14(5): 488–499.
5. Long noncoding RNA MEG3 induces cholestatic liver injury by interaction withPTBP1 to facilitate shp mRNA decay.Hepatology 2017 02;65(2)
.png)