概述
可能大部分环状RNA的研究者都没想到,就算根据标准的环状RNA检验方法,环状RNA仍然会上演“真假美猴王”的剧情。特别是这里的六耳猕猴也非常能打~
前不久才在一篇文章中提到过环状RNA分子识别与验证的标准指南……(lll¬ω¬)
Nature Methods丨大咖组团发表环状RNA检测工具的评估报告以及使用指南
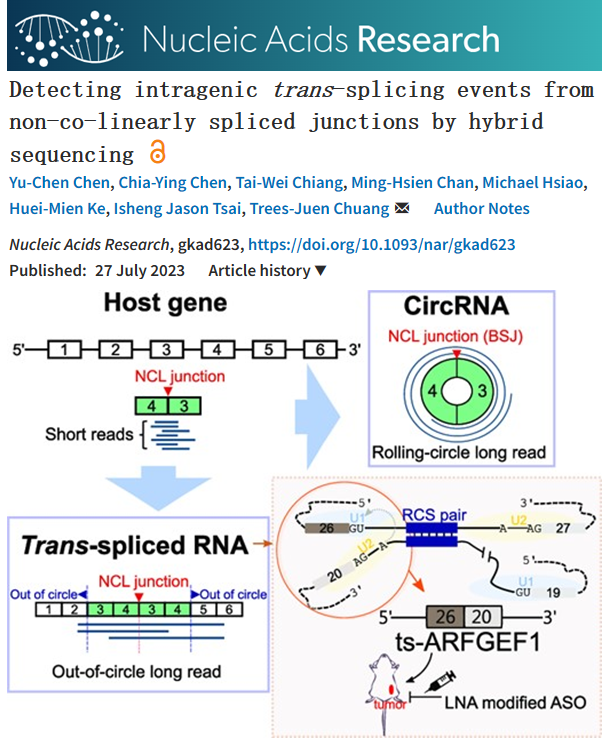
2023年7月27日,台湾省中央研究院的庄树谆教授团队在Nucleic Acids Research(IF=14.9)上发表了一篇关于ts-RNA的文章【Detecting intragenic trans-splicing events from non-co-linearly spliced junctions by hybrid sequencing】。
作者通过整合二代和三代测序数据开发了一个识别基因内ts-RNA的生信工具NCLscan-hybrid,提出了用out-of-circle和rolling-circle特征来区分基因内ts-RNA和circRNA;通过该工具,作者发现环状RNA公共数据库中一些环状RNA可能是ts-RNA,二者都具有相同的序列特征BSJ。为此,作者设计了一系列的实验步骤证实了ts-RNA的存在,排除了环状RNA的可能。此外,作者用 CRISPR/Cas9探索了ts-RNA生物合成的机制,发现和环状RNA一样,侧翼内含子中的互补序列能够促进ts-RNA的形成。最后,作者对其中一个 tsRNA分子ts-ARFGEF1进行了体外和体能的功能实验,发现该分子在乳腺癌细胞中通过影响 PERK/eIF2a/ATF4/CHOP信号通路在p53介导的凋亡过程中扮演着重要角色。
基本概念
要看懂这篇文章,我们首先要了解一些基本概念:
1、共线性与非共线性
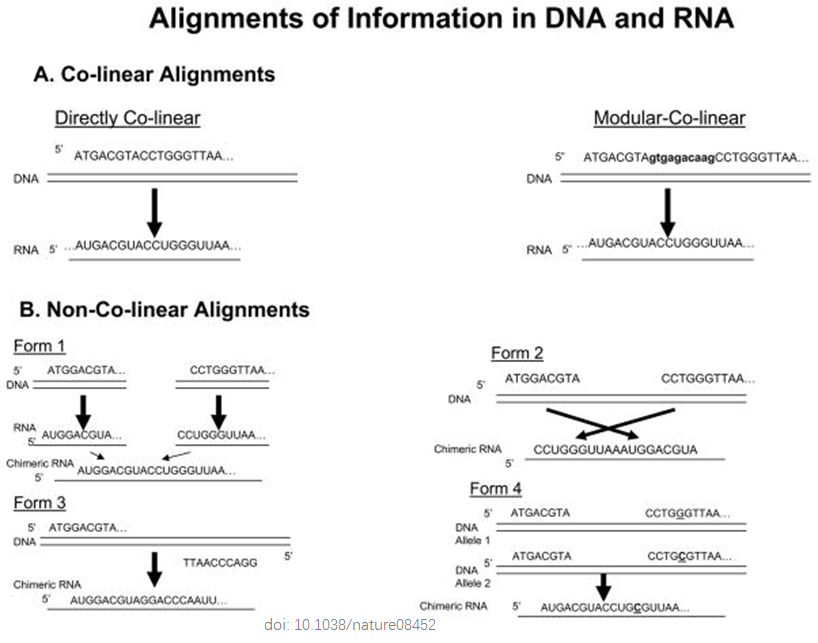 2009年Nature发表的Implications of chimeric non-collinear transcripts文章给出了转录中共线性(co-linear)以及非共线性(non-co-linear,NCL)的定义。
2009年Nature发表的Implications of chimeric non-collinear transcripts文章给出了转录中共线性(co-linear)以及非共线性(non-co-linear,NCL)的定义。
所以,环状RNA属于非共线性RNA分子。
2、什么是ts-RNA
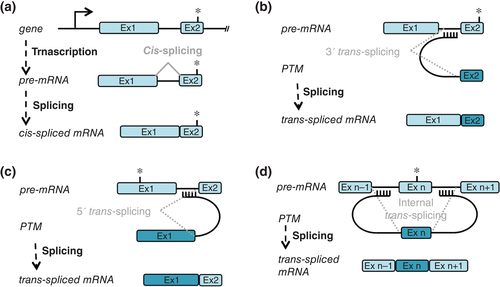 RNA剪接发生在一个前体mRNA(pre-mRNA)的分子内部称为cis-splicing;如果发生在两个前体 RNA分子之间,形成杂合的mRNA,那么这种剪接方式就称为trans-splicing。trans-splicing的两个前体RNA可以来自同一基因的同义链,也可以来自正反义链,甚至可以来自不同染色体的两个基因。
RNA剪接发生在一个前体mRNA(pre-mRNA)的分子内部称为cis-splicing;如果发生在两个前体 RNA分子之间,形成杂合的mRNA,那么这种剪接方式就称为trans-splicing。trans-splicing的两个前体RNA可以来自同一基因的同义链,也可以来自正反义链,甚至可以来自不同染色体的两个基因。
ts-RNA即trans-spliced RNA,本篇文章主要研究的是基因内ts-RNA(见下图)。
3、基因内ts-RNA与circRNA
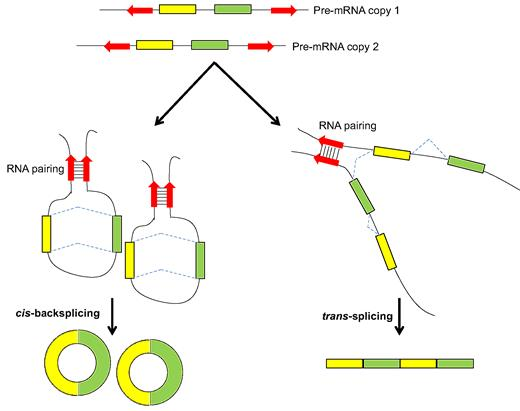 从图中我们可以看出,circRNA实际上是cis-spliced RNA,而基因内的ts-RNA是由同一基因的两条前体RNA拼接而成。
从图中我们可以看出,circRNA实际上是cis-spliced RNA,而基因内的ts-RNA是由同一基因的两条前体RNA拼接而成。
从示意图我们可以看出,基因内ts-RNA也包含 back-spliced junction,文章中称为NCL junction。
早在2018年circRNA公众号就解读过ts-RNA,大家有兴趣可以访问:
Nucleic Acids Research:反向拼接产物也可能是分子间拼接的产物!
检测ts-RNA面临的挑战
由于成熟的ts-RNA包含poly(A)而环状RNA不包含,因此,poly(A)+的二代测序比较适合检测ts-RNA。
然而,基因内ts-RNA的识别仍要面临诸多挑战:
• 目前的二代测序短读长仍存在错误基因组比对的问题;
• 通过二代测序技术难以区分ts-RNA与环状 RNA,特别是它俩可能具有相同的NCL junctions;
• 模板交换事件(template swithching events)等实验产物经常出现在cDNA产物中,可能会被误认为NCL事件;
• DNA水平的遗传重组所形成的NCL也是检测真实 ts-RNA的另一大挑战;
• 目前仍不清楚ts-RNA是否只是pre-mRNA剪切不完善的副产物,或者是否能够介导对应共线性宿主的表达;
• 基因内ts-RNA生物合成仍不清楚。
敢情这种分子比环状RNA检测还要难。
为了解决上面的问题,作者还采用了三代长度长测序。相比二代测序,三代测序在识别ts-RNA具有巨大的优势:
• 避免了短读长测序识别NCL junctions所存在的偏差;
• 能够区分ts-RNA与circRNA。
二代+三代测序检测ts-RNA
作者通过整合二代测序与三代测序数据,开发了 NCLscan-hybrid流程用于检测基因内ts-RNA(如下图)。
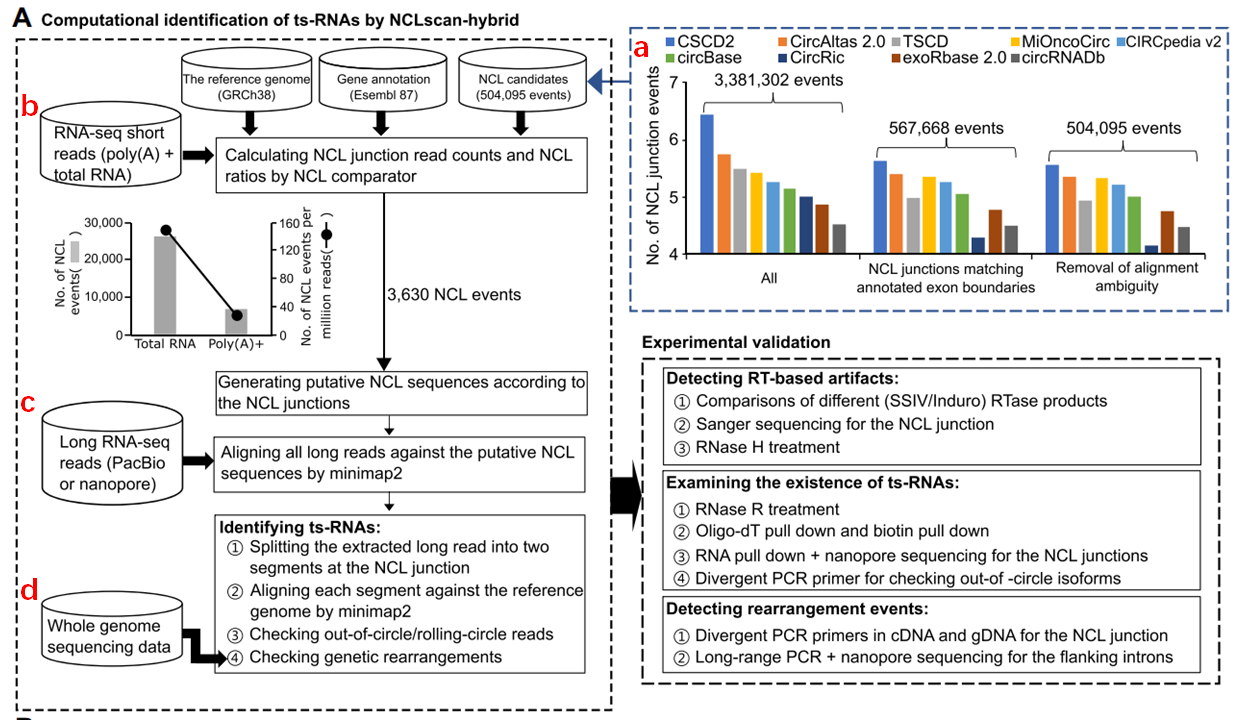
 NCLscan-hybrid检测ts-RNA的流程:
NCLscan-hybrid检测ts-RNA的流程:
作者收集了9个环状RNA数据库的基因内的NCL junctions(即之前被识别到的back-spliced junctions)作为候选NCL junction(图A-a),通过total RNA建库以及poly(A)+两种建库方式对 MCF-7细胞系进行测序,然后对候选NCL junction进行定量,只有被两种文库支持的NCL junctions才被保留(图A-b)。
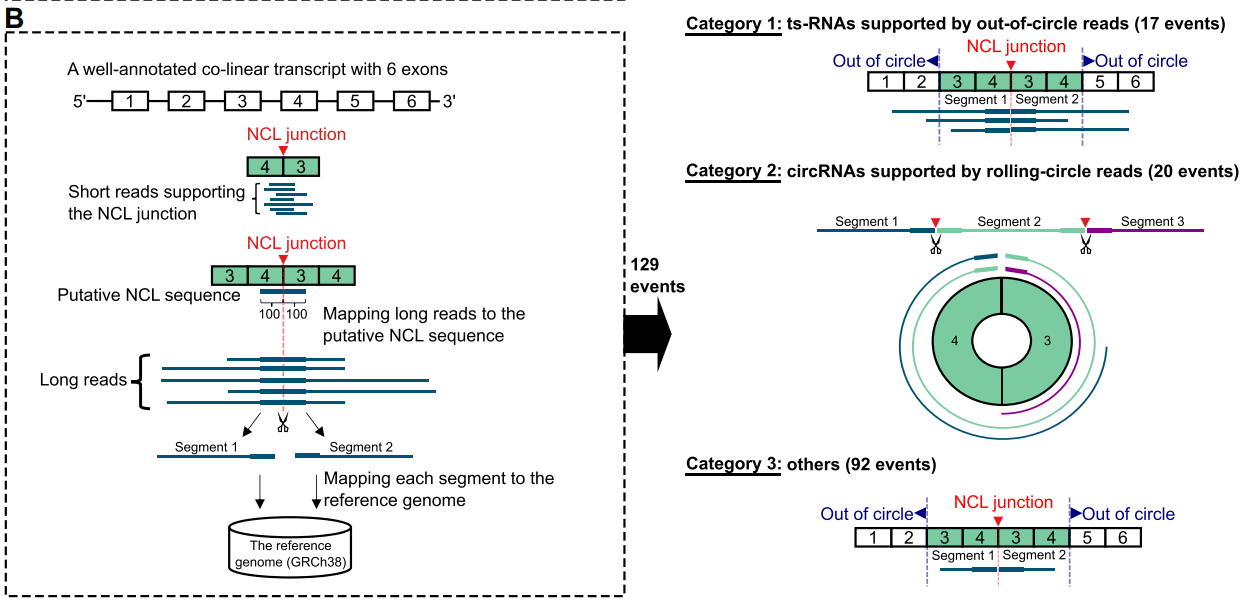
基于被保留的NCL junctions,NCLscan-hybrid 将NCL junction侧翼外显子拼接生成假参考序列,然后将三代测序的reads通过比对到假参考序列上(图A-c,图B)。只有包含三代测序reads的 NCL事件被保留。
随后,包含NCL junction的三代测序reads根据 NCL break points被分割成了两个部分,然后每一部分被比对到GRCh38参考基因组上(图B)。为了最大限度的减少被其他共线性或多重比对带来的假阳性,这里的NCL事件必须满足:被分割两部分的三代测序reads在比对时必须唯一匹配到相同的基因位点。
如果至少有一个分割部分超出了NCL供体或受体的剪接位点,那么这条三代测序的read被称为 “out-of-circle-read”(图B右侧),而这条read所对应的RNA被认定为ts-RNA。
如果一条read包含两个或多个NCL break points,并且不存在”out-of-circle-read”的序列,那么这条read被称为“rolling-circle-read”(图B右侧)。
在这项研究中,作者并没有发现同时包含”out-of-circle-read”以及“folling-circle-read”的事件。
最后,作者识别到了17个来自8个ts-RNA的事件,其中7个事件来自于ts-ARFGEF1。
实验验证ts-RNA
为了排除真实ts-RNA的干扰项,包括:
• 反转录产物,例如模板交换事件(template swithing events);
• 环状RNA;
• 遗传重组。
作者设计了一系列验证步骤对8个候选ts-RNA进行实验检验(下图是汇总):
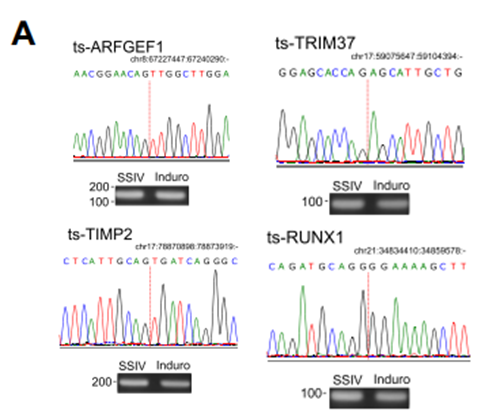
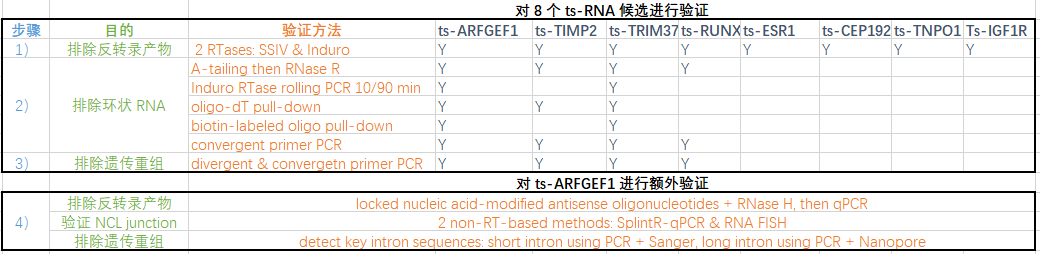 1) 由于反转录产物往往依赖于反转录酶,因此,通过两种不同的反转录酶(逆转录病毒的反转录酶 SSIV和细菌II型内含子反转录酶 Induro)进行并行实验,然后通过sanger测序检测NCL junction。作者发现8个候选ts-RNA都与反转录酶没有关系(A)。
1) 由于反转录产物往往依赖于反转录酶,因此,通过两种不同的反转录酶(逆转录病毒的反转录酶 SSIV和细菌II型内含子反转录酶 Induro)进行并行实验,然后通过sanger测序检测NCL junction。作者发现8个候选ts-RNA都与反转录酶没有关系(A)。
 2) 为了区分ts-RNA与circRNA,作者首先对total RNA进行加A尾(A-tailing),然后用RNase R消化(B)。
2) 为了区分ts-RNA与circRNA,作者首先对total RNA进行加A尾(A-tailing),然后用RNase R消化(B)。
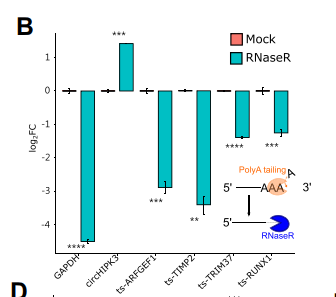
然而,有许多存在高级结构或G四联体的环状RNA对RNase R耐受。为了解决该问题,作者通过在第10分钟和第90分钟停止Induro反转录酶(具有强烈且稳定的滚换反转录活性)的反转录反应,然后用qPCR检测NCL junction。如果在90min的cDNA产物比10min时更高,那么该NCL junction更可能来自环状RNA(C)。
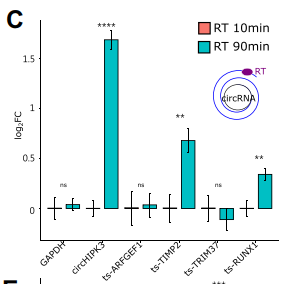
同时,作者用oligo-dT pull-down检测了这些候选分子是否包含poly(A)尾(D)。
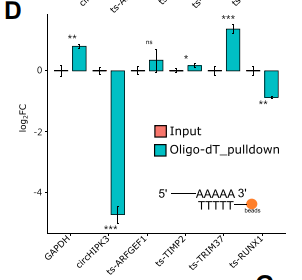 为了进一步检测候选分子是线性而非环状,作者通过生物素标记的寡核苷酸检测(biotin-labeled oligonucleotide assay)来捕获线性转录本,并观察与oligo-dT pull-down结果的相似性(E)。
为了进一步检测候选分子是线性而非环状,作者通过生物素标记的寡核苷酸检测(biotin-labeled oligonucleotide assay)来捕获线性转录本,并观察与oligo-dT pull-down结果的相似性(E)。
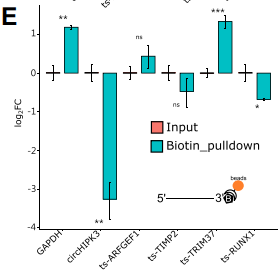
另外,为了证实ts-RNA存在,作者设计了 convergent primer pairs——其中一条引物跨越 NCL junction,另一条落于NCL供体位点外侧——并在多个乳腺癌细胞系对ts-RNA进行检测(F)。
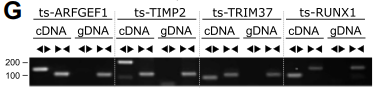
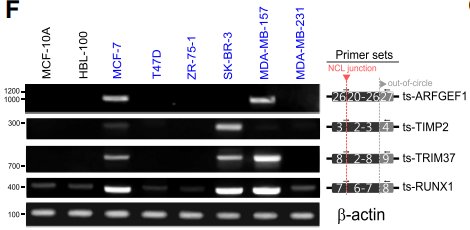 3) 为了验证ts-RNA主要来自转录后而不是遗传重组,作者设计了divergent以及convergent primers对ts-RNA转录本进行扩增。PCR结果显示,divergent primers扩增的产物来自cDNA而不是gDNA,而convergent primers对cDNA和gDNA 都有扩增(G)。
3) 为了验证ts-RNA主要来自转录后而不是遗传重组,作者设计了divergent以及convergent primers对ts-RNA转录本进行扩增。PCR结果显示,divergent primers扩增的产物来自cDNA而不是gDNA,而convergent primers对cDNA和gDNA 都有扩增(G)。
 4) ts-ARFGEF1额外验证:
4) ts-ARFGEF1额外验证:
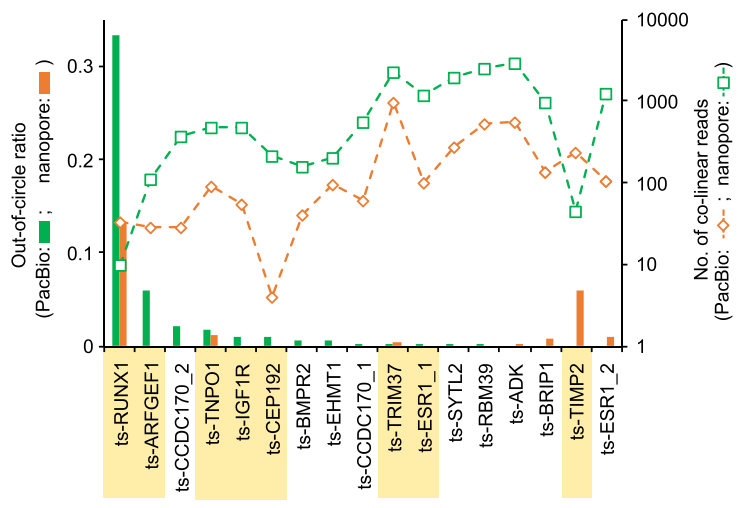
上面一系列验证步骤表明ts-ARFGEF1与ts-TRIM37主要来自trans-splicing,并且ts-ARFGEF1具有较高的out-of-circle比率(如下图)。
 因此,作者采用了更多的验证步骤来检验ts-ARFGEF1:
因此,作者采用了更多的验证步骤来检验ts-ARFGEF1:
1、反转录产物的排除
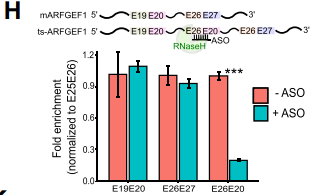
作者针对NCL junction设计了锁核酸的反义寡核苷酸(locked nucleic acid-modified antisense oligonucleotides,LNA-ASO),然后用RNase H处理来自MCF-7细胞系的total RNA(H, top),随后qRT-PCR分析对包含NCL junction的转录本进行了检测。RNase H会消化杂交到DNA 上的RNA,而来自反转录的产物表达将不会受严重影响(H,bottom)。
 随后,作者采用了两种非反转录的验证方式来检测 ts-ARFGEF1的NCL junction。
随后,作者采用了两种非反转录的验证方式来检测 ts-ARFGEF1的NCL junction。
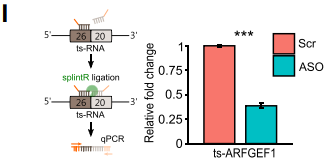
– SplintR-qPCR
通过两条探针与NCL junction末端杂交,然后用 SplintR连接酶连接两条探针(I,left)。随后用 ASO对NCL junction进行敲除,并用qPCR进行丰度检测(I,right)。
– RNAFISH
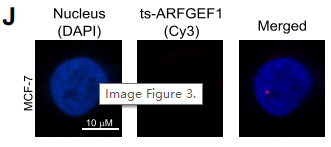
RNA荧光原味杂交显示ts-ARFGEF1主要定位与细胞核中(J),亚细胞组分分析也有同样的结果。
 2、排除circRNA
2、排除circRNA
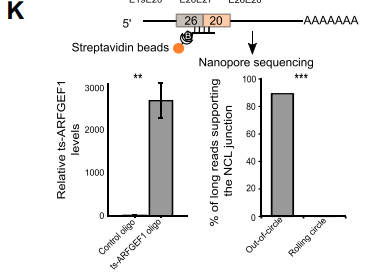
首先设计convergent primers来证实ts-RNA序列跨越了NCL供体和受体位点,然后靶向NCL junction的生物素标记的寡核苷酸来pull down对应的转录本,随后用nanopore对捕获到的分子进行测序(K,top)。
qRT-PCR表明pull down效率较高(K,buttom left),nanopore测序的reads表明89%的reads 属于out-of-circle(K,buttom)。
 3、排除遗传重组
3、排除遗传重组
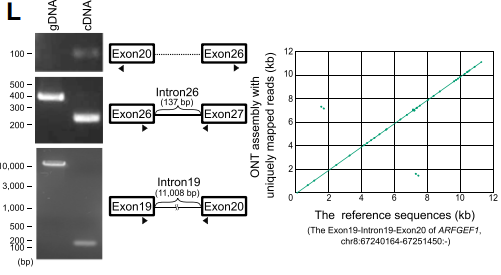
针对Intron26和Intron19(L,middle),作者通过 PCR+Sanger测序证实了短序列的Intron26,然后用长的PCR实验+Nanopore测序证实了长序列的 Intron19(L,left,right)。同时将Nanopore reads比对到Exon26,证实了Intron19确实不包含 Exon26序列。以上结果排除了ts-RNA来自遗传重组的可能。
 ts-RNA的起源
ts-RNA的起源
已有研究表明ts-RNA的形成与NCL junction侧翼内含子中的反向互补序列(RCS)相关,但目前仍缺少内源性的实验证据。
因此,作者首先检测了ts-ARFGEF1 NCL junction侧翼内含子Intron 19和Intron 26中是否包含RCS,结果发现了一对RCS(A,buttom)。随后,作者用异位表达系统(ectopic expression system)和内源基因组修饰实验(endogenous genome modification experiment)检测RCS是否能够触发ts-ARFGEF1的生物合成。
1、异位表达系统构建
作者将包含Exon26-Intron26-Exon27(WT)与 Exon26-Intron26mut-Exon27(Mut,mutant RCS in intron 26)的区域分别插入到pFLAG-CMV2载体(A,top)。
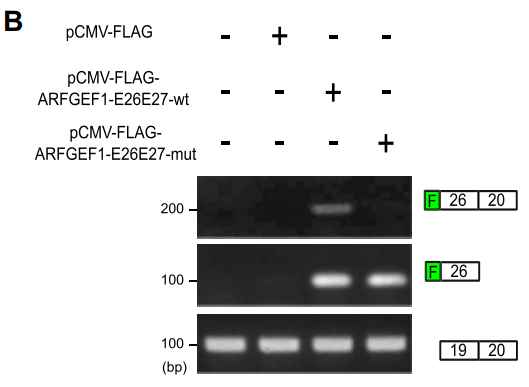
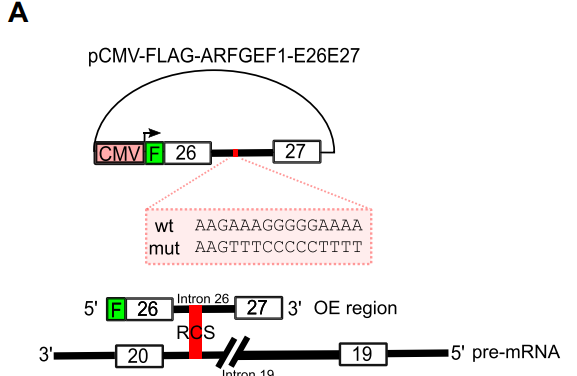 随后,这两种载体被分别转染到MCF-7细胞中,随后用带有FLAG跨junction的primer FLAG-Exon26-Exon20对ts-ARFGEF1的表达进行监控。结果显示,FLAG-Exon26-Exon20仅在WT 中能够被检测到,而Mut并没有(B)。
随后,这两种载体被分别转染到MCF-7细胞中,随后用带有FLAG跨junction的primer FLAG-Exon26-Exon20对ts-ARFGEF1的表达进行监控。结果显示,FLAG-Exon26-Exon20仅在WT 中能够被检测到,而Mut并没有(B)。
 用同样的方法,ts-TRIM37表达也被证实能够被 RCS诱导。
用同样的方法,ts-TRIM37表达也被证实能够被 RCS诱导。
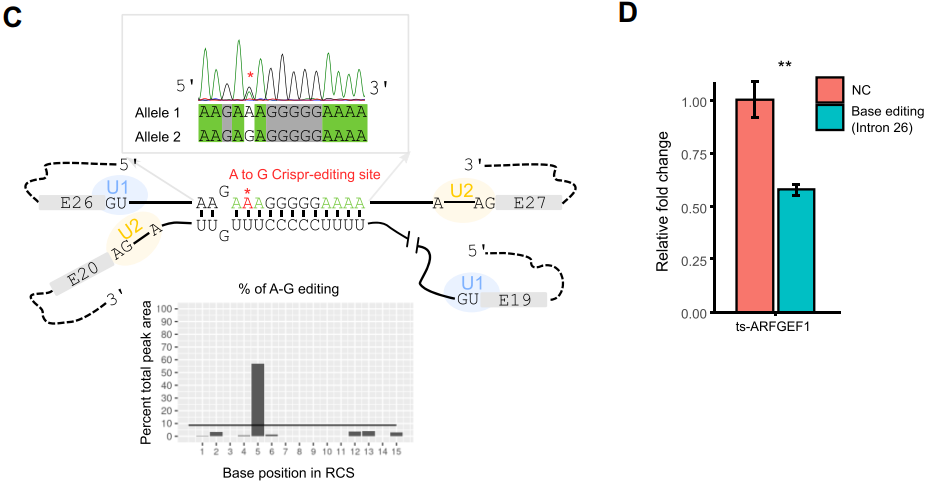
2、内源基因组修饰实验
作者用CRISPR/Cas9对Intron 26上的RCS位点进行了突变(C),ts-ARFGEF1的表达显著降低(D)。
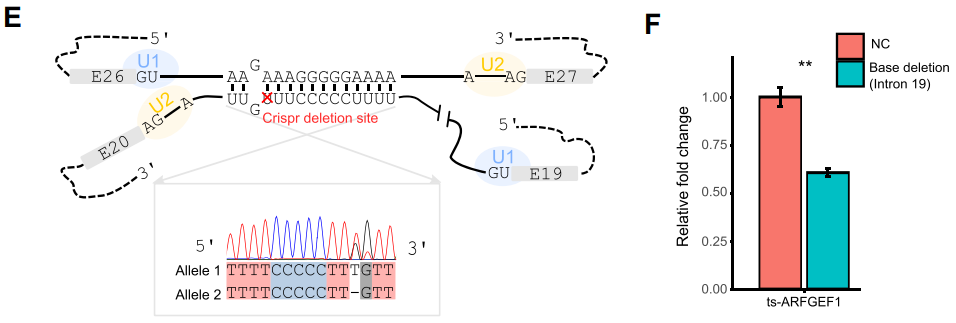
 随后,作者用高保真的CRISPR/Cas9系统将 Intron 19内的RCS删除了一个碱基(E),同样地,ts-ARFGEF1的表达显著降低(F)。
随后,作者用高保真的CRISPR/Cas9系统将 Intron 19内的RCS删除了一个碱基(E),同样地,ts-ARFGEF1的表达显著降低(F)。
 另外,值得注意的是,通过WB实验我们可以发现,上面的突变/删除实验并没有影响来源基因 ARFGEF1共线性转录本的蛋白表达水平(G)。
另外,值得注意的是,通过WB实验我们可以发现,上面的突变/删除实验并没有影响来源基因 ARFGEF1共线性转录本的蛋白表达水平(G)。
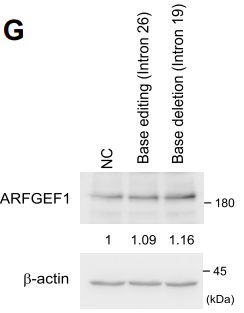
所有这些结果证明了侧翼内含子上的RCS对能够调控ts-ARFGEF1 NCL junction的形成。
ts-ARFGEF1的功能
作者发现ts-ARFGEF1在MCF-7以及MDA-MB-157两种乳腺癌细胞系中特异的表达,但在非肿瘤上皮乳腺细胞系MCF-10A和HBL-100中不表达(如下图)。
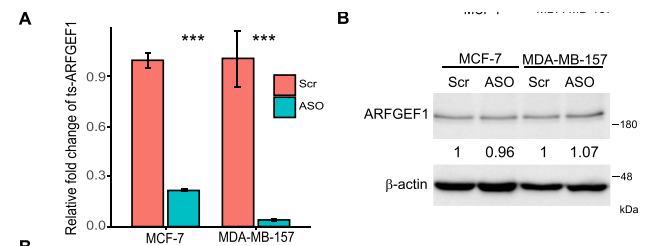
 因此,作者怀疑ts-ARFGEF1在乳腺癌细胞中具有功能。为此,作者设计了LNA-ASO来靶向它的 NCL junction,试图在两种乳腺癌细胞中沉默ts-RNA(A),并通过以下三种实验来验证ts-ARFGEF1的功能。同时,WB结果显示,这种敲低并不能影响来源基因ARFGEF1蛋白质的表达(B)。
因此,作者怀疑ts-ARFGEF1在乳腺癌细胞中具有功能。为此,作者设计了LNA-ASO来靶向它的 NCL junction,试图在两种乳腺癌细胞中沉默ts-RNA(A),并通过以下三种实验来验证ts-ARFGEF1的功能。同时,WB结果显示,这种敲低并不能影响来源基因ARFGEF1蛋白质的表达(B)。
 1、CCK-8 assay
1、CCK-8 assay
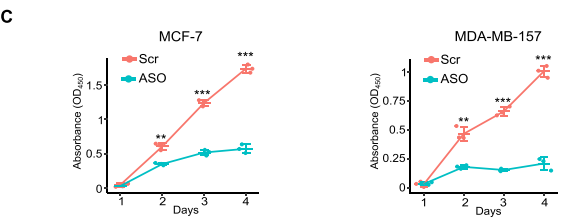
ts-ARFGEF1的敲低显著抑制了MCF-7和MDA-MB-157的增殖能力(C)
 2、colony formation assay
2、colony formation assay
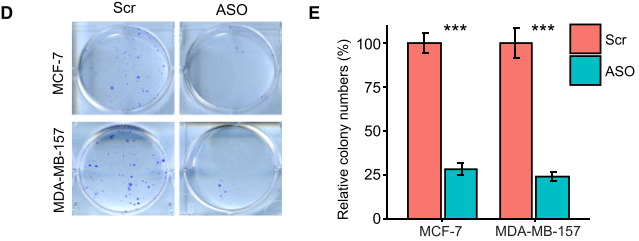
ts-ARFGEF1敲低后,细胞克隆形成的数据量和大小显著减少(D-E)
 3、flow cytometry
3、flow cytometry
ts-ARFGEF1的敲低能够显著增加两种乳腺癌细胞的凋亡(F-G)
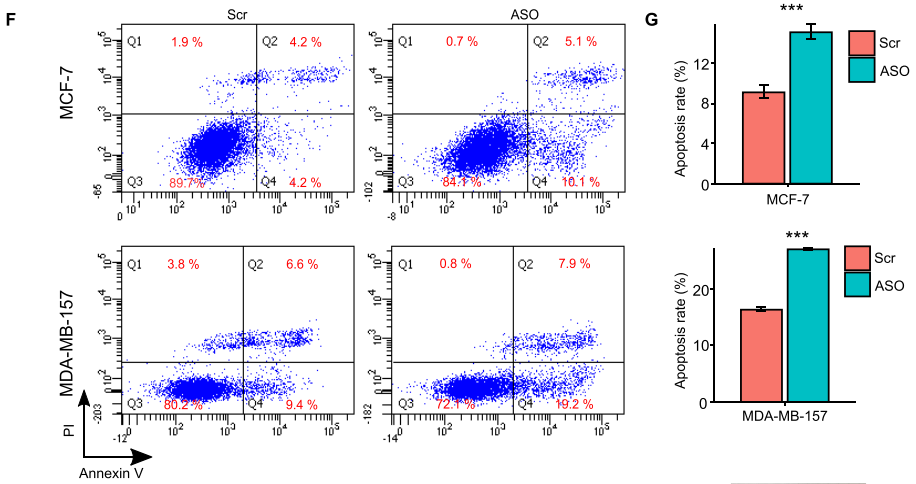 随后,作者想了解ts-ARFGEF1是否是通过编码蛋白发挥功能,因此预测了它的蛋白质编码序列 ——与ARFGEF1蛋白质序列部分重合。然而通过设计N端抗体对其蛋白质进行检测,并没有发现 ts-ARFGEF1具有蛋白质编码能力。
随后,作者想了解ts-ARFGEF1是否是通过编码蛋白发挥功能,因此预测了它的蛋白质编码序列 ——与ARFGEF1蛋白质序列部分重合。然而通过设计N端抗体对其蛋白质进行检测,并没有发现 ts-ARFGEF1具有蛋白质编码能力。
最后,作者试图检测ts-ARFGEF1在体内对肿瘤进展的影响,因此对裸鼠原位注射了control MCF-7 和ts-ARFGEF1-knockdown MCF-7,从而构建异位移植瘤模型。作者发现ts-ARFGEF1-knockdown小鼠中的肿瘤更小。
为了进一步确认ts-ARFGEF1敲低确实能够作为抑制肿瘤的治疗手段,作者先对裸鼠原位注射了 control MCF-7,当肿瘤生长到14天达到~25mm 时,对小鼠通过腹膜注射LNA-ASOs(H)。结果显示,LNA-ASOs注射的肿瘤生长率显著低于 control(I)。而到了接种后的第28天,LNA-ASOs肿瘤的容量以及重量被明显抑制(J-L)。
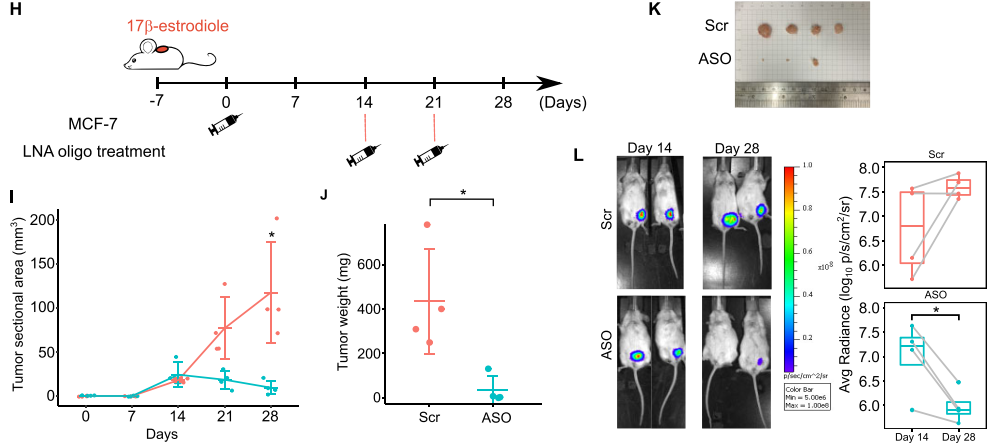 以上体外以及体内实验表明,ts-ARFGEF1表达被干扰能够显著抑制肿瘤细胞的生长。
以上体外以及体内实验表明,ts-ARFGEF1表达被干扰能够显著抑制肿瘤细胞的生长。
ts-ARFGEF1的潜在调控作用
由于ts-ARFGEF1主要定位在细胞核,因此作者怀疑它主要在转录或转录后调控基因的表达。为此,作者对ts-ARFGEF1敲除前后的MCF-7细胞进行了 microarray检测。
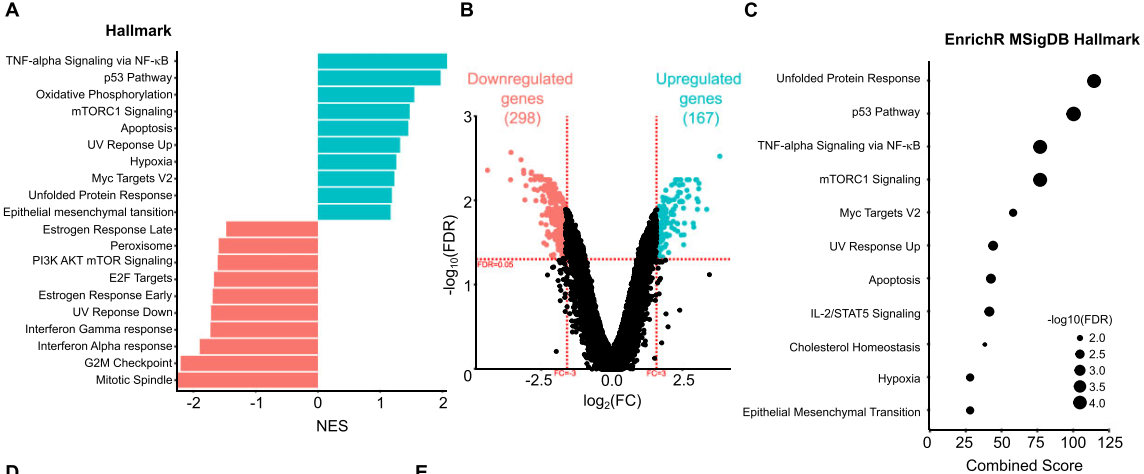
GSEA分析的上调和下调的前10个基因集合表明 ts-ARFGEF1敲低主要与细胞压力或细胞死亡信号通路相关,例如p53 signaling、apoptosis、unfolded protein responses(UPR)等(A)。差异分析获得了167个上调和298个下调的基因(B),且与GSEA结果一致,上调基因显著富集到了UPR和p53 signaling(C)。
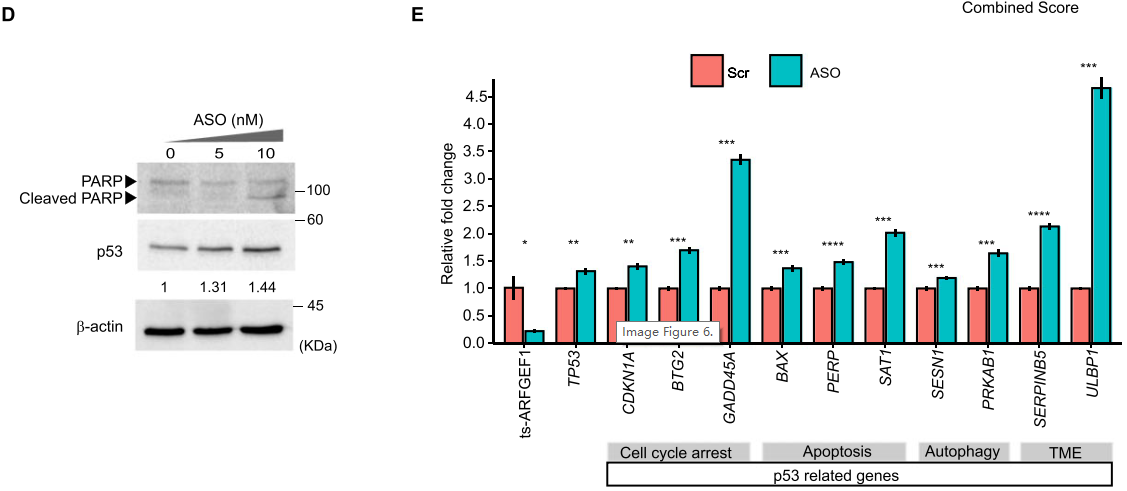
 由于p53是与凋亡相关的著名肿瘤抑制子,且p53 信号通路是凋亡信号通路中主要的通路之一。因此,作者对ts-ARFGEF1敲低进行表达检测,发现 p53蛋白质以及凋亡重要hallmark之一的PARP水解的表达水平都升高了(D),而TP53以及下游通路相关的基因表达都升高了(E)。
由于p53是与凋亡相关的著名肿瘤抑制子,且p53 信号通路是凋亡信号通路中主要的通路之一。因此,作者对ts-ARFGEF1敲低进行表达检测,发现 p53蛋白质以及凋亡重要hallmark之一的PARP水解的表达水平都升高了(D),而TP53以及下游通路相关的基因表达都升高了(E)。
 UPR是调控细胞内蛋白质稳态的关键通路,在长期或强烈的内质网(ER)应激下,强烈和持续的 UPR激活可引起癌症细胞凋亡。因此,作者试图检测ts-ARFGEF1是否能够调控ER内稳态并导致细胞凋亡。qRT-PCR显示UPR的两个关键基因 TSPYL2和ATF4以及下游效应因子ATF4、ASNS、CHAC1和SLC7A5的mRNA表达水平在 ts-ARFGEF1敲低后显著上调(F)。已知ER过载能够激活PERK/eIF2α/ATF4/CHOP通路从而诱导凋亡,作者通过WB检测到ts-ARFGEF1敲低后磷酸化后的eIF2α、ATF4以及CHOP蛋白质水平显著上升(G)。为了检测ts-ARFGEF1敲低是否会引起ER应激,作者检测了不正常堆叠蛋白质形成的聚集小体(aggresome),结果显示ts-ARFGEF1敲低的确会引起聚集小体的形成(H)。
UPR是调控细胞内蛋白质稳态的关键通路,在长期或强烈的内质网(ER)应激下,强烈和持续的 UPR激活可引起癌症细胞凋亡。因此,作者试图检测ts-ARFGEF1是否能够调控ER内稳态并导致细胞凋亡。qRT-PCR显示UPR的两个关键基因 TSPYL2和ATF4以及下游效应因子ATF4、ASNS、CHAC1和SLC7A5的mRNA表达水平在 ts-ARFGEF1敲低后显著上调(F)。已知ER过载能够激活PERK/eIF2α/ATF4/CHOP通路从而诱导凋亡,作者通过WB检测到ts-ARFGEF1敲低后磷酸化后的eIF2α、ATF4以及CHOP蛋白质水平显著上升(G)。为了检测ts-ARFGEF1敲低是否会引起ER应激,作者检测了不正常堆叠蛋白质形成的聚集小体(aggresome),结果显示ts-ARFGEF1敲低的确会引起聚集小体的形成(H)。
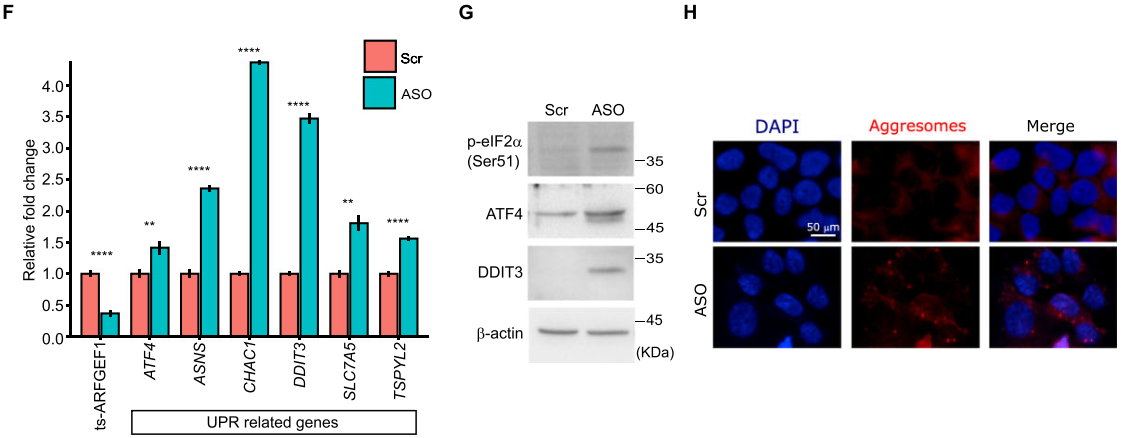 以上结果表明ts-ARFGEF1敲低会引起unfolded protein response进而通过激活 PERK/eIF2α/ATF4/CHOP信号通路诱导细胞凋亡,暗示了ts-ARFGEF1在MCF-7细胞的ER内稳态中具有重要作用。
以上结果表明ts-ARFGEF1敲低会引起unfolded protein response进而通过激活 PERK/eIF2α/ATF4/CHOP信号通路诱导细胞凋亡,暗示了ts-ARFGEF1在MCF-7细胞的ER内稳态中具有重要作用。
讨论
从本文我们可以看出,ts-RNA存在且具有重要的生物学功能。值得注意的是,ts-RNA与circRNA 都存在相同的分子特征——类BSJ——NCL junction。因此,在研究ts-RNA或circRNA时需要注意区分两者,特别是那些能够被RNase R消化的circRNA也许是ts-RNA。大家可以参考文章的实验步骤来区分ts-RNA以及circRNA。
文章中的软件NCLscan-hybrid可以访问 https://github.com/TreesLab/NCLscan-hybrid,但需要注意的是,该软件针对的是基因内的ts-RNA,且只能识别包含out-of-circle特征的ts-RNA。
原文链接:
https://doi.org/10.1093/nar/gkad623

.png)
