本期给大家介绍桑基图(Sankey Diagram),聊聊它在生物信息学领域的用途。
01简介
在基因表达调控的研究中,理解不同基因之间的相互作用和调控关系至关重要。然而,传统的研究方法往往只能处理单个基因或蛋白质的表达水平,而忽略了它们之间复杂的交互关系。为了解决这个问题,桑基图作为一种强大的可视化工具被引入到生物信息学领域,特别是在可视化ceRNA (competing endogenous RNA) 共表达网络中,它发挥着重要的作用。
众所周知,细胞内受miRNA调控的各种分子(包括mRNA、lncRNA和circRNA等),会以miRNA为中心相互影响,构成复杂的ceRNA网络。那么桑基图在其中有什么用武之地呢?我们先来看看下面这张图:
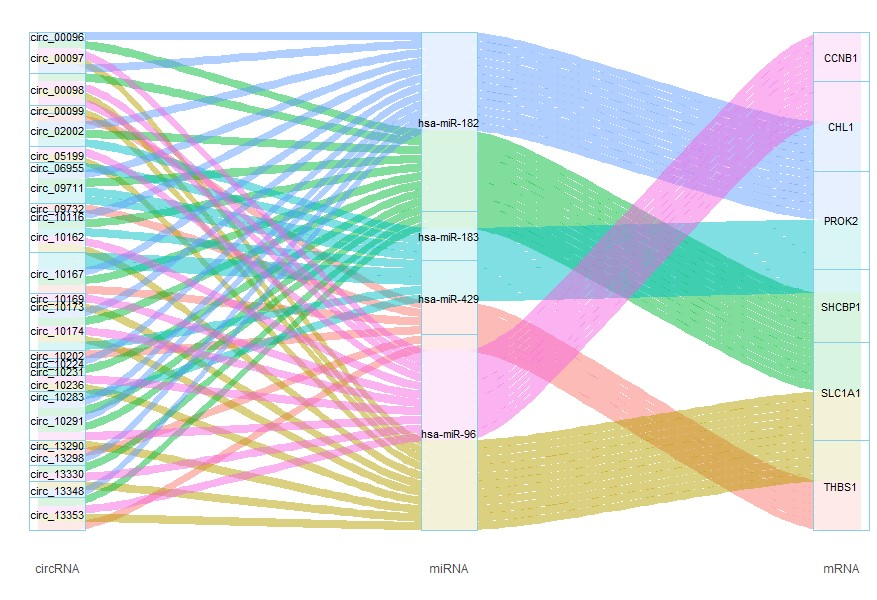
桑基图是一种流式图形表示方法,在上面这张示例图中,分子(circRNA、miRNA、mRNA)用节点来表示,而它们之间的共表达关系则用边来表示,这些边可以是箭头或线条,如果三个分子之间具有相互作用和调控关系,那么就会用一条具有特定颜色的线条来连接,多条这样的关系网就会形成能量分流一样的形状,极具美感。通过对这些节点和边进行布局与排列,整个网络的拓扑结构和核心分子也就一目了然地呈现在我们面前,我们可以很清晰地看到某个miRNA具体调控着几个mRNA和几个circRNA,以及哪些分子之间可以形成关系网络等要素。桑基图将复杂的关系网络简洁化,正好为我们解决了痛点。
那如何绘制桑基图呢?目前有不少工具可以实现,比如ggalluvial、sankeyD3和networkD3等,这些R包自带的函数均可达到自动统计和快速出图的效果,只需要简单地输入基因或蛋白质共表达关系的数据,即可轻松实现可视化,同时它们也支持自定义颜色和修改标签等功能。
02图解
节点:从左至右,第一列是circRNA,第二列是miRNA,第三列是mRNA。
边:连线表示circRNA、miRNA、mRNA三者之间的相互关系,每种关系都会标记相应的颜色。
03应用
在生物信息学领域,桑基图被广泛应用于基因表达谱、代谢通路和蛋白质互作网络的可视化。此外,在单细胞亚群分类分布以及物种门类分类等场景也都可以用桑基图来呈现。
好了,本期关于桑基图的介绍完毕,感兴趣的小伙伴们可参考一下软件的官方文档进行绘制:
ggalluvial (http://corybrunson.github.io)
networkD3(https://christophergandrud.github.io/networkD3/)
sankeyD3(https://github.com/fbreitwieser/sankeyD3)
.png)
