值此元旦佳节之际,山人首先预祝各位身体健康,工作顺利。刚刚过去的2019年,circRNA研究继续保持快速发展。中国科学院与科睿唯安联合发布的《2019研究前沿》中,“环状RNA作为癌症新的生物标志物”成为生物科学领域6个新兴前沿之一,这也是环状RNA作为关键词连续三年进入《研究前沿》报告的名单。2019年共发表circRNA相关SCI论文885篇,较2018年增长20%,其中大于10分的文章多达58篇,是2018年的3倍!呈现量质齐升的良好态势。国家自然科学基金项目总数维持稳定,说明国内circRNA研究团队数目增速有所放缓。下面让我们一起回顾一下2019年circRNA研究的总体情况和几个主要方向的详细情况:
1. 2019年circRNA研究总体情况概览:
1.1 circRNA相关发表文章情况汇总
2019年circRNA研究论文呈现量质齐升的良好态势,发表论文总数量较2018年有较大的增长,尤其是影响因子大于10 的文章数目更是有了巨大的增长。发表文章的通讯作者单位情况来看,我国是circRNA研究的绝对主力阵地,但2019年美国,德国,加拿大等传统生物医学研究强国也陆续发表了一些有重要影响的研究论文,说明circRNA的研究已经慢慢被国际学术界接受和认可,也预示着将来circRNA会有更多重要的研究成果。
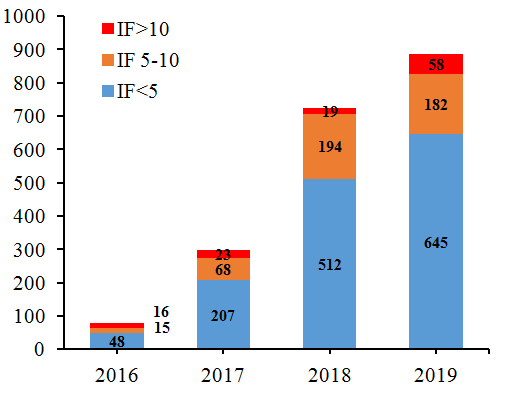
图1 2016 ~ 2019年circRNA相关发表文章信息汇总
1.2 circRNA相关国家自然科学基金情况汇总
2019年circRNA研究领域共有257个项目获批,其中包括杰出青年基金项目1项,重点项目2项,创新研究群体项目1项,面上项目113项,地区基金项目24项,青年科学基金项目116项。
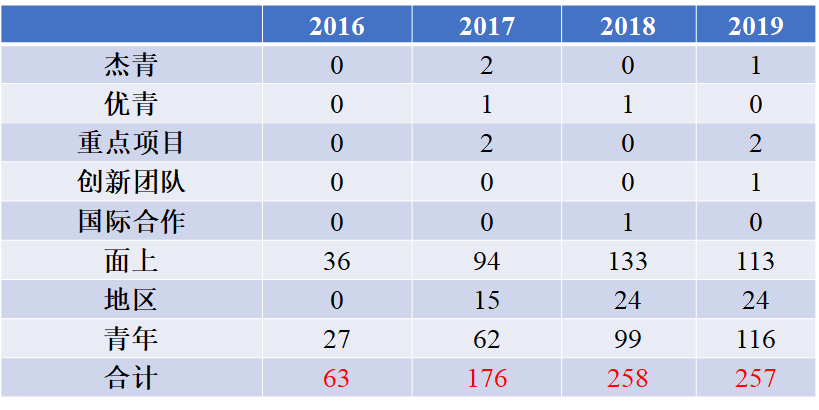
表1 2016 ~ 2019年国家自然科学基金circRNA相关项目信息汇总
2019年获批项目绝大部分的内容是针对特定circRNA的功能研究,大部分都给出了明确的机制和通路,表明circRNA的功能研究已进入全新的阶段。总体呈现的一些特征如下:
(1) 学科方向方面,医学领域依然具有压倒性优势,动物学、植物学以及生物信息学方向也有获批的项目。但生物化学和分子生物学,细胞生物学,遗传学,干细胞与再生医学等学科方向的项目偏少。可能与circRNA基础生命科学问题的研究难度有一定关系。
(2)研究内容更加明确和具体。2019年获批的项目从题目中不难看出,大部分都是基于某个特定circRNA分子和特定的通路和机制来写的,说明申请者已对相关分子的功能和作用机制有了较详细的认知。这也说明circRNA的功能和机制的研究已经越来越细致和深入。
(3)circRNA的功能机制模型和通路呈现多样化趋势。2019年获批的项目中除了传统的竞争性结合miRNA功能模式,circRNA直接翻译多肽,与蛋白的相互作用以及circRNA的m6A修饰等功能模型相关的项目越来越多。此外,近几年热度较高的分子细胞生物学机制和通路也在今年获批的cirRNA项目中多次出现,包括m6A修饰,外泌体,细胞焦亡,相分离,铁死亡等等。说明circRNA的研究正越来越融入分子细胞生物学研究中。
2. circRNA基本生命科学问题研究进展
circRNA早在1976年就发现了,但真正的研究热潮是从2012年开始的,主要是伴随着高通量测序技术的广泛应用而逐渐兴起的。目前人类组织来源的circRNA已经发现了几十万种,但关于它们的表达机制,功能作用方式,修饰及其调控方式,二级/高级结构,降解机制等基本生命科学问题的研究和认识还在持续进展中。2019年关于circRNA的生成机制,m6A修饰,翻译产物,互作分子,降解机制方面有一些进展,有关circRNA的特殊生物学功能机制研究也有一些报道,现分别汇总如下:
2.1 circRNA生成机制研究进展
2019年关于circRNA生成和表达机制有一些进展,包括通过解析酵母剪切体E复合体的结构,揭示了反向剪切的结构生物学机制,发现了几种能调控circRNA生成的特殊RNA结合蛋白,首次报道人类线粒体来源circRNA等等。
酵母剪切体E复合体结构揭示circRNA形成机制
2019年9月6日,Nature杂志发表了一项酵母剪切体E复合体的结构生物学研究成果,揭示了E复合体的组装机制,首次从结构生物学角度证明了“Intro-definition”和“Exon- definition”两种E复合体组装的机制均可存在。基于这一结构作者分析了长外显子circRNA的形成机制,外显子足够长的情况下,EDC复合体可以介导pre-mRNA内部反向剪切,最终形成circRNA。论文的通讯作者是美国科罗拉多大学安舒茨医学校区的Rui Zhao和加州大学洛杉矶分校的Z. Hong Zhou([1])。
推荐阅读:Nature:酵母剪切体E复合体结构揭示circRNA形成机制
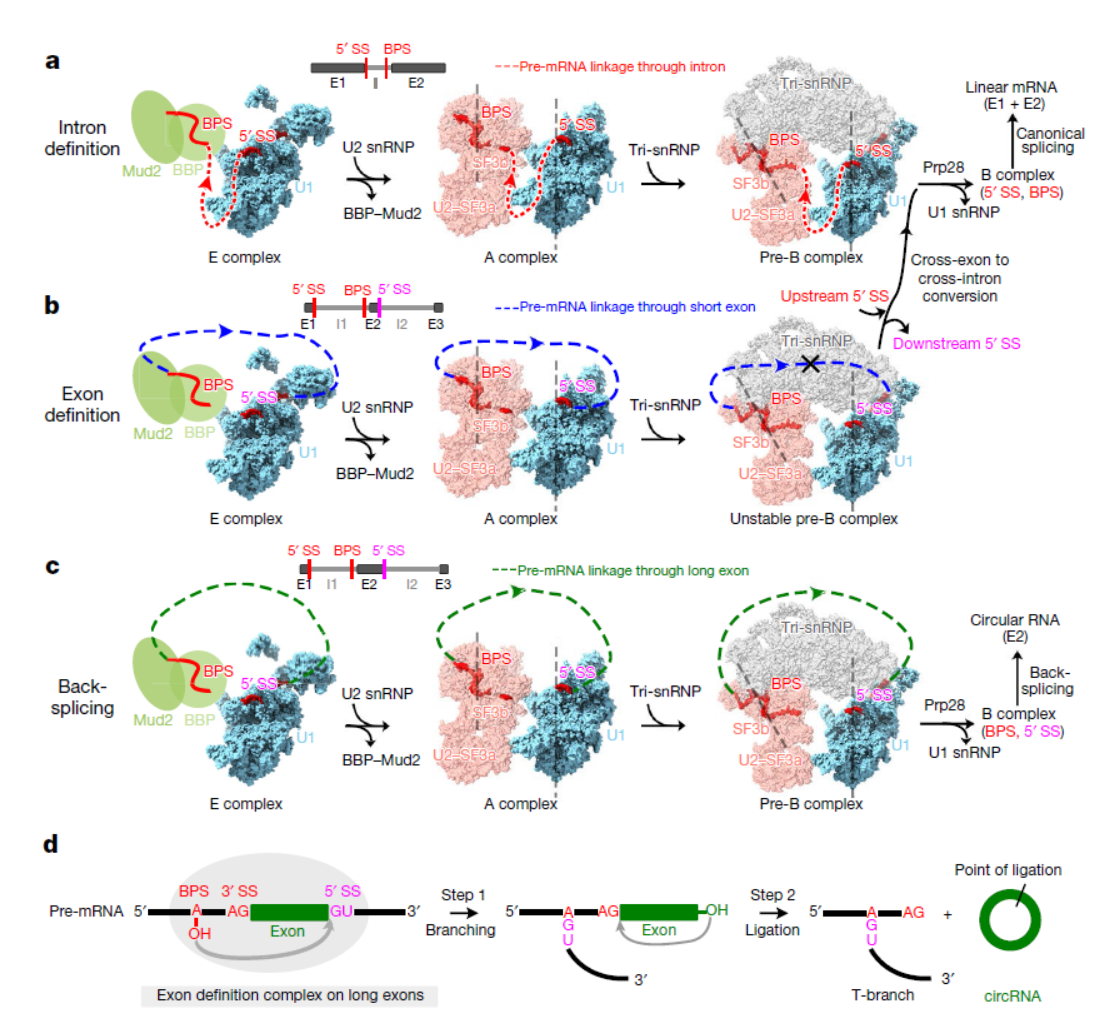
图2 剪切体E复合体介导pre-mRNA和circRNA剪切的机制 ([1])
调控circRNA生成的RNA结合蛋白
RNA结合蛋白(RBP)是调控circRNA生成的重要因素,很可能是circRNA组织/疾病特异性表达的主要机制。人们一直在不断探索和发现可调控circRNA生成的RBP蛋白, 2019年共有5篇文章涉及到RBP调控circRNA生成的研究,具体信息汇总如下:
| RBP | 所调控基因/circRNA | 论文题目 | 杂志 |
| Sam68 | SMN基因circRNA | Sam68 binds Alu-rich introns in SMN and promotes pre-mRNA circularization | Nucleic Acids Res |
| ESRP1 | circUHRF1 | Splicing factor derived circular RNA circUHRF1 accelerates oral squamous cell carcinoma tumorigenesis via feedback loop | Cell Death & Differentiation |
| RBM3 | SCD-circRNA 2 | The RNA-binding protein RBM3 promotes cell proliferation in hepatocellular carcinoma by regulating circular RNA SCD-circRNA 2 production | Cell Death & Disease |
| PTBP1 | circRNA_001160(hsa_circ_0001417, circ-ANKRD17) | Biosynthetic CircRNA_001160 induced by PTBP1 regulates the permeability of BTB via the CircRNA_001160/miR-195-5p/ETV1 axis | EBioMedicine |
| SRSF3 | 多种基因的circRNA | The role of SRSF3 splicing factor in generating circular RNAs | BioRxiv |
表5 2019年报道调控circRNA生成的RBP
哺乳动物的散在重复元件(MIR)调控 circRNA的生成
2019年6月4日,知名预印本杂志BioRxiv发表一项circRNA生成机制有关的研究成果,报道发现CDR1as也可以由哺乳动物的散在重复元件(Mammalian-Wide Interspersed Repeats,MIR)介导生成([2])。文章的通讯作者是日本藤田健康大学的Akila Mayeda。
推荐阅读:BioRxiv:MIR元件介导circRNA生成
图3 MIR调控CDR1as 的生成 ([2])
2.2 特殊类型circRNA的发现
常见的circRNA主要由mRNA的外显子通过反向剪切形成,但还有一些circRNA来源比较特殊,比如融合基因形成的circRNA等等。2019年陆续报道了一些新的circRNA形式,包括融合基因来源的circRNA,转录通读型circRNA,线粒体DNA来源circRNA。
融合基因来源circRNA研究
融合基因是很多肿瘤的驱动因素,如白血病,肺癌等等。早在2016年就曾报道融合基因来源的circRNA具有致癌作用(推荐阅读:cell杂志:融合环状RNA文献解读)。四川大学彭勇教授2018年也曾报道肺癌中EML4-ALK融合基因来源的circRNA(F-circrEA)可以作为肺癌诊断标志物(推荐阅读:新型融合基因来源环形RNA可能作为肺癌诊断标志物)。2019年,彭勇教授再次发表文章报道融合基因来源circRNA,报道SLC34A2-ROS1来源的circRNA(F-circrSR1和F-circrSR2)可促进肺癌侵袭([3]),相关工作发表于5月24日的Molecular Cancer杂志。
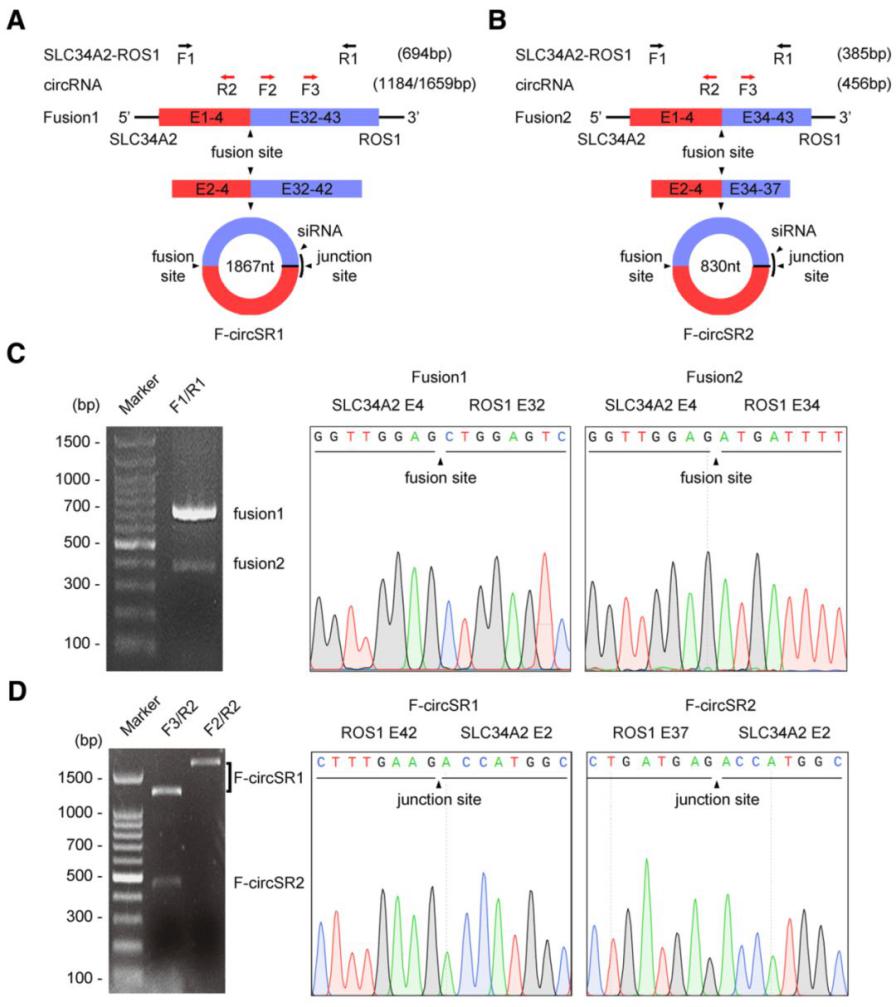
图4 SLC34A2-ROS1融合基因来源circRNA鉴定 ([3])
转录通读型circRNA
常见的基因转录活动在基因全序列被完全转录完成后终止,但在一些特殊情况下,转录过程还会继续往下进行,如果下游刚好在DNA的同一链上也有个基因,就会产生同一个转录本携带两个基因的形式,这就是转录通读(Read Through)。2017年就曾有文章报道转录通读过程与circRNA的形成有关(推荐阅读:重磅!Molecular Cell杂志发表circRNA形成机制的重要文章),当时发现干扰调控转录终止的基因可有助于转录通读产物的生成,并且可促进下游基因来源circRNA的生成([4])。
2019年2月7日的一篇Cell文章在全外显子组芯片数据中挖掘得到了大量的circRNA,其中也有转录通读型的circRNA产物(rt-circRNA)。文章分析了超过2000例不同组织来源的人类肿瘤标本,二十多种肿瘤中circRNA的总体表达情况,并汇总形成了MiOncoCirc数据库([5])。文章通讯作者为密歇根大学Alexey I. Nesvizhskii和Arul M. Chinnaiyan。
推荐阅读:重磅! Cell杂志同期发表两篇circRNA研究文章
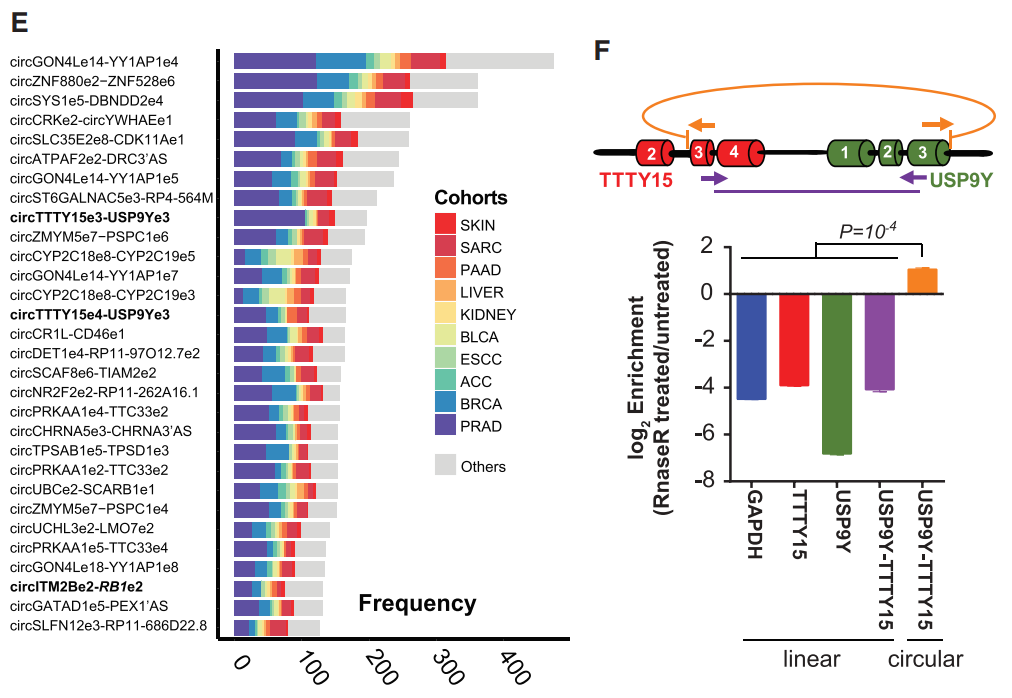
图5 人类肿瘤中存在转录通读型circRNA([5])
首次报道线粒体编码基因来源circRNA
2019年6月12日,预印本杂志BioRxiv发表了中国科学技术大学单革教授的一项研究成果,报道发现人与小鼠线粒体来源的circRNA(mecciRNAs)。这是首次报道发现哺乳动物线粒体来源的circRNA分子,丰富了circRNA的来源和认识([6])。
推荐阅读:BioRxiv | 线粒体来源circRNA及其功能
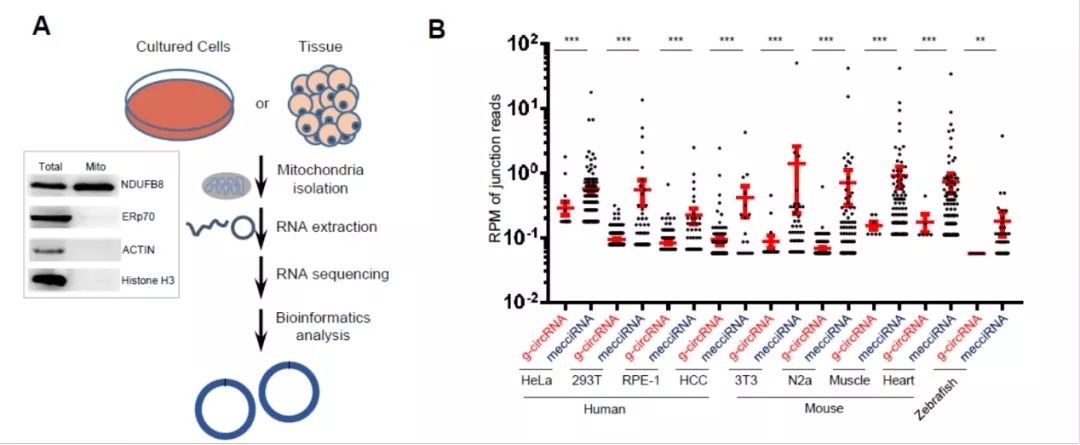
图6 mecciRNAs鉴定 ([6])
2.3 circRNA的m6A修饰研究进展
转录后修饰是RNA功能调控的一个维度,以m6A为代表的RNA修饰方式和调控机制的研究是近期RNA研究领域的重要热点方向之一。早在2016年就曾报道发现了circRNA存在特异性的m6A修饰(推荐阅读:重大发现:circRNA存在细胞特异性的m6A修饰!),并且介导了circRNA的翻译(推荐阅读:重磅:m6A修饰促进环状RNA的翻译)。
2019年circRNA的m6A修饰取得了不少进展,包括m6A修饰相关的circRNA降解机制,m6A修饰介导的circRNA出核现象,m6A修饰在细胞中区分内源和外源circRNA的机制等等。circRNA降解在下面有专门的内容介绍,细胞区分内源和外源circRNA的机制也在后面有专门的探讨,因此本部分主要介绍其他的内容:
m6A修饰介导circRNA出核
2019年10月16日,Nature Communications杂志在线发表了发表了一项circRNA m6A修饰的文章,报道发现m6A修饰的circNSUN2可结合YTHDC1并促进出核,进一步结合IGF2BP2促进HMGA2 mRNA的稳定,最终促进大肠癌的肝转移增强。文章的通讯作者是中山大学附属肿瘤医院的谢丹教授,徐瑞华教授和Wang Fengwei。本文从小样本临床标本的分析入手,找到CRC肝转移相关的circRNA分子,然后基于RNA Pull-down分析,发现了circNSUN2的相互作用蛋白YTHDC1和IGF2BP2,进而从干扰circNSUN2前后转录组差异分析结合作者所发现的现象找到HMGA2 mRNA是circNSUN2促进CRC肝转移的介导分子([7])。
推荐阅读:circRNA的m6A修饰再发一篇Nature Communications文章
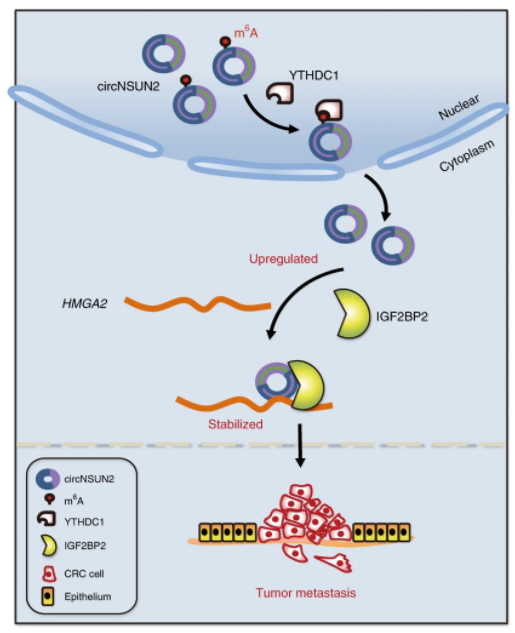
图7 m6A修饰促进circNSUN2出核介导CRC肝转移 ([7])
大鼠缺氧性肺动脉高压模型中m6A修饰研究
2019年5月1日,预印本杂志BioRxiv发表了浙江大学医学院应可净和章锐锋为通讯作者的文章,报道大鼠缺氧性肺动脉高压(HPH)模型中。circRNA m6A修饰状态的研究结果([8])。文章的结果表明一些circRNA的m6A修饰会伴随缺氧条件而发生修饰或去修饰的改变,说明m6A修饰与缺氧条件有密切的关系。
推荐阅读:BioRxiv | HPH模型中circRNA的m6A修饰
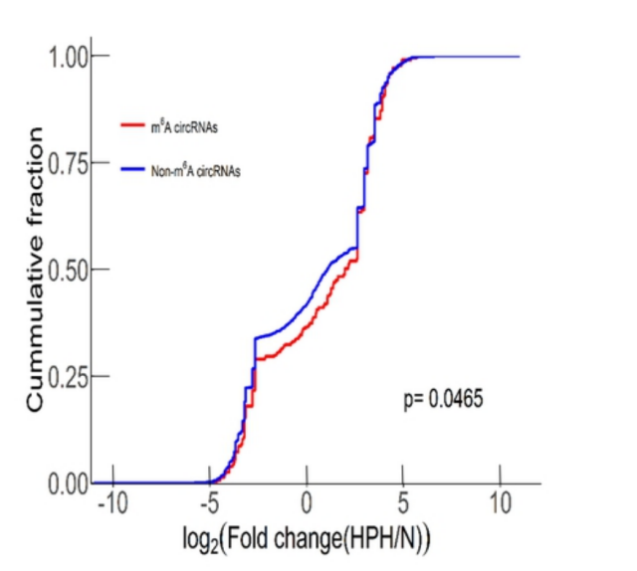
图8 大鼠HPH 模型中circRNA m6A修饰分析([8])
2.4 circRNA翻译研究进展
自2017年首次报道哺乳动物内源circRNA可以翻译多肽/蛋白以来,有关circRNA翻译的研究就一直备受关注。2019年circRNA的翻译研究也取得了一些进展,包括心脏翻译组学的研究中发现了可翻译的circRNA分子,Akt3,β-catenin等基因来源circRNA的翻译产物鉴定和研究。
心脏翻译组学研究发现可翻译circRNA
2019年5月30日,Cell杂志以Resource形式发表了一项心脏翻译组学的研究工作,系统分析了心脏组织中能被翻译的RNA分子,心脏中RNA翻译的规律和机制,研究中发现了一些非编码RNA来源的翻译产物,其中包括circRNA。文章的通讯作者是德国柏林Max Delbruck分子医学中心的Sebastiaan van Heesch和Norbert Hubner。本文作者分析了80例人心脏组织的翻译组学(其中65例扩张型心肌病,15例健康对照),发现了心脏组织特异的蛋白翻译机制。在捕获的可翻译的RNA分子中发现了169种lncRNA,40种circRNA,进一步佐证了非编码RNA,包括circRNA能被翻译的事实([9])。
推荐阅读:Cell | 心脏翻译组学文章,证明circRNA可翻译
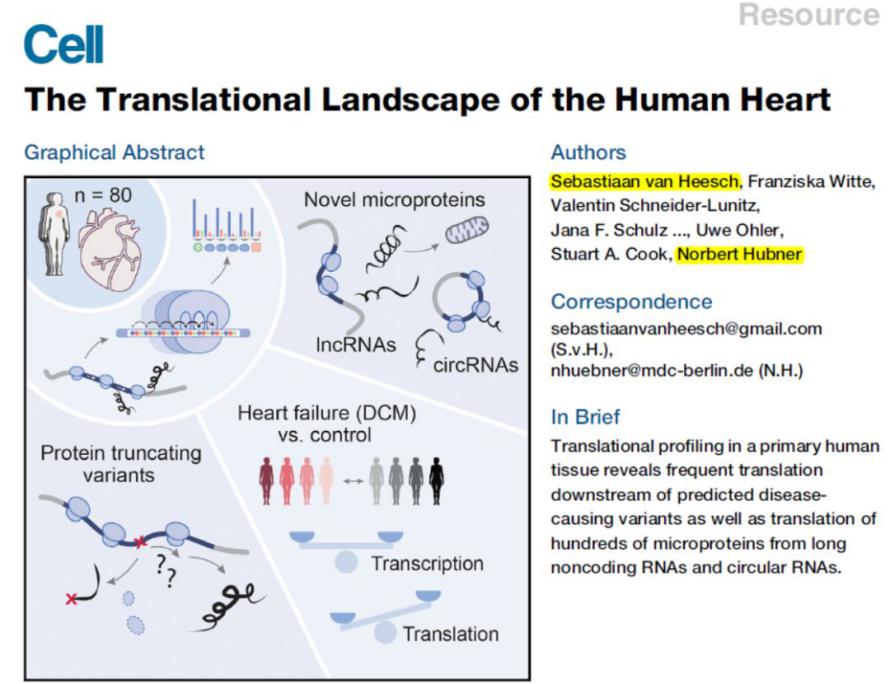
图9 心脏翻译组学研究 ([9])
2019年还有另外一篇翻译组学的研究工作发现了一些可能翻译的circRNA分子。12月20日,cells杂志发表了一项在分化型神经母细胞瘤中进行的circRNA表达谱分析,作者也进行了核糖体测序分析,在Ribo-seq的结果中共发现了173种可能被翻译的circRNA分子([10])。
Circ-AKT3编码174aa的多肽
2019年8月30日,Molecular Cancer杂志在线发表了中山大学附属第一医院张弩副教授的最新研究成果,报道发现AKT3来源的一个circRNA(Circ-AKT3)可编码一种174aa的多肽(AKT3-174aa)。在GBM细胞中过表达AKT3-174aa可抑制细胞增殖,抗辐射和体内致瘤能力,而敲低circ-AKT3后则增强了星形细胞瘤细胞的恶性表型。机制研究表明,AKT3-174aa与磷酸化PDK1竞争性相互作用,减少AKT-thr308磷酸化,并在调节PI3K/AKT信号强度中起负调节作用([11])。
推荐阅读:Molecular Cancer: 环状RNA AKT3翻译新功能蛋白负调控PI3K/AKT信号通路抑制脑胶质瘤
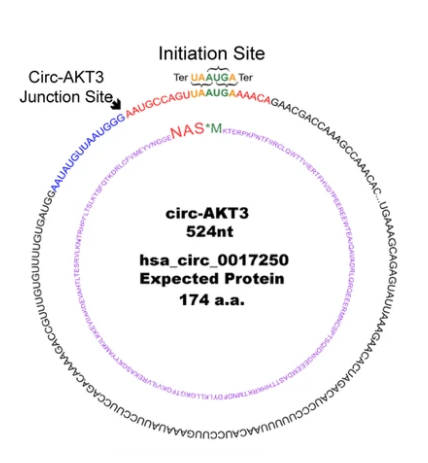
图10 Circ-AKT3编码一个174aa的蛋白([11])
circPPP1R12A翻译多肽通过Hippo-YAP 通路调控结肠癌侵袭
2019年3月29日,Molecular Cancer杂志在线发表了苏州大学附属第三医院蒋敬庭为通讯作者的文章,报道发现circPPP1R12A在结肠癌中显著高表达,进一步的研究发现circPPP1R12A可以编码一种73aa的小肽(circPPP1R12A-73aa),circPPP1R12A-73aa可以通过Hippo-YAP 通路参与结肠癌的转移侵袭([12])。
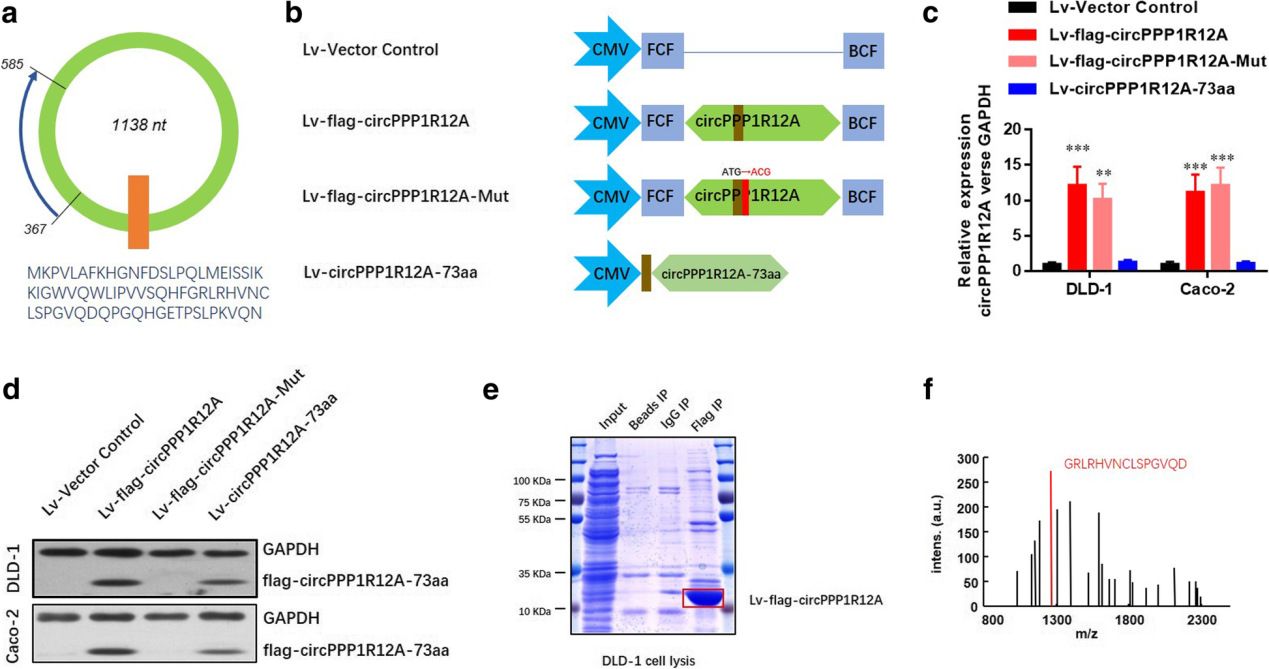
图11 circPPP1R12A编码一种73aa的小肽 ([12])
β-catenin来源circRNA翻译产物通过激活Wnt途径促进肝癌细胞生长
2019年4月26日,Genome Biology杂志发表了一项circRNA翻译产物的研究工作,报道发现circβ-catenin可翻译一种370aa的蛋白,可通过竞争性抑制GSK3β,阻断GSK3β对全长β-catenin的磷酸化和随后的泛素化降解([13])。文章的通讯作者为广州中医药大学第一附属医院的Jin-Fang Zhang、南方医科大学药学院的Wei-Ming Fu和中山大学附属第三医院的张琪。
推荐阅读:circβ-catenin翻译蛋白产生的“正反馈”效应
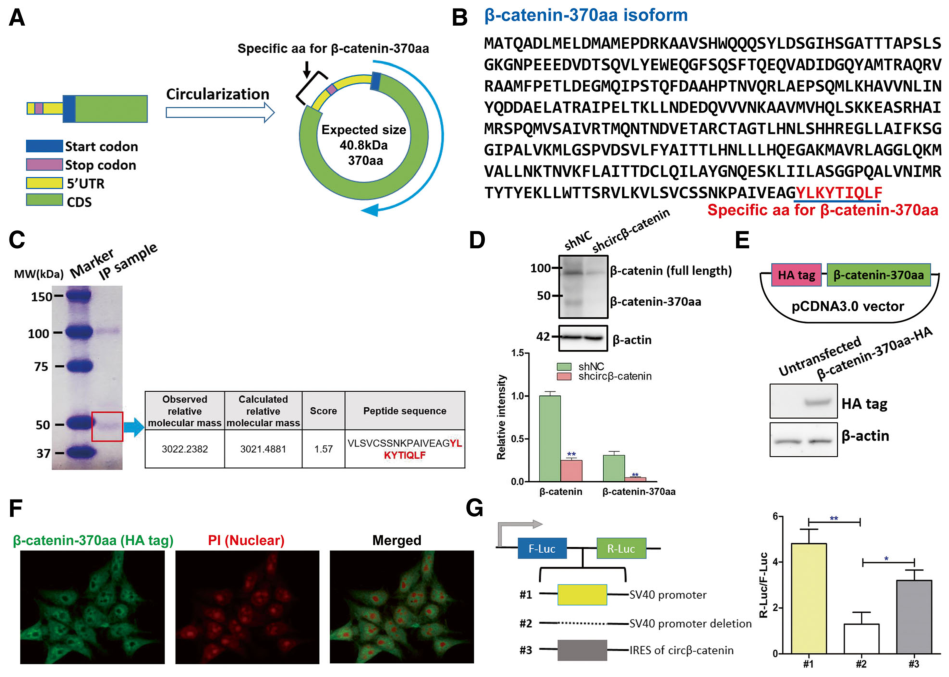
图12 circ β-catenin翻译新蛋白鉴定 ([13])
HPV编码circRNA翻译产物助纣为虐
2019年5月24日,Nature communications在线发表了一项HPV来源circRNA翻译产物致病性作用机制的研究。文章发现HPV16来源的circE7携带了完整的E7基因ORF序列,并证明circE7可翻译出对应的蛋白,可促进子宫颈癌细胞发生转化([14])。文章的通讯作者是来自美国德克萨斯大学西南医学中心皮肤科的Richard C. Wang和芝加哥西北大学Feinberg医学院微生物免疫学系的Laimonis Laimins。
推荐阅读:Nature communications | HPV circRNA编码蛋白“助纣为虐”?
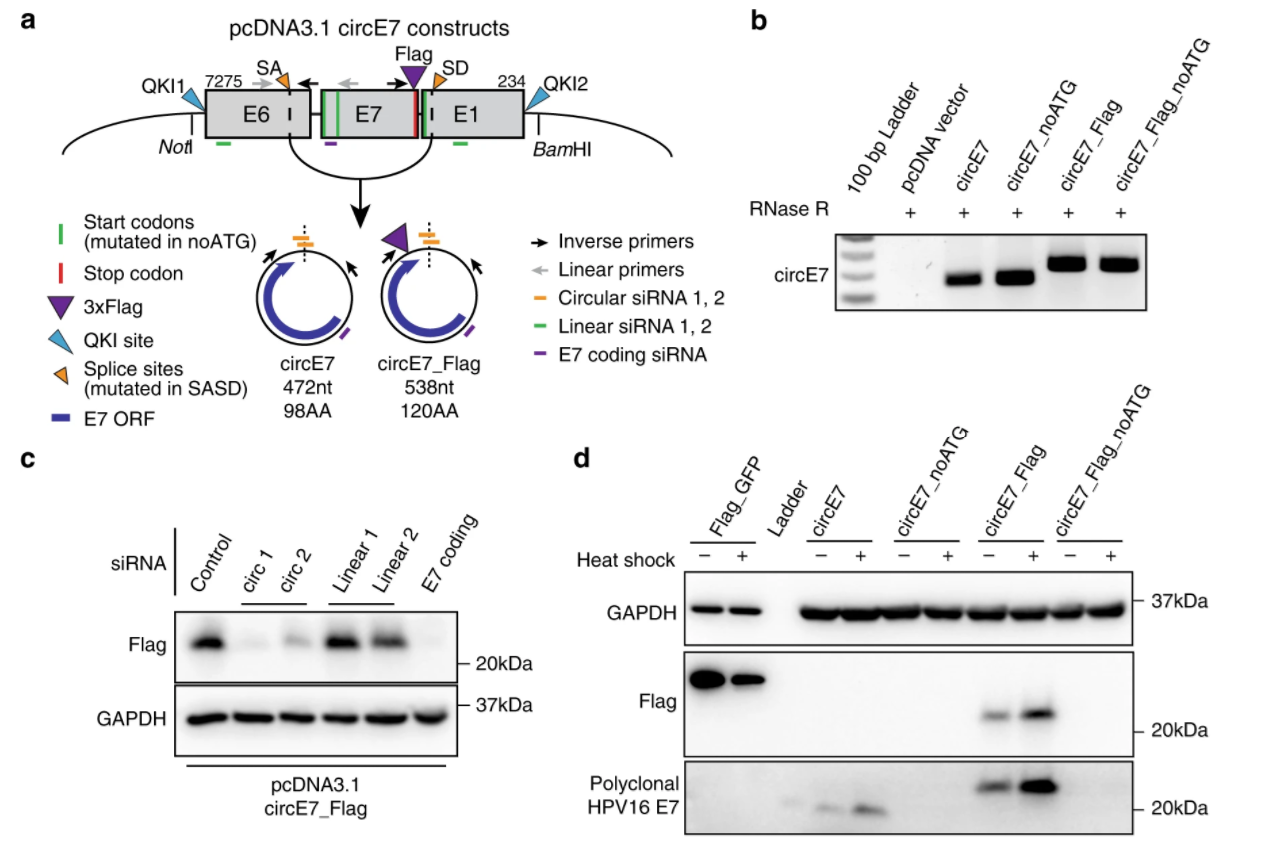
图13 HPV16来源circE7编码产物鉴定 ([14])
circRNA翻译起始机制研究
2019年9月25日,预印本杂志BioRxiv发表了中科院计算生物学研究所王泽峰教授为通讯作者的一项研究工作,通过设计随机序列后筛选的技术,分析了circRNA中IRES-like元件。共筛选到97种可以在circRNA中驱动翻译的富含AU的IRES-like元件([15])。
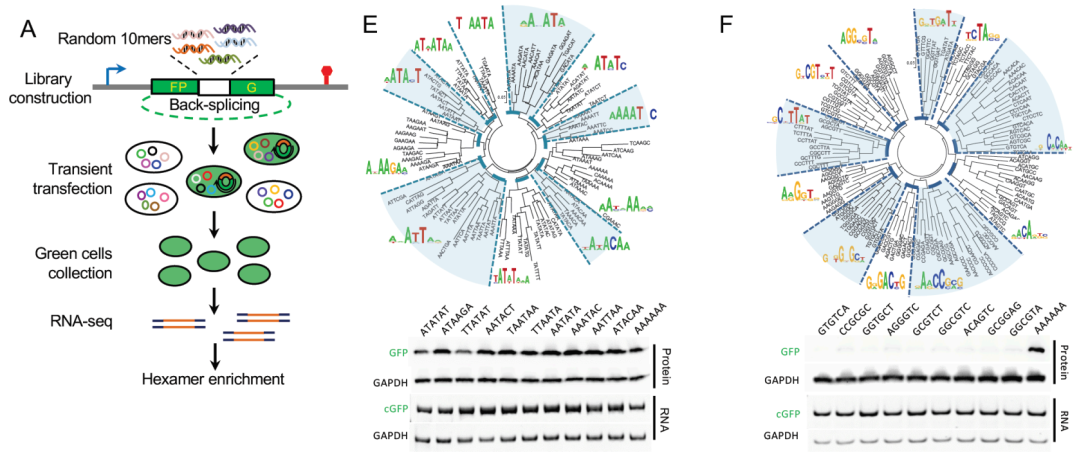
图14 circRNA中具有IRES-like功能的序列元件分析 ([15])
2.5 circRNA相互作用分子研究
分子间相互作用是生物分子发挥功能的重要方式,circRNA很多功能也是基于与其他分子的相互作用实现的,典型的包括竞争性结合miRNA的“miRNA Sponge”模型,与蛋白相互作用。2019年circRNA的研究的深度比此前有了很大的进步,绝大部分涉及到特定circRNA的研究都对其功能机制进行了分析。结合miRNA和结合蛋白的研究工作数量都非常大,下面主要选取最有代表性的研究工作进行介绍:
circRNA结合miRNA的研究
2019年4月26日,Cell杂志发表了加拿大多伦多大学Paul C. Boutros教授和Housheng Hansen He教授为共同通讯作者的circRNA研究工作,通过大样本测序和高通量shRNA文库筛选对局限性前列腺癌生长必须的circRNA (essential circRNA)。这项工作是首次利用高通量shRNA文库筛选功能性circRNA分子,这部分将在后面详细探讨。作者一共获得了171种局限性前列腺癌的essential circRNA。作者选择circCSNK1G3进行进一步的分析,作者发现circCSNK1G3可以竞争性结合miR-181b/d。他们的数据表明circCSNK1G3与miR-181b/d的表达数量大致在相同的水平(表达量相差太悬殊不能很好的起到竞争性结合抑制的作用!)。RIP和RNA pull-down实验证明circCSNK1G3可以结合miR-181b/d。干扰circCSNK1G3和过表达miR-181b/d后对CBX7,CDK1和CDC25A(miR-181b/d的靶基因)表达的影响相反。干扰circCSNK1G3后过表达miR-181b/d反而促进增殖,这说明circCSNK1G3结合miR-181b/d后并没有抑制miR-181b/d的活性,而降低circCSNK1G3则导致miR-181b/d无法抑制靶基因的表达([16])。这一结果表明circRNA与miRNA的结合并不仅仅通过抑制miRNA的功能这一种途径发挥作用,还存在其他作用机制。
推荐阅读:重磅! Cell杂志同期发表两篇circRNA研究文章
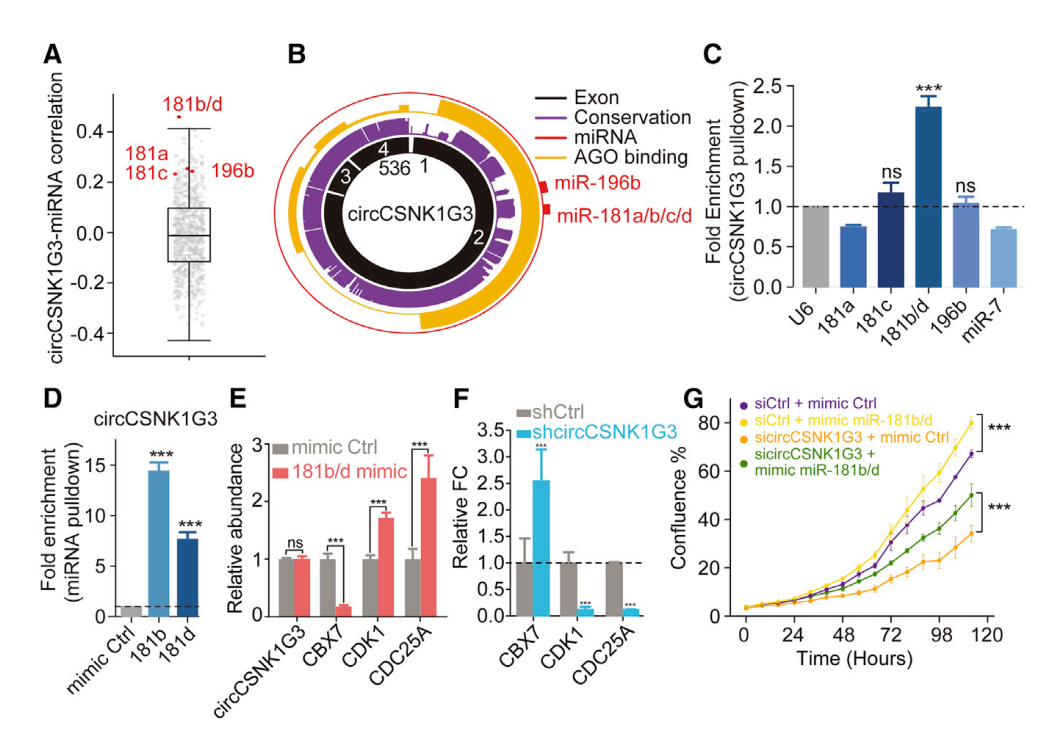
图15 circCSNK1G3结合miR-181b/d ([16])
circRNA结合蛋白研究
circRNA结合蛋白是另一种基于相互作用发挥功能的机制模型。2019年这方面也报道了很多重要的发现,包括circACC1与AMPK酶的结合,circYAP拮抗其YAP蛋白翻译过程等等。
2019年5月30日,Cell Metabolism杂志发表了一篇circRNA的研究论文,报道发现circACC1可以与AMPK的β和γ亚基相互作用,并促进AMPK的稳定性,同时参与维持AMPK基础活性。文章的通讯作者是中国科学技术大学的吴缅教授和安徽医科大学的胡汪来教授。文章先从筛选脂代谢相关基因来源的circRNA是否与脂代谢相关入手,找到了circACC1分子,然后进一步确认了circACC1与脂代谢的相关性,进一步分析了ACC1,AMPK等代谢酶和调控基因的表达和磷酸化状态和表达水平,发现了干扰circACC1能够降低ACC1,FPKFB3的磷酸化水平,同时也降低AMPK磷酸化水平及亚基的丰度。这些发现暗示了circACC1可能与AMPK的功能有关系,相互作用分析确认了circACC1与AMPK的β和γ亚基可直接相互作用,突变分析找出了它们相互作用的区段。作者还发现了血清饥饿可通过c-Jun激活circACC1的表达。最后肿瘤学功能分析表明circACC1具有促进肿瘤生长的作用,病人标本中也存在较高比例的circACC1高表达情况([17])。
推荐阅读:这个circRNA为什么能发表20分的文章?
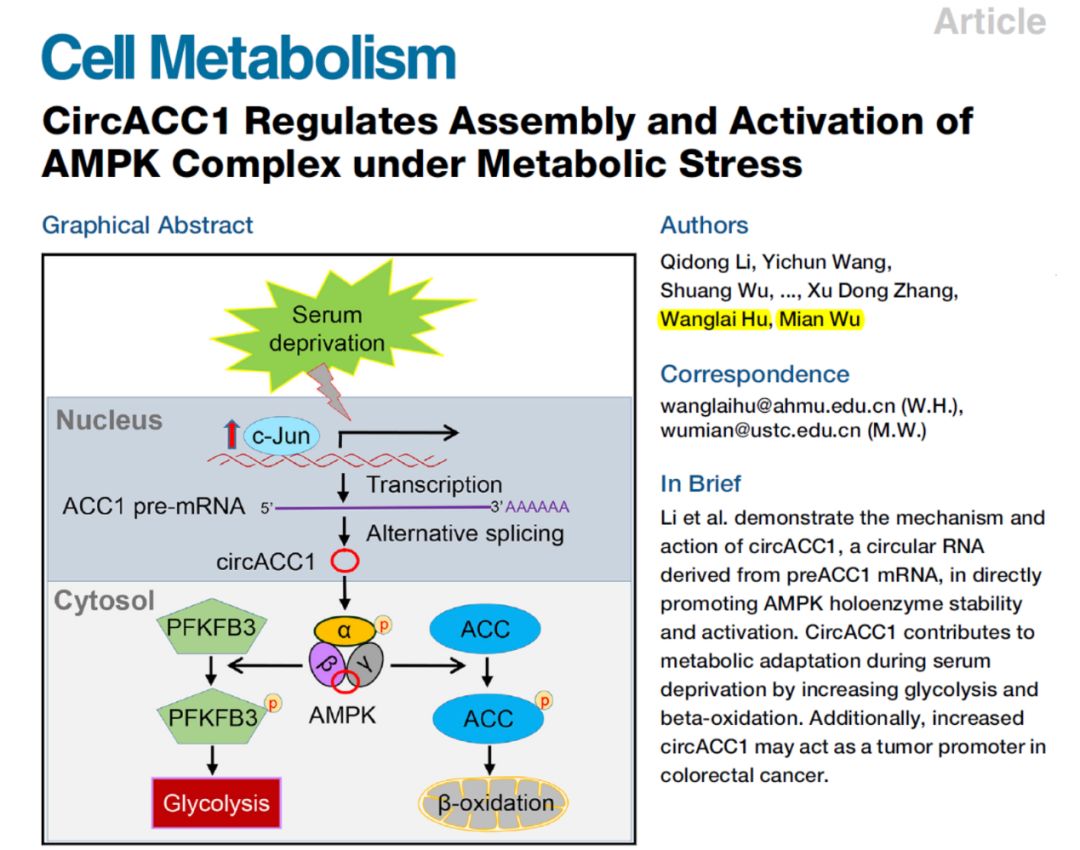
图16 circACC1与AMPK β和γ亚基相互作用 ([17])
2019年5月15日,加拿大多伦多大学杨柏华教授团队首次报道发现YAP基因来源的circRNA可拮抗YAP的mRNA翻译起始过程,调控其母基因的翻译效率。YAP是Hippo通路的关键分子之一,与肿瘤发展进程密切相关。本文作者发现来自YAP1基因的4-5外显子的circRNA,circYAP(circBank ID:hsa_circYAP1_008)可以通过结合翻译起始蛋白eIF4G和PABP,抑制母基因YAP的mRNA翻译起始,调控YAP蛋白的表达量,首次发现circRNA通过调控翻译起始效率调控母基因功能([18])。
推荐阅读:Nature子刊再发杨柏华教授新作:circYAP调控YAP蛋白翻译
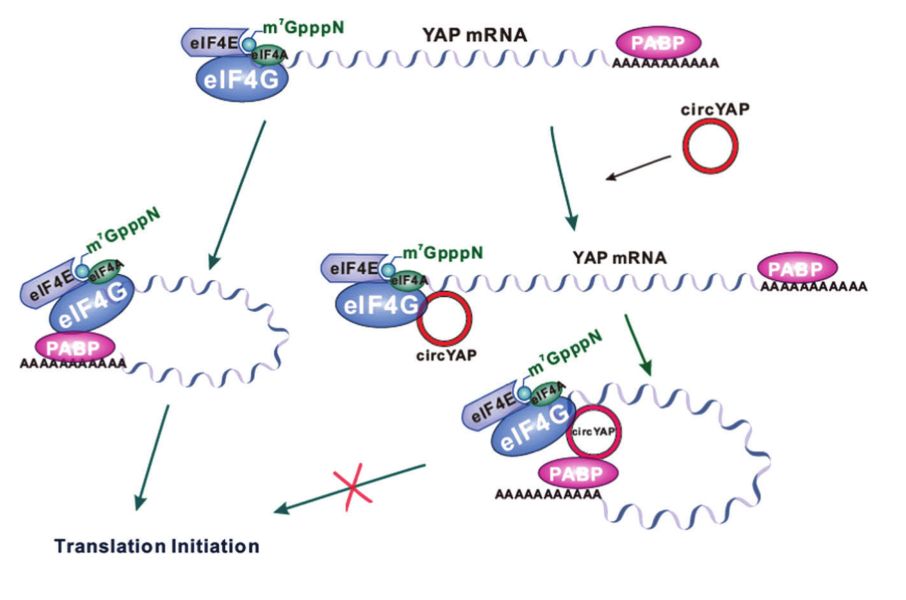
图17 circYAP调控YAP蛋白翻译 ([18])
2.6 circRNA降解机制研究进展
circRNA的降解机制此前的研究相对薄弱,2019年在circRNA降解机制方面有了很大的进步,首次报道m6A修饰可以通过募集RNase P/MRP复合物介导RNA的切割和降解,这一机制的底物可以是线性RNA,也可以是circRNA,此外还报道发现了果蝇GW182可以介导circRNA的降解,人类的三个同源基因(TNRC6A、TNRC6B和TNRC6C)也具有降解circRNA的作用。
m6A通过募集RNase P/MRP复合物介导circRNA降解
2019年4月2日,Molecular Cell杂志在线发表了一篇有关RNA降解的文章,介绍发现一种m6A介导的RNA降解机制。m6A识别蛋白YTHDF2结合靶分子,并募集HRSP12,进而介导RNA内切核酸酶RNase P/MRP复合物切割靶分子。这一过程可作用于mRNA和circRNA([19])。文章的通讯作者是韩国高丽大学的Yoon Ki Kim。
推荐阅读: m6A介导mRNA和circRNA降解
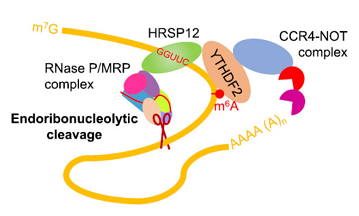
图18 m6A通过介导线性RNA和circRNA降解的机制 ([19])
果蝇GW182可调控circRNA的降解,人类的同源基因也有相似功能
2019年9月17日,Nature 子刊Cell Discovery在线发表了一篇circRNA降解机制的研究论文,报道发现GW182介导circRNA的降解作用。作者基于RNAi文库技术在果蝇DL1 和 S2细胞中进行筛选分析,文库由靶向31种已知的RNA代谢和降解作用有关的基因构成。通过QPCR检测dati和 laccase2基因的线性和环状RNA的稳定性状态的结果来分析靶分子是否参与circRNA的降解作用。最终发现GW182具有介导circRNA降解的作用。人类基因中GW182的同源基因有三个:TNRC6A、TNRC6B和TNRC6C。分别干扰三种基因后均可显著富集circRNA,说明TNRC6A/B/C均参与了circRNA的降解作用([20])。文章的通讯作者是重庆大学黄川教授。
推荐阅读:Cell Discovery | circRNA降解机制新进展
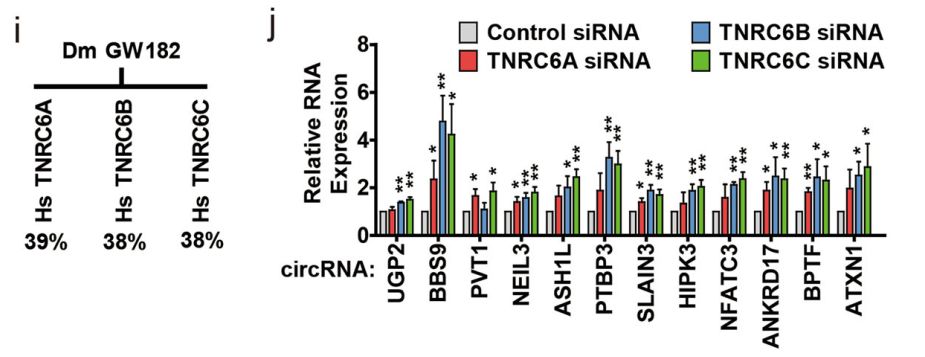
图19 人类TNRC6A、TNRC6B和TNRC6C可介导circRNA 降解 ([20])
2.7 circRNA与免疫效应
免疫系统是机体抵御病原体侵害的重要保障,很多病毒基因组以RNA形式存在,机体抵御外源RNA分子的免疫效应机制是一个重要的科学问题,目前已知机体主要通过RIG-I,TLR-3/7/8等受体对外源RNA分子进行识别,随后激活免疫系统,达到清除病毒的作用。。circRNA作为常见的RNA分子形式,在免疫通路中如何发挥作用此前还没有系统的研究报道。2019年关于circRNA与免疫效应关系的研究取得了不小的进展,包括首次报道发现circRNA特定结构在先天免疫调控中发挥重要作用,外源合成circRNA诱导细胞免疫效应通路激活机制的发现。
circRNA调控天然免疫通路
2019年4月25日,Cell杂志发表文章介绍发现circRNA参与细胞抗病毒免疫效应机制,该机制在系统性红斑狼疮等自身免疫疾病中发挥特殊作用。文中作者发现许多circRNA倾向于形成16-26bp不等的小颈环结构,该结构可结合并抑制双链RNA依赖性蛋白激酶R(double-stranded RNA-activated protein kinase,PKR),当细胞受到poly(I:C)刺激处理或病毒感染时,核酸内切酶RNase L激活并降解circRNA分子,PKR被释放并激活下游抗病毒效应机制。系统性红斑狼疮(Systematic lupus erythematosus,SLE)患者外周血单个核细胞(peripheral blood mononuclear cells,PBMC)中circRNA总体降低,PKR活性紊乱,但人为过表达circRNA有助于降低SLE患者PBMC中PKR的活性,将来或许有助于SLE等自身免疫疾病的治疗([21])。文章的通讯作者是陈玲玲教授,杨力教授和上海交通大学医学院附属仁济医院沈南教授。
推荐阅读:重磅! Cell杂志再发circRNA研究论文
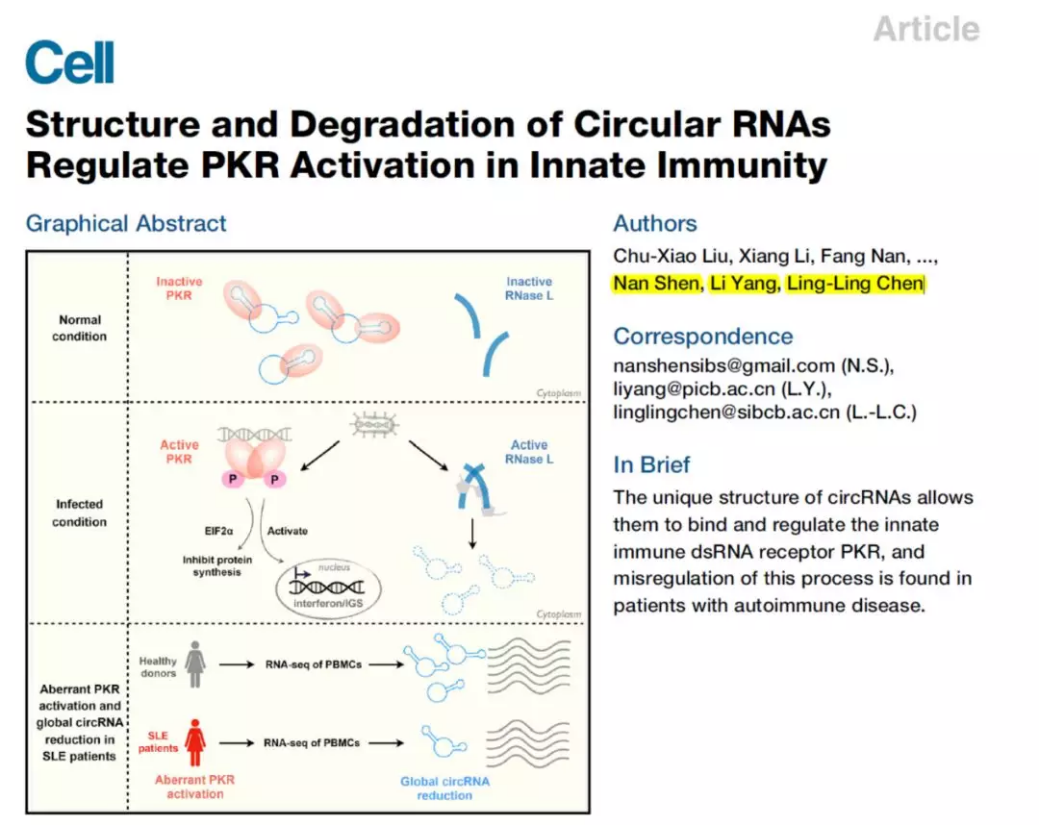
图20 circRNA特殊结构调控天然免疫效应通路 ([21])
外源circRNA与免疫效应通路的关系
早在2017年,Howard Y. Chang就曾经在Molecular Cell杂志报道细胞可以通过RIG-I识别细胞内生成的circRNA(内源circRNA)和体外合成环化的circRNA(外源circRNA),RIG-I识别外源circRNA后可激活自身免疫效应通路([22])(推荐阅读:Molecular Cell:细胞识别内源/外源circRNA的机制)。然而2019年3月19日发表在Molecular Cell的一篇文章却给出结论,外源合成的circRNA不诱发免疫效应通路,是很好的表达蛋白的工具([23])(推荐阅读:环化的RNA不诱发免疫反应,且可以高效翻译蛋白),这篇文章的通讯作者是麻省理工学院的Daniel G. Anderson教授。
但随后在8月30日Howard Y. Chang团队再次在Molecular Cell杂志发表文章证明了外源合成的circRNA确实可以诱导自身免疫效应相关的通路,他们发现细胞通过识别是否携带m6A修饰来识别内源和外源circRNA,外源的不带m6A修饰的circRNA可以激活内源性自身免疫效应相关的通路([24])。
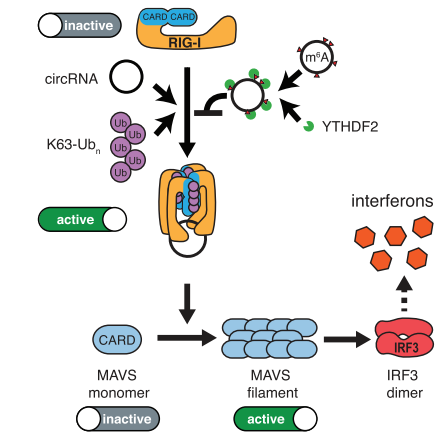
图21 circRNA通过RIG-I诱导先天免疫应答分子机制 ([24])
2.8 circRNA与表观遗传
表观遗传学是调控基因表达的重要机制,circRNA在表观遗传机制中的作用研究相对比较薄弱,2018年曾经有一篇研究文章报道circFLI1可调控启动子的甲基化修饰状态([25])。2019年在该方向取得了一些进展,包括发现超级增强子调控circNfix的表达。circHuR等核定位的circRNA通过调控相关转录因子的结合特性调控基因表达。
超级增强子调控circNfix的表达
2019年6月18日,Circulation杂志发表了南方医科大学南方医院宾建平团队的一项研究成果,报道超级增强子调控的circNfix缺失可诱导成年小鼠心肌梗死后心脏再生。该研究鉴定出与心脏再生相关的circRNA:circNfix,转录因子Meis1可结合在circNifx的超级增强子上,促进circNifx的高表达;circNifx表达下调可以促进心肌细胞的增殖和血管生成,而抑制心肌梗死后心肌细胞的凋亡,从而改善心肌功能和预后([26])。
推荐阅读:Circulation | CircNfix可削弱心脏再生的能力
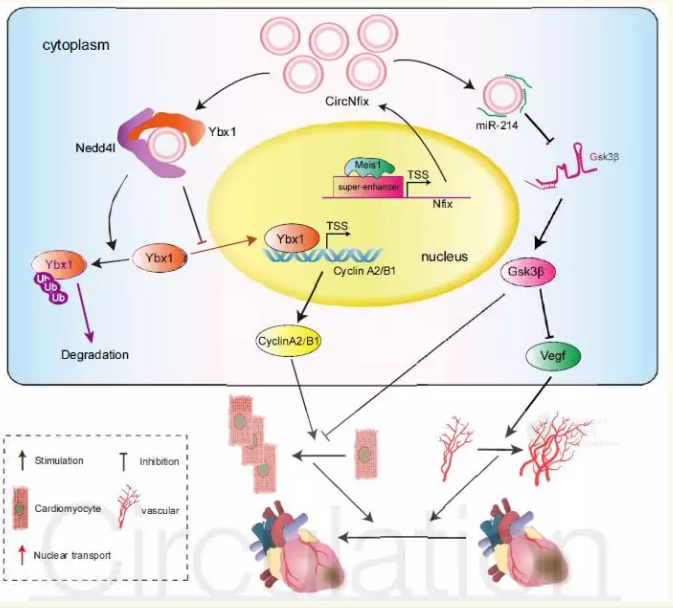
图22 circNfix减弱小鼠心肌梗死后心脏再生的作用机制 ([26])
核定位circRNA调控转录因子结合特性
转录因子结合到基因的启动子区并调控下游基因的表达,一些表观遗传调控的机制就是通过影响转录因子结合能力而实现对特定基因表达状态的调控的,如启动子区的DNA甲基化,组蛋白的甲基化,乙酰化等修饰状态。一些非编码RNA也可以通过影响转录因子的结合特性调控相关基因的转录状态,因此也是表观遗传调控的一种机制。2019年多篇文章报道circRNA也可以通过影响转录因子的结合特性实现对一些基因的表达状态调控,因此可以认为这些circRNA也参与了表观遗传行为。
2019年7月19日,Molecular Cancer杂志发表了桂林医学院附属医院 Li Bin为通讯作者的文章,介绍环状RNA circRHOT1通过启动NR2F6表达促进肝细胞癌的进程。作者基于在线数据分析和验证,发现circRHOT1在HCC中显著上调。研究者用生物素标记的circRHOT1特异性探针进行RNA pulldown实验,发现circRHOT1将TIP60募集到NR2F6启动子区并启动NR2F6的转录([27])。
2019年11月13日,华中科技大学同济医学院附属协和医院的童强松教授和郑丽端教授团队在Molecular Cancer杂志上发表了一项circRNA的研究工作,报道Circ-HuR(hsa_circ_0049027)可以与CNBP结合并抑制其与HuR启动子的结合,从而导致HuR的下调([28])。
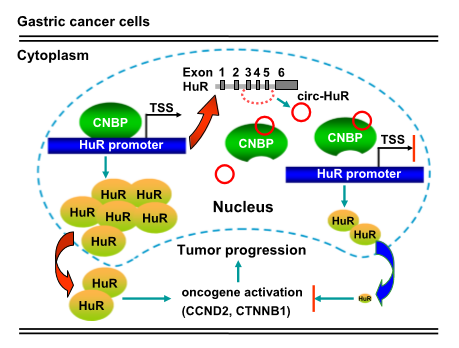
图23 circ-HuR结合CNBP阻止其与HuR启动子的结合 ([28])
2019年11月11日,EMBO Molecular Medicine杂志在线发表了华中科技大学同济医学院附属协和医院的童强松教授和郑丽端教授团队的一项circRNA研究工作,报道转录因子CUX1来源circRNA(circ-CUX1)通过与EWS RNA结合蛋白1(EWSR1)结合并促进EWSR1与MAZ(MYC-associated zinc finger protein)的相互作用,从而促进神经母细胞瘤细胞的有氧糖酵解,生长和侵袭,从而导致MAZ反激活以及 CUX1和其他与肿瘤进程相关的基因的转录变化([29])。
推荐阅读:童强松&郑丽端教授连发两篇10分circRNA研究论文
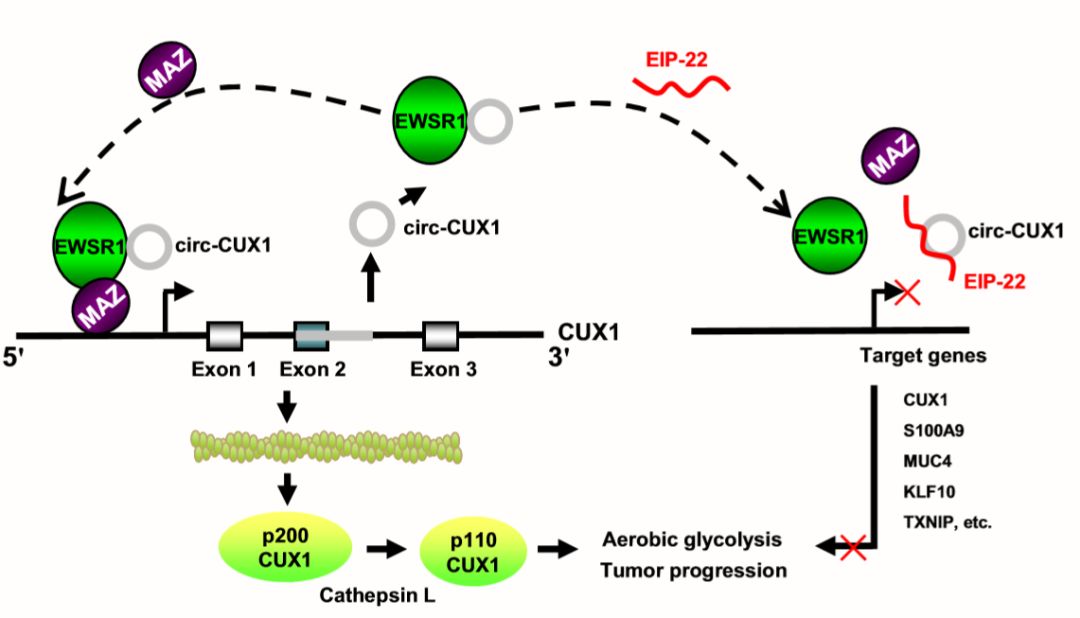
图24 circ-CUX1结合EWSR1,促进后者与MAZ的结合 ([29])
3. 人类疾病相关circRNA主要进展概述
3.1 肿瘤相关circRNA研究进展汇总
肿瘤一直是circRNA研究最活跃的疾病类型,2019年circRNA的研究论文和国家自然科学基金项目中,肿瘤学依然是绝对的主角。由于肿瘤相关circRNA研究体量非常巨大,完整的汇总需要非常大的篇幅,在此仅选择几项比较有代表性的研究工作:
肿瘤相关circRNA表达分析
2019年2月7日,Cell杂志在线发表了美国密歇根大学的Arul M. Chinnaiyan教授团队是一项工作,从外显子芯片数据分析了2000例,26种肿瘤中circRNA的表达情况,汇总形成了人类泛肿瘤circRNA表达数据,并构建了数据库MiOncoCirc(https://mioncocirc.github.io/) ([5])。
推荐阅读:重磅! Cell杂志同期发表两篇circRNA研究文章。
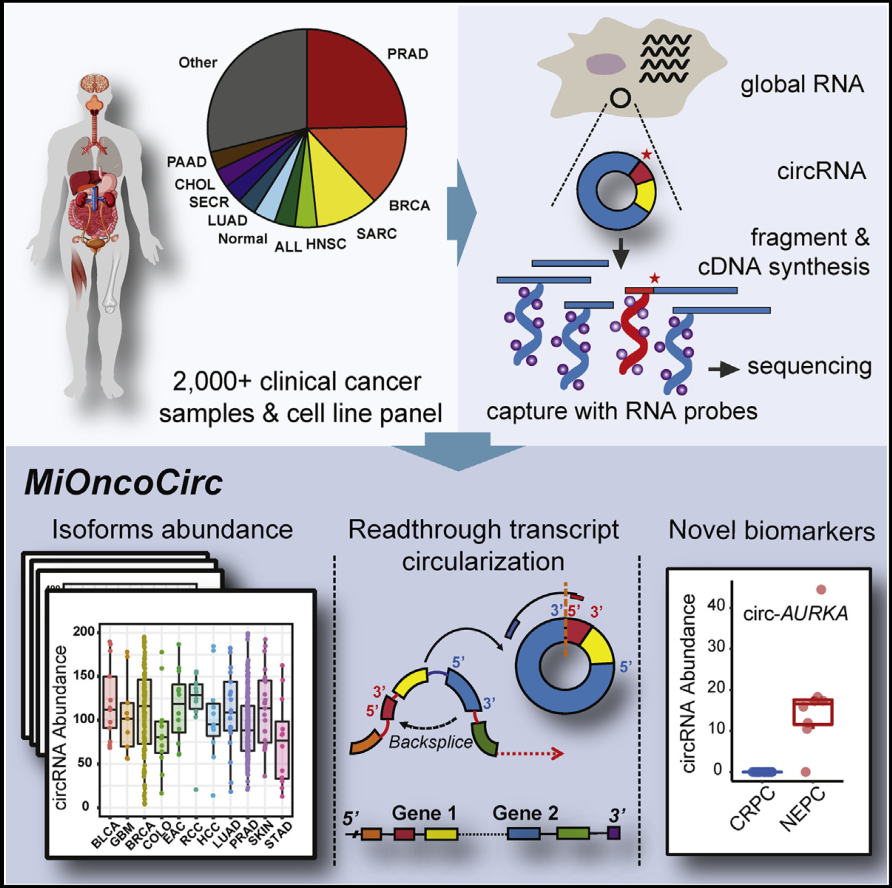
图25 人类泛肿瘤circRNA表达数据库 ([8])
circMYBL2是FLT3-ITD型AML潜在的治疗靶点
2019年8月18日,血液学顶级杂志Blood发表了一篇circRNA的研究论文,报道在FLT3-ITD型急性髓系白血病(AML)中circMYBL2通过结合PTBP1促进FLT3蛋白翻译。文章的通讯作者是中山大学的陈月琴教授。
FLT3-ITD是在FLT3基因中间的一段串联重复序列突变,该突变可导致Y591的自磷酸化,进而激发下游通路的过度激活,最终促进白血病的进程。FLT3-ITD突变型的AML病人预后较差且容易复发。FLT3的抑制剂可缓解这类白血病患者病情,但往往会因为FLT3基因的二次突变而导致耐药。因此,依然需要开发FLT3突变AML疾病新的治疗靶点。本文基于GEO数据分析,筛选到来自MYBL2基因的一个circRNA分子(circMYBL2),circMYBL2可以特异性的在FLT3-ITD阳性的AML细胞系中促进细胞增殖。机制方面,circMYBL2可通过结合PTBP1促进FLT3蛋白的翻译效率,进而促进疾病进程。有趣的是,作者的结果表明干扰circMYBL2对于FLT3抑制剂耐药的细胞也是有效的,说明circMYBL2是一个潜在的FLT3-ITD AML的治疗靶点([30])。
推荐阅读:Blood | circMYBL2是FLT3-ITD型AML潜在的治疗靶点
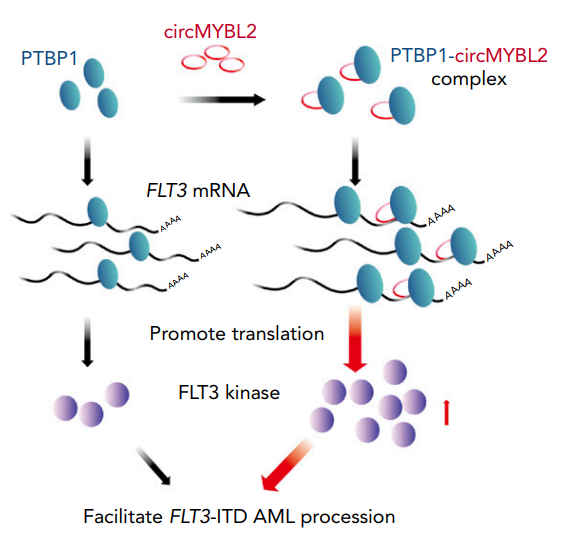
图26 circMYBL2结合PTBP1促进FLT3蛋白翻译 ([30])
Hepatology杂志发表多篇肝癌相关circRNA文章
2019年著名肝脏学杂志Hepatology发表了多篇circRNA的研究论文,报道肝癌相关circRNA的研究。
2019年4月20日, 南京医科大学附属鼓楼医院姜润秋和东南大学医学院王学浩为共同通讯作者在Hepatology在线发表了一项肝癌circRNA的文章,介绍发现circMAT2B通过调控miR-338-3p影响PKM2,促进肿瘤细胞糖酵解效应和肿瘤恶变([31])。推荐阅读:Hepatology | 肝细胞癌中circMAT2B促进糖酵解与肿瘤恶变
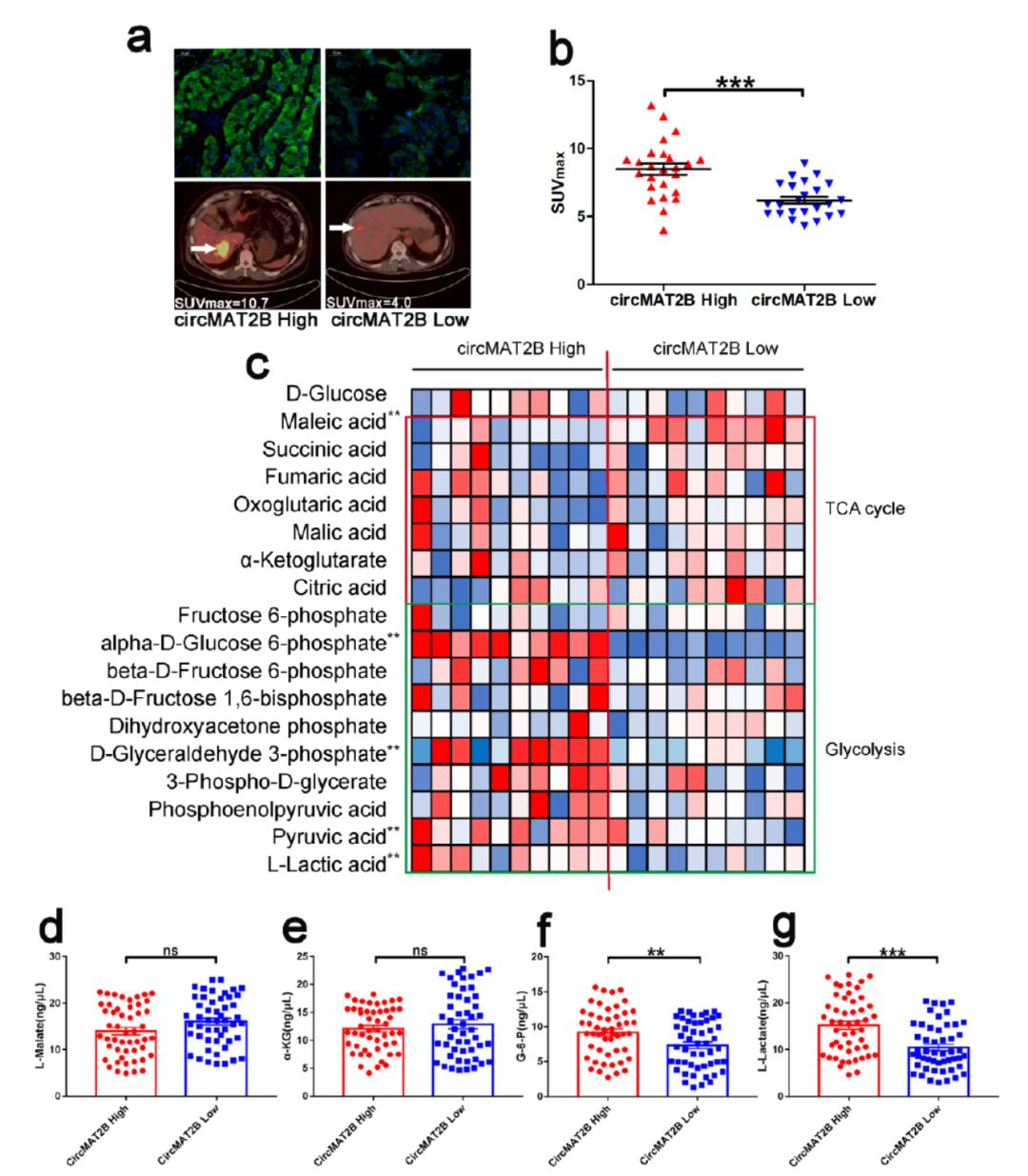
图27 circMAT2B与糖酵解有关 ([31])
2019年5月30日,第二军医大学东方肝胆外科研究所的王红阳院士和Li Liang研究员在Hepatology杂志发表文章,报道发现Circ-CDYL在肝癌早期诊断中的价值。作者对早期肝癌组织进行全面的mRNA, circRNA和microRNA表达谱分析,筛选到在早期肝癌中特异性表达上调的Circ-CDYL,体内和体外实验验证了Circ-CDYL调控 HDGF下游的NCL-PI3K-AKT和HIF1AN下游NOTCH2信号通路的活化,上调致癌蛋白c-myc和SURVIVIN表达,促进早期肝癌的发生发展。结合临床样本分析,验证联合Circ-CDYL、HDGF 和 HIF1AN三个生物标志物可有效诊断早期肝癌,提高了其诊断的灵敏性和特异性([32])。推荐阅读:HEPATOLOGY | Circ-CDYL在早期肝癌中特异性表达
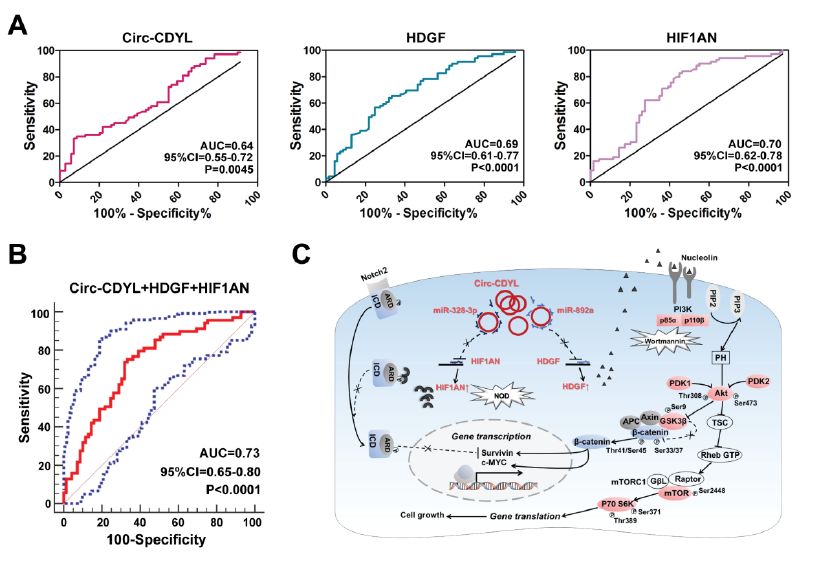
图28 Circ-CDYL 联合 HDGF、HIF1AN分子诊断早期肝癌 ([32])
2019年12月15日,复旦大学附属中山医院肝癌研究所周俭教授在Hepatology杂志发表文章,报道发现CircASAP1是肝细胞癌转移的关键调节因子。作者发现circASAP1与HCC患者根治性切除术后的肺转移有关,在miR-326 / miR-532-5p-MAPK1 / CSF-1信号转导中起着重要作用,可作为HCC患者的预后预测指标([33])。
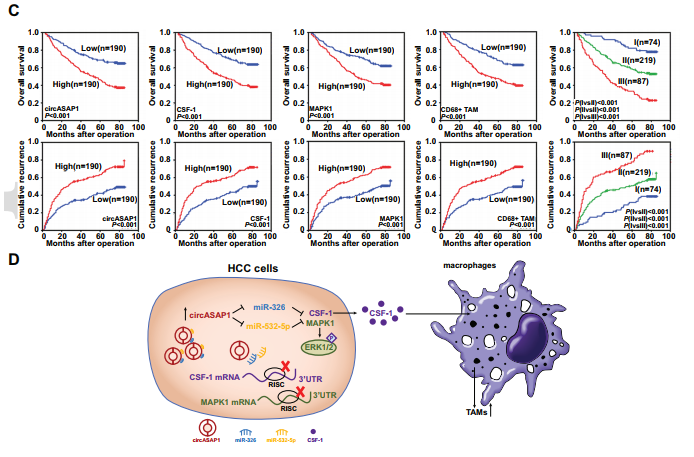
图29 CircASAP1在HCC中的表达与作用机制 ([33])
肠癌相关circRNA组合标志物cirScore
2019年9月2日,EMBO Mol Med在线发表一篇circRNA文章,来自中山大学附属肿瘤医院的研究团队采用高通量测序数据系统的鉴定和筛选了一批可用于中期结肠癌复发预测的circRNA,并在第三方数据中得到验证。中山大学附属肿瘤医院的徐瑞华教授为该文章通讯作者,鞠怀强副研究员,赵齐博士、王峰副主任医师、王梓贤博士及中山大学附属第六医院的兰平教授为文章共同第一作者。作者将筛选的circRNA进行线性组合,利用训练数据集构建了基于circRNA预测中期结肠癌复发的分子标签cirScore。性能评估表明,该指标可以在结肠癌患者中有效区分高危复发组和低危复发组。此外,作者还收集了本院122例和来自中山大学附属第六医院的180结肠癌病人样本作为内部验证集和外部验证数据集对标签性能进行验证。结果显示,该指标可以在外部的数据集中得到验证。通过比较,作者发现高cirScore的患者复发风险是低cirScore的2倍以上。且高cirScore的患者总生存期也显著长于低cirScore患者([34])。
推荐阅读:EMBO Mol Med|环状RNA可作为结肠癌复发预测分子标记物
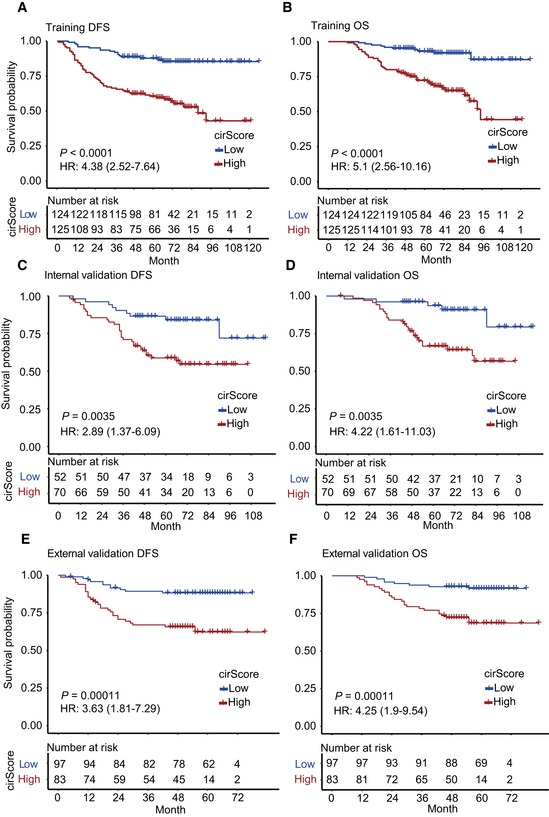
图31 cirScore用于肠癌临床预后分析 ([34])
3.2 心血管疾病相关circRNA研究进展汇总
2019年circRNA在心血管疾病方面也有一系列进展,下面选取几个有代表性的研究工作进行介绍:
circ_Lrp6调控VSMCs的功能
2019年2月15日,Circulation Research在线发表了意大利Humanitas Research Hospital的Gianluigi Condorelli和Leonardo Elia为共同通讯作者的文章,报道发现在血管平滑肌细胞(VSMCs)中Lrp6来源的circ_Lrp6可以通过竞争性结合miR-145上调其靶基因ITGβ8, FASCIN, KLF4, Yes1和Lox的表达,影响血管疾病的发病过程。在小鼠和人的血管疾病过程中,miR-145和circ_Lrp6的表达水平是存在差异调控的,而调节circ_Lrp6的表达可以影响到血管疾病的发展。在VSMCs中,circ_Lrp6具有促细胞增殖的效应,而miR-145具有促细胞分化的效应。当血管疾病发生发展的时候,内膜增生,血管平滑肌细胞中的miR-145表达下调,circ_Lrp6的表达可不发生改变。而当人为地敲低circ_Lrp6的表达,则上调miR-145的表达,进一步抑制内膜增生的程度,抑制血管变窄导致的后续血管疾病的发生发展([35])。
推荐阅读:circ_Lrp6调控VSMCs的功能
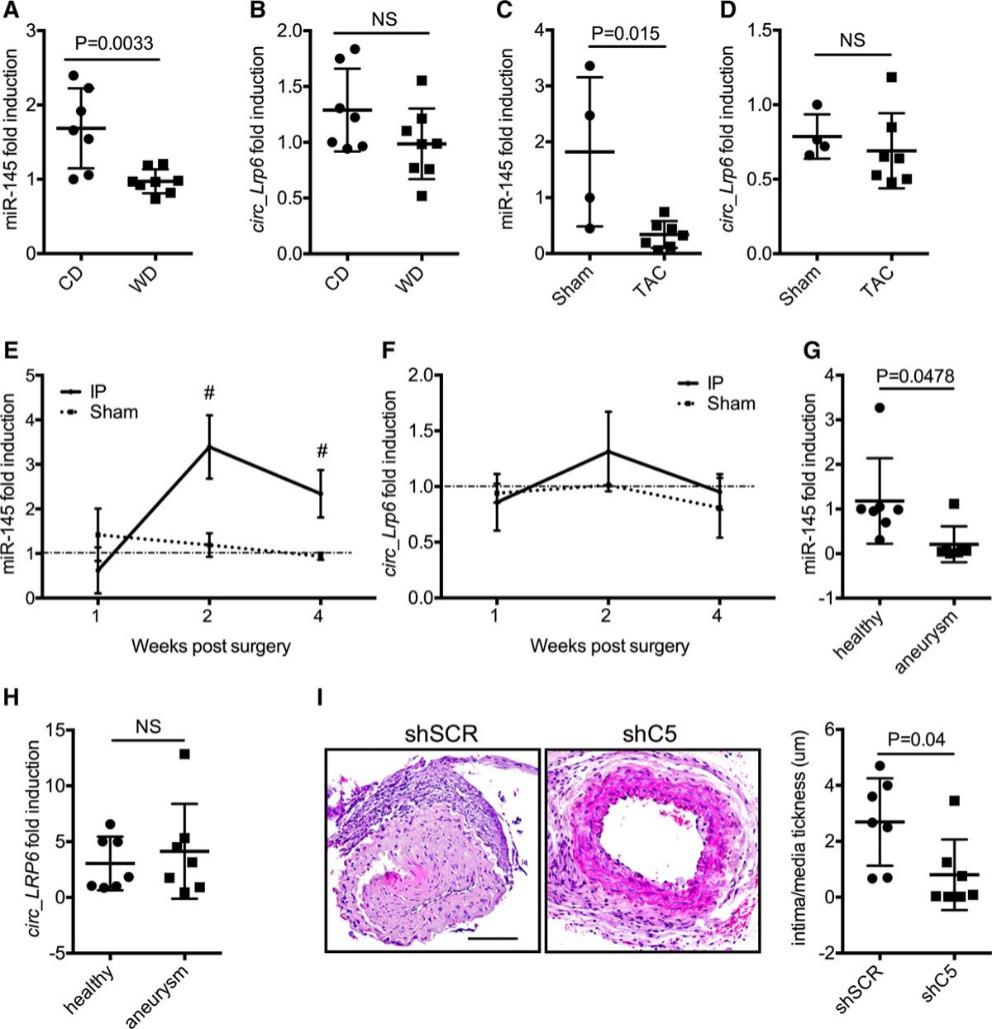
图32 Circ_Lrp6的组织表达和调控 ([35])
circFndc3b促进心肌修复
2019年9月20日,Nature Communications期刊在线发表了美国费城天普大学 Raj Kishore为通讯作者的文章,报道发现在发生心肌梗死MI的小鼠心脏中表达出现明显下调的circFndc3b,体内和体外结果均显示circFndc3b过表达有助于减轻心肌细胞和内皮的凋亡,改善心肌功能。进一步的机制研究,证明circFndc3b并不作为miRNA的海绵而发挥功能,而是与RNA结合蛋白FUS相互结合,正向调控VEGF-A的表达,从而改善梗死后心肌的功能和重建([36])。
推荐阅读:Nature Communications | circFndc3b调控FUS/VEGF-A信号通路,促进心肌修复
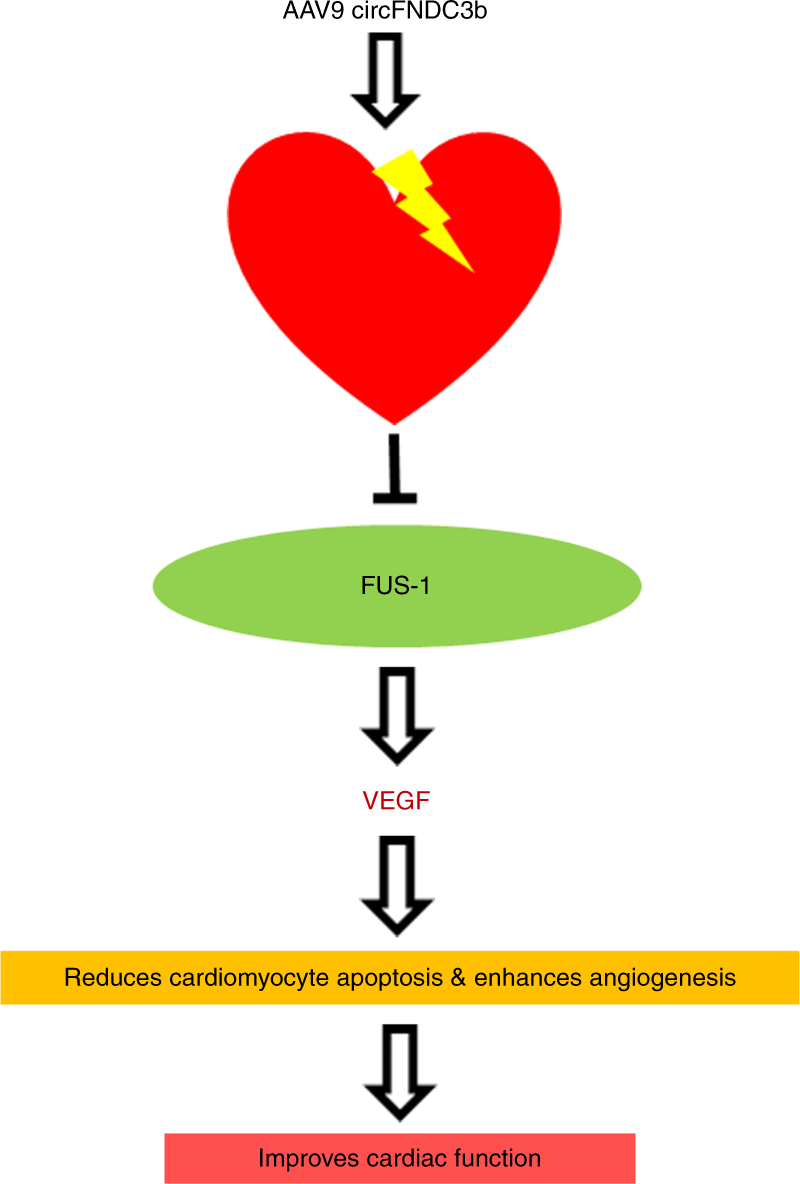
图33 circFndc3b介导的心脏修复的机制 ([36])
cPWWP2A改善糖尿病引起的视网膜血管病变
2019年4月9日,PNAS杂志在线发表了复旦大学上海医学院附属耳鼻喉眼科医院颜标研究团队的一项研究成果,报道发现cPWWP2A可以改善糖尿病引起的视网膜血管病变。该研究显示糖尿病应激上调视网膜周细胞,而不是内皮细胞中cPWWP2A的表达水平。体外研究表明,cPWWP2A充当内源性miR-579的海绵,调控Angiopoietin 1/Occludin/SIRT1基因的表达,直接调节周细胞生物学功能,而通过携带cPWWP2A的外泌体间接调节ECs生物学功能,促进ECs的迁移和血管形成能力。体内研究表明,cPWWP2A过表达或抑制miR-579表达可减轻糖尿病引起的视网膜血管功能障碍。相反,抑制cPWWP2A表达或过表达miR-579会加重视网膜血管功能障碍。这项研究揭示了周细胞和ECs “对话”的新机制,干预cPWWP2A或miR-579的表达可为治疗糖尿病微血管并发症提供治疗策略([37])。
推荐阅读:cPWWP2A改善糖尿病引起的视网膜血管病变
3.3 其他人类疾病相关circRNA研究进展汇总
除了肿瘤和心血管疾病,其他类型的人类疾病也有不少circRNA的研究工作,也是由于体量较大,这里仅选择在神经性疼痛中探索circAnks1a机制的研究工作,这个故事中circRNA既有miRNA Spong的功能,也可以结合蛋白,因此是非常有趣,也非常典型的circRNA研究工作,因此选这个文章给大家做介绍。
circAnks1a在神经性疼痛中的作用
2019年9月11日,Nature Communications在线发表了一篇circRNA的研究文章,报道脊髓中circAnks1a在神经性疼痛的啮齿类模型中调节超敏反应。文章的通讯作者是中山大学医学院信文君教授,徐婷和中山大学孙逸仙纪念医院马超教授。本文主要基于大鼠脊神经结扎的模型分析了差异变化的circRNA,找到了circAnks1a,并验证了circAnks1a与神经性疼痛的相关性。机制方面作者发现circAnks1a可以促进YBX1的入核和结合VEGFB启动子,促进其表达,还可以通过竞争性结合miR-324-3p调控VEGFB的表达。两种机制共同促进了VEGFB的表达,最终介导神经性疼痛的生理过程([38])。
推荐阅读:Nature Communications | 神经性疼痛模型中circRNA参与调节超敏反应
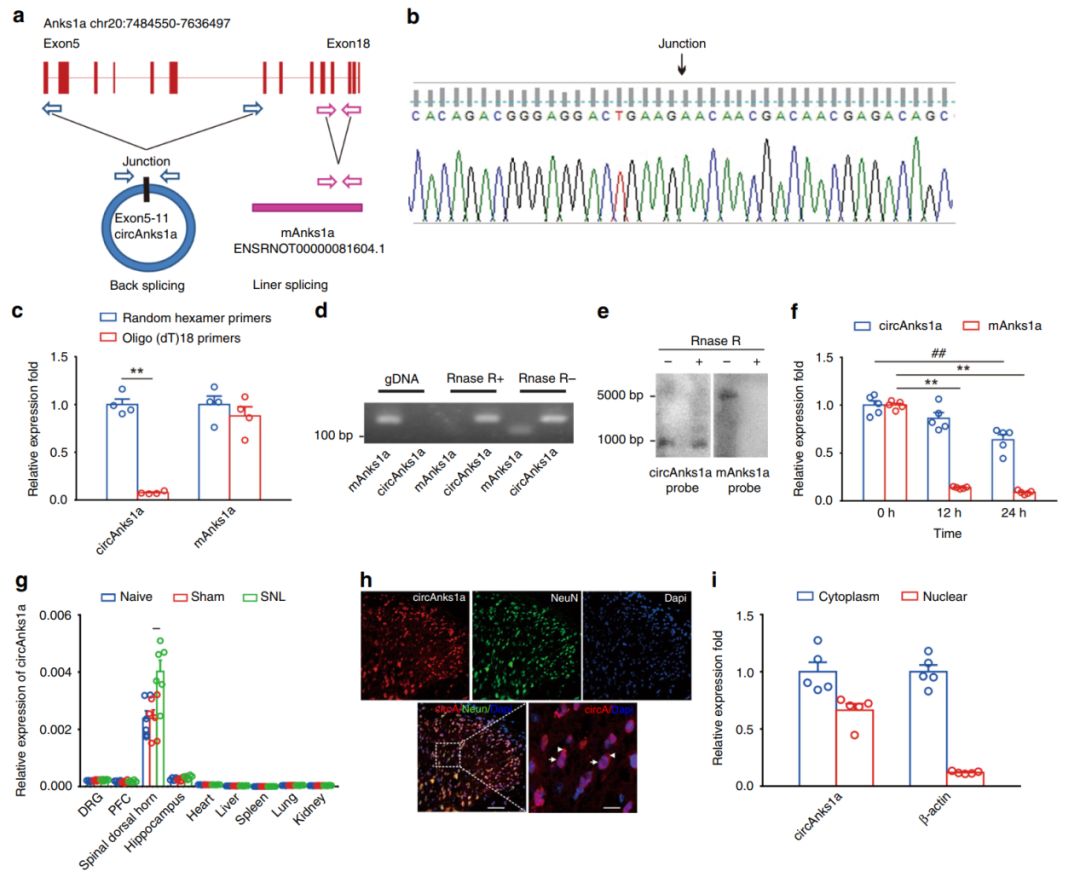
图34 circAnks1a的验证和分析 ([38])
4. circRNA研究工具与方法进展
2019年circRNA取得了骄人的成绩,也涌现出一批新的circRNA研究技术方法和工具,这一部分我们将梳理一下这方面的主要进展。
4.1 circRNA研究新技术方法
靶向circRNA分子的高通量shRNA文库技术
2019年4月26日,Cell杂志发表了加拿大多伦多大学Paul C. Boutros教授和Housheng Hansen He教授为共同通讯作者的circRNA研究工作,通过大样本测序和高通量shRNA文库筛选对局限性前列腺癌生长必须的circRNA (essential circRNA)。作者首先通过对144对前列腺癌/癌旁配对标本进行深度测序,分析得出前列腺癌特异性的circRNA,挑选表达丰度靠前的2000个circRNA设计shRNA,为减少脱靶效应带来的假阳性结果的干扰,作者在这些circRNA所对应的mRNA中也设计了对照shRNA。最终所获得的shRNA共靶向1507种circRNA和对应来源基因的1075种mRNA的shRNA文库。经过负选择筛选分析,最终获得了171种前列腺癌相关的essential circRNA分子([16])。
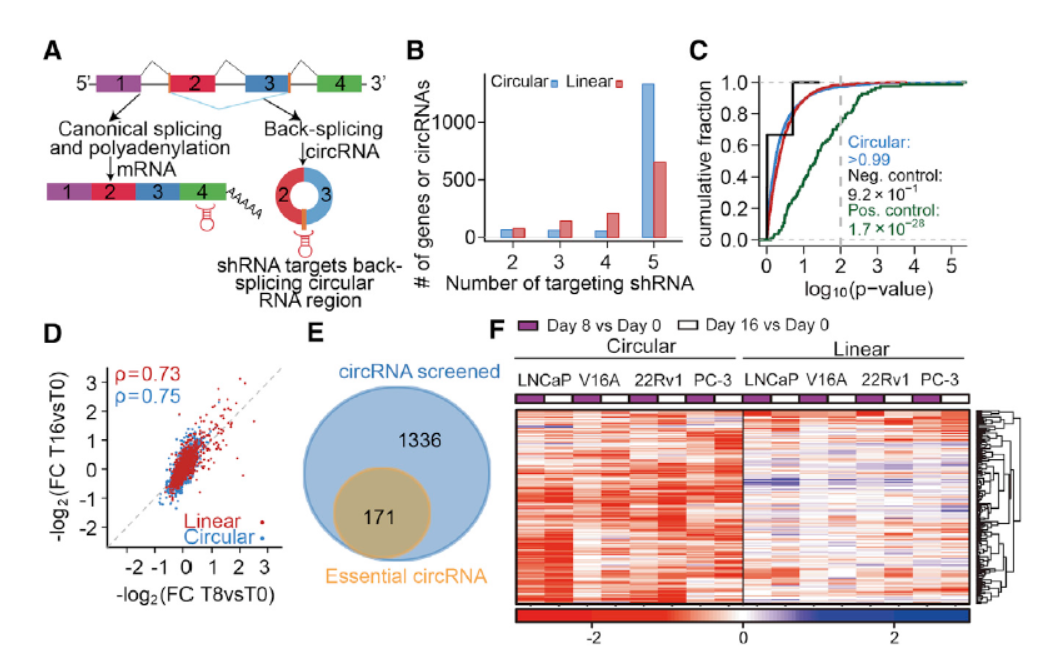
图35 高通量shRNA文库筛选前列腺癌essential circRNA ([16])
circRNA敲除体系
2019年1月15日,Nature Immunology在线发表了中国科学院生物物理研究所田勇教授和范祖森教授为共同通讯作者的文章,介绍发现来自Pan3基因的环状RNA circPan3在IL-13调控的肠道干细胞自我更新过程中发挥重要调控作用。文章构建了circPan3的敲除模型。小鼠中不存在Alu序列,促进circRNA形成的往往是一些SINE元件,因此作者需要首先确认需要打靶的circRNA是由什么序列介导形成circRNA的。作者的策略是在小鼠的Lgr5+ISCs细胞中通过慢病毒载体构建mini-gene,模拟体内成环的过程。基于这一技术路线,可以通过构建删除特定内含子片段分析介导所感兴趣的circRNA由哪些序列元件介导的。作者就是通过这种技术路线找出了介导circPan3生成的内含子序列,如下图中a所示的#1和#2位置,Northern杂交也证明了删除#2的mini-gene不再表达circPan3。有了这个基础才能设计出专门针对circPan3的敲除方案,作者设计了靶向#2位置的打靶体系,最终获得了准确删除该位点的小鼠,经过鉴定,该小鼠中circPan3被准确敲除,与此同时,Pan3基因的其他转录产物(下图f中靠上侧的一坨)没有受到明显的影响,Pan3蛋白也没有明显的变化([39])。
推荐阅读:环状RNA circPan3调控肠道干细胞自我更新
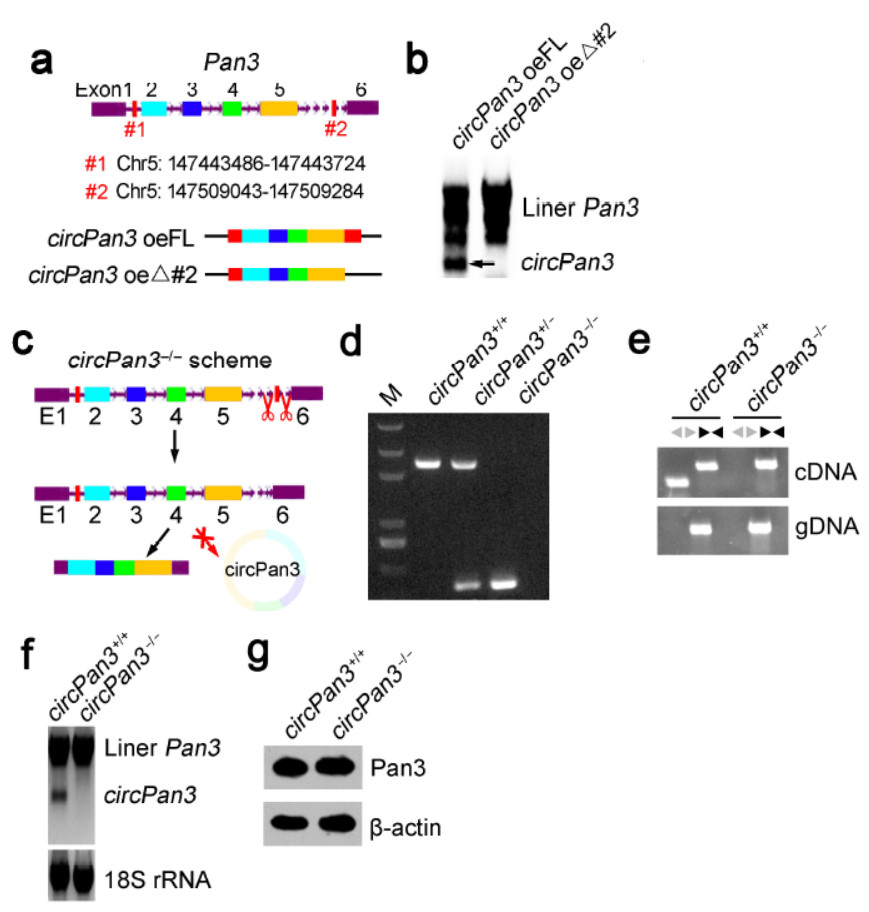
图36 circPan3敲除模型构建策略([39])
基于环状RNA高效表达核酸适配体
2019年4月8日, Nature Biotechnology在线发表了洛克菲勒大学Samie R. Jaffrey教授为通讯作者的文章,报道开发一种基于circRNA高效表达核酸适配体(aptamer)的技术体系。该环状RNA表达体系被命名为“Tornado”, 该技术是基于具有自剪切功能的核酶(Ribosome)实现待环化序列的上下游切割,环化作用则借助于内源的RNA连接酶RtcB实现,最终的产物包含了待环化序列以及为实现环化而携带的Ligated stem序列(racRNA) ([40])。Tornado环状RNA过表达体系具有自动剪切,自动环化,表达高效且可实现适体体内过表达的优点,为环状RNA的过表达开辟了新途径。
推荐阅读:新型环状RNA表达载体
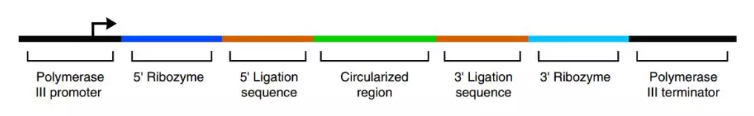
图37 Tornado环状RNA过表达载体 ([40])
改良的基于RNase R富集circRNA技术
2019年9月19日,Nucleic Acids Res杂志发表了一种circRNA富集纯化技术的文章,报道一种改良的基于RNase R富集circRNA的技术方法。该研究中,作者证明涉及RNase R的标准方案> 20%高表达的线性RNA不能消化,但这些缺点可以在很大程度上克服。具有高度结构化的3’末端的RNA(包括snRNA和组蛋白mRNA)天然地对RNase R具有抗性,但是一旦将poly(A)尾添加到其末端,就可以有效降解。此外,RNase R在许多多腺苷酸化mRNA的体内停滞,特别是在以前注释为G-四链体(G4)结构的富含G的序列中。在用反应缓冲液中的Li +代替K +(其稳定G4s)后,作者发现RNase R能够将这些序列进行并完全降解mRNA。该研究的结果为目前用于分离环状RNA的方法提供了重要的改进,以及揭示可以天然抑制细胞核酸外切酶降解RNA结构的方法([41])。文章的通讯作者是美国宾夕法尼亚大学佩雷尔曼医学院的Jeremy E. Wilusz。
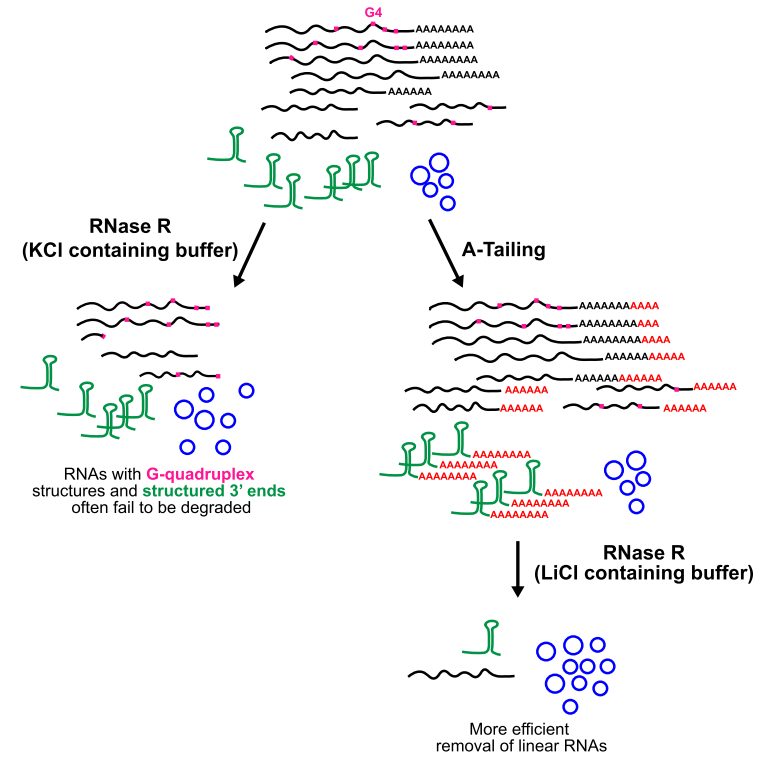
图38 改良的基于RNase R富集circRNA技术 ([41])
4.2 circRNA研究的信息学工具与数据库
CircSplice新工具全面挖掘癌症特异性circRNA选择性剪切事件
2019年3月8日,Molecular Cancer杂志在线发表了武汉大学何春江教授团队开发了一种全新的从头算法工具CircSplice,它可以识别circRNA中的内部选择性剪接,并比较不同条件之间的差异circRNA剪接事件。通过CircSplice对透明细胞肾细胞癌和膀胱癌数据挖掘后,分别在两个数据集中检测到4498和2977个circRNA可变剪接(circ-AS)事件,并通过RT-PCR证实了circ-AS事件的表达。研究者进一步检查了癌症和邻近正常组织中circ-AS的分布和模式,为了进一步了解癌症特异性circ-AS的潜在功能,将这些剪切事件分类为肿瘤抑制因子和癌基因,进行了信号通路富集分析。该研究开发的工具可首次全面了解癌症特异性circRNA选择性剪接,对癌症中circRNA的调控和功能研究有重要贡献([42])。
该工具网址链接http://gb.whu.edu.cn/CircSplice或https://github.com/ GeneFeng / CircSplice
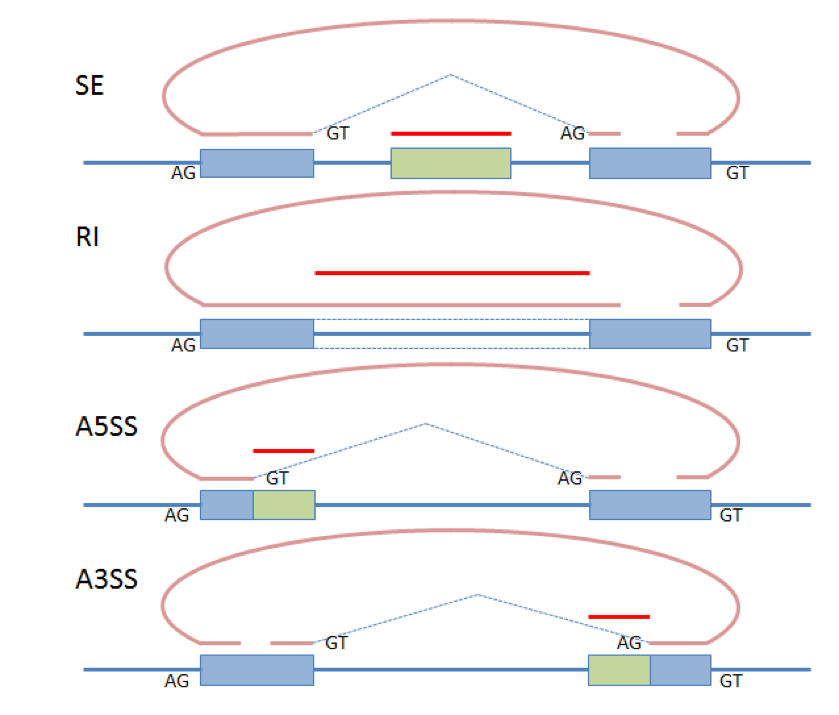
图39 CircSplice支持鉴定四种circRNA可变剪切事件类型 ([42])
circAtlas: 多物种circRNA表达数据库
2019年3月19日,Cell Reports杂志发表了中国科学院北京生命科学研究院赵方庆研究员团队的一项研究成果,利用高通量测序技术对人、猴和小鼠的44个正常组织进行转录组评估,鉴定出大量circRNA分子,并重建72.6%的circRNA全长序列,为circRNA的可变剪切、保守性以及与线性RNA的关系研究提供了重要数据资源。该研究成果已汇总到circAtlas数据库中。circAtlas数据库提供界面友好的数据检索和呈现方式,包括了多项circRNA资源,如circRNA及其宿主基因在各组织中表达值,miRNA和RBP(RNA结合蛋白)预测信息;circRNA在人、猴、小鼠、大鼠、猪、鸡和狗7物种中保守性;circRNA蛋白翻译相关的ORF和IRES信息;文献报道疾病相关的circRNA收录等信息([43])。
推荐阅读:circAtlas: 多物种circRNA表达数据库
图40 circAtlas数据库 ([43])
Ularcirc: circRNA分析及可视化工具
2019年11月18日,在线发表了澳大利亚张任谦心脏研究所David T. Humphreys为通讯作者的文章,报道Ularcirc工具。该文献作者开发的Ularcirc的软件工具,它集成了识别反向剪接位点和正向剪接位点的可视化功能,并能对鉴定的circRNA进行下游分析。 Ularcirc可利用CIRI、circExplorer或STAR比对软件的原始输出结果,组装环状RNA的反向剪接位点(BSJ)计数表,并允许多样本分析。研究者使用Ularcirc工具分别从公共和内部生成的数据集中识别和表征circRNA,并演示了它如何用于(i)发现新的亲本转录本剪接模式,(ii)检测circRNA的内部剪接模式,(iii)以及揭示反向剪接位点(BSJ)形成的复杂性。 此外,该研究还确定了circRNA可能具有比亲本转录本线性序列更长的开放阅读框。最后,我们检测并验证了由ApoA4转录本产生的一类新型环状circRNA的存在,其反向剪接位点(BSJ)来自编码外显子内的多个非经典剪接位点([44])。
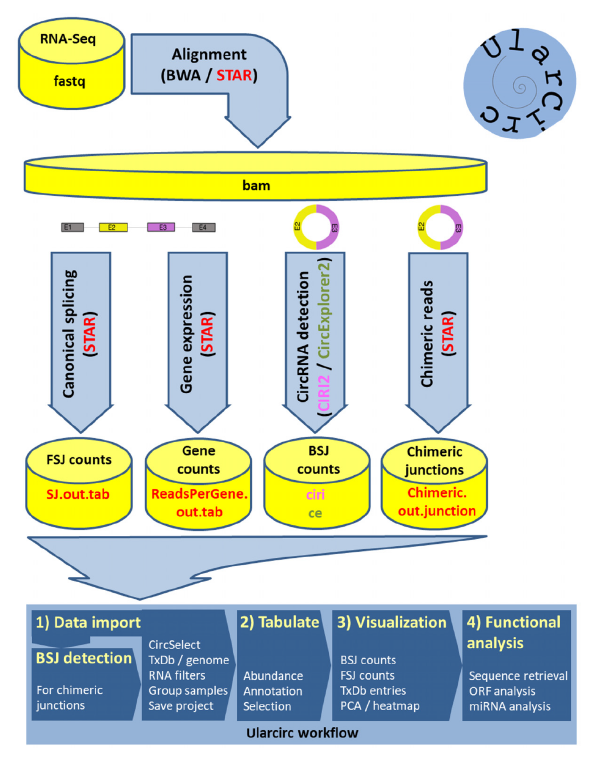
图41 Ularcirc工具集工作流程([44])
环状RNA在约1000种人癌细胞系中的综合表征研究
2019年8月26日,Genome Medicine杂志发表了德州大学休斯敦健康科学中心韩冷教授团队的一项工作,报道分析了超过1000种人类癌症细胞系中circRNA表达分析的结果。人癌细胞系是癌症研究和开发治疗策略的基本模型。然而,目前在许多癌细胞系中并没有表征环状RNA(circRNA)的数据。该研究中,研究者对CCLE(Cancer Cell Line Encyclopedia,癌症细胞系百科全书)数据库中的RNA-seq数据,应用四种circRNA识别算法分析了约1000种人癌细胞系中circRNA的表达谱特征,还综合分析探索不同癌症谱系中circRNA的表达情况,生物形成,细胞功能和药物反应等。研究结果揭示了circRNA在不同癌症细胞中具有高度特异性表达模式,表明circRNA可能是癌症治疗中强有力的诊断或预后标志物。研究者还通过实验鉴定了涉及circRNA生物形成的关键基因,证实TGF-β信号通路在该过程中起到重要作用。更引人注目的是,研究者发现一些癌症临床上常见基因上富集RNA结合蛋白(RBP),更可能产生circRNA。其中,circMYC可促进细胞增殖。我们观察到circRNA的表达与药物反应之间的强烈关联,特别是那些靶向染色质组蛋白乙酰化的药物。同时,该研究还开发了一个癌细胞系circRNA数据库——CircRiC(https://hanlab.uth.edu/cRic),该数据库目前包含了来自CCLE的22个癌症谱系中935个癌细胞系中circRNA的表达特征、生物形成、药物应答和整合分析等数据,对circRNA研究提供了非常有意义的参考([45])。
推荐阅读:Genome Medicine:逾千种肿瘤细胞系circRNA表达谱
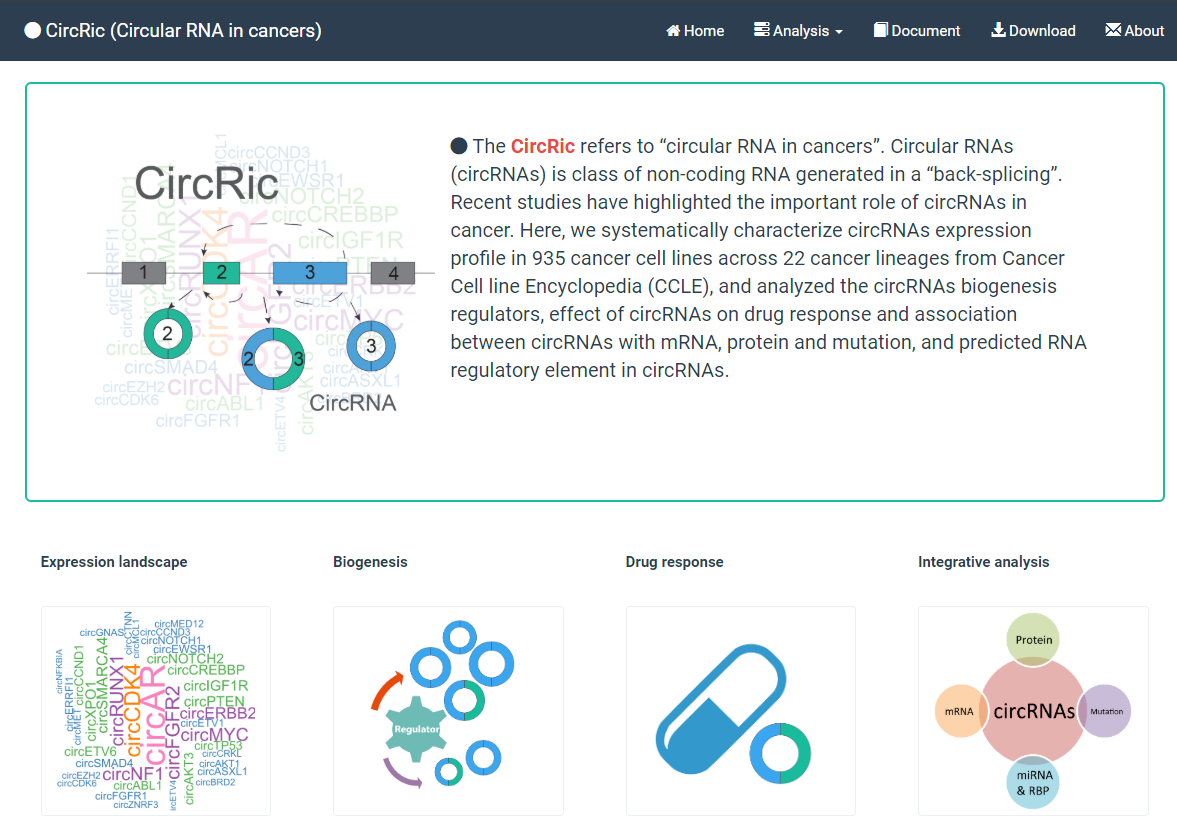
图42 circRic数据库主页 ([45])
NPInter v4.0:非编码RNA互作数据库。
2019年10月31日,Nucleic Acids Res发表了更新版的NPInter v4.0数据库,文章的通讯作者是中国科学院生物物理研究所陈润生院士,何顺民研究员和中国科学院计算技术研究所赵屹研究员。非编码RNA(ncRNA)在多种生物信号通路中起着至关重要的调控作用。已有研究表明非编码RNA可以与蛋白、RNA以及基因组相互作用,调控复杂生物过程。陈润生院士课题组早在2006年发布了NPInter数据库,近日该数据库已更新至第4版,NPInter(NPInter v4.0)数据库系统地收录了绝大多数种类非编码RNA(新增circRNA)的相互作用,并对相互作用以及相关分子进行了详细的注释以及可视化,提供了一个全面、系统的非编码RNA相互作用的研究平台。环状RNA(circRNA)的相互作用和ChIRP-seq获得的非编码RNA-基因组的相互作用首次被收录进来。总数据量上升到11万条,涵盖了35个物种。相互作用以及作用分子还增加了疾病注释,可帮助研究者更好地理解相互作用的功能。除了整合数据之外,整个数据库的网站也进行了重新构建,提高了原有搜索模块的易用性([46])。数据库网址为:http://bigdata.ibp.ac.cn/npinter4
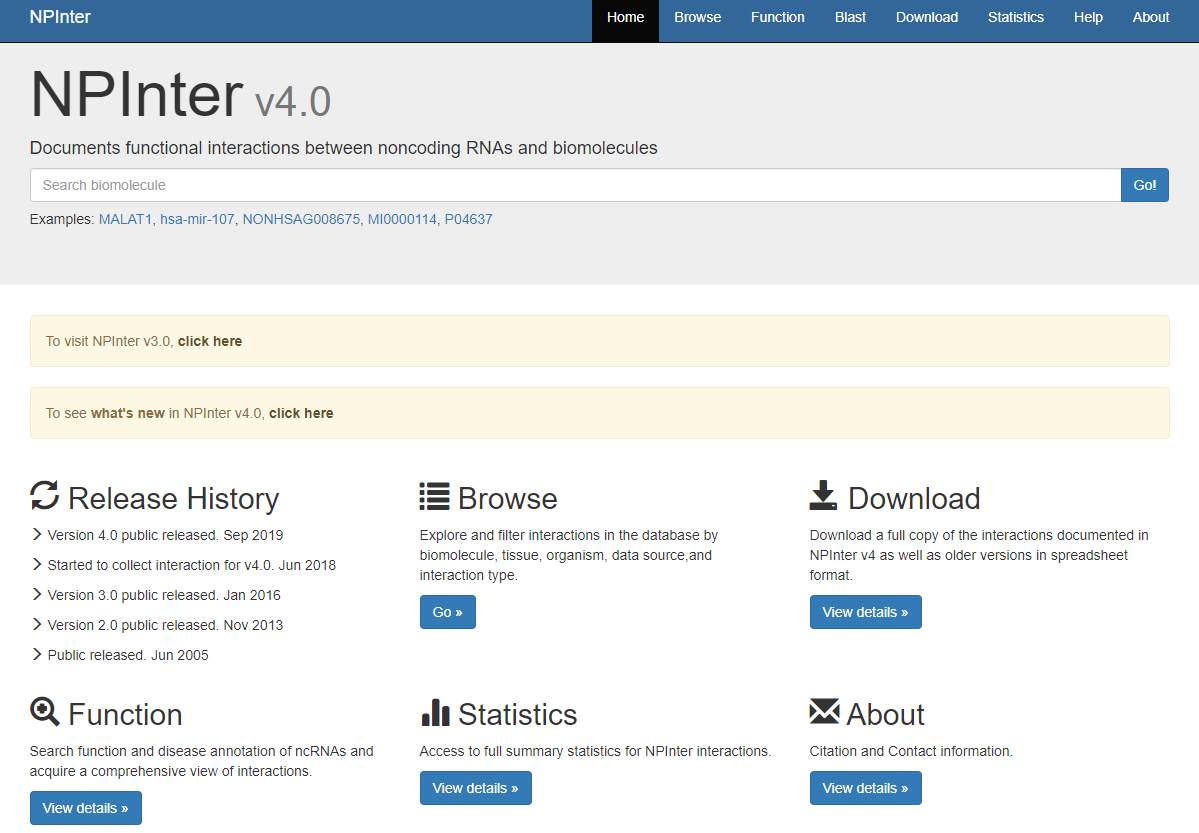
图43 NPInter v4.0数据库首页 ([46])
5. 非医学相关circRNA研究进展
非医学相关的circRNA研究总体体量比较小,受众面也比较窄,常见的研究主要基于表达谱分析和基于miRNA的互作网络预测分析,因此选择下面这个工作作为代表进行介绍:
EpCAM敲除小鼠circRNA表达谱分析
2019年7月8日,International Journal of Molecular Medicine杂志在线发表了中科院广州生物医药与健康研究院赖良学教授和广东中医药大学广东省中西医结合代谢病研究中心郭姣教授为共同通讯作者的文章,报道分析了EpCAM敲除老鼠中circRNA的表达状态。作者利用Cas-9技术构建了EpCAM敲除老鼠,并分析了肝脏中非编码RNA的表达情。测序的结果显示11个上调和12个下调的circRNA([47])。
图44 EpCAM敲除老鼠肝脏circRNA表达分析 ([47])
6. circRNA重要综述
Nature Reviews Genetics:circRNA的生成和特征
2019年8月8日,Nature Reviews Genetics发表了一篇circRNA的综述文章,系统回顾介绍了circRNA研究的主要进展,包括circRNA的形成,检测方法,主要功能机制,主要研究技术等。文章的通讯作者是丹麦奥胡斯大学的Lasse S. Kristensen,其中著名circRNA研究专家Jørgen Kjems教授是本文的共同作者。
推荐阅读:Nature Reviews Genetics发表circRNA重要综述
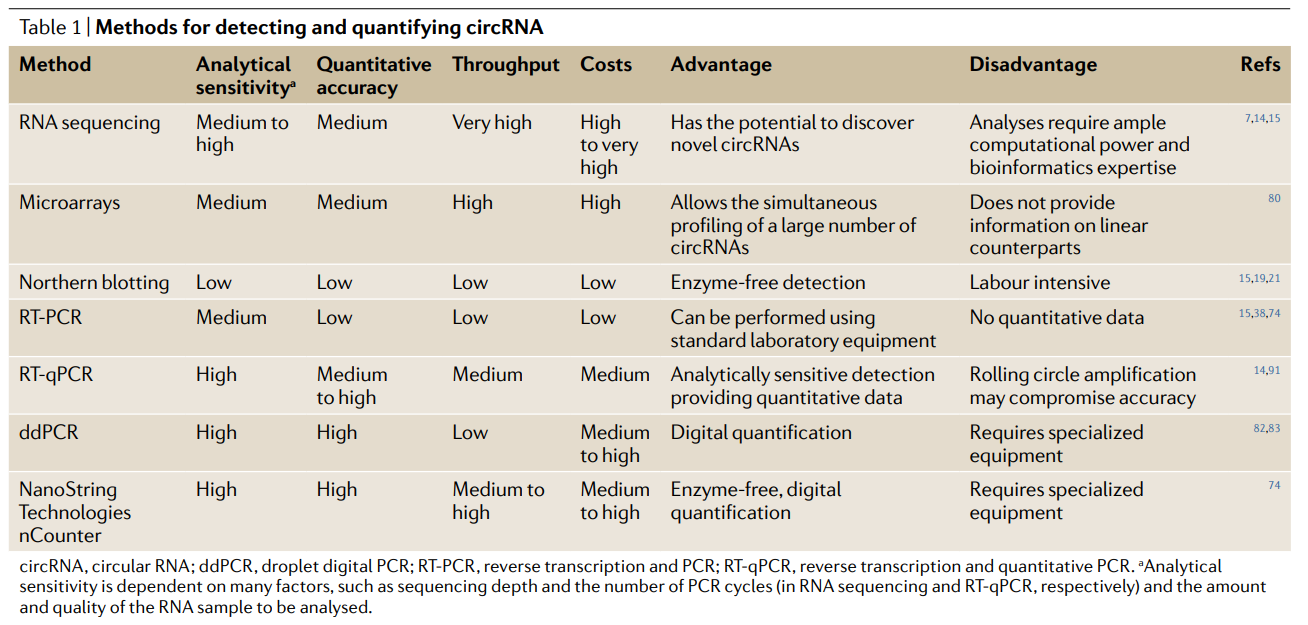
图45 目前检测和定量circRNA的主要技术列表([48])
EMBO Journal:circRNA的过去,现在和将来
2019年7月25日,美国Brandeis大学生物系的Sebastian Kadener等人在EMBO Journal杂志上在线发表了circRNA的综述,回顾了circRNA的研究历程,主要进展和将来的发展趋势 ([49])。
7. circRNA研究总体趋势与未解决的问题汇总分析
2019年circRNA延续了迅猛的发展态势,在发表论文总数目,尤其是大于10分的高质量论文数目出现了巨大的增长,国家自然科学基金的项目数也维持了去年的水平。circRNA虽然已经有了多年的研究,但依然有若干重要的问题没有得到有效解决,概括而言,包括如下问题:
(1)circRNA组织/疾病特异性表达的机制;circRNA的组织/疾病特异性表达特征已经有了一定的认知,但产生这种特异性的机制的认识还非常有限。
(2)泛组织circRNA表达谱分析;目前绝大部分研究工作均聚焦于特定组织或疾病标本中的表达分析,但从一些泛组织/疾病表达谱分析的结果来看,组织特异性的表达特征认识还比较局限,因此这方面的工作依然需要加强,尤其是同步分析不同组织间的表达差异。
(3)敲除技术和基因改造动物模型问题;circRNA的敲除是非常有挑战性的工作,目前也仅有少数几篇文章用到了敲除模型,但从回答circRNA的生理/病理意义的角度而言,敲除模型是非常重要的科学证据,因此未来针对特定circRNA分子的敲除模型可能会越来越多。
(4)circRNA精确的亚细胞定位和活细胞成像观测;生物分子的亚细胞定位是决定其功能和互作分子的一个重要特征。目前对circRNA的亚细胞定位认识非常有限,这方面的研究几乎刚刚起步的状态。
(5)circRNA二级结构分析与动力学研究;结构是决定功能的关键,circRNA的二级结构的研究一直比较有限,2019年陈玲玲教授和杨力教授的Cell文章就是发现了circRNA一种特殊的结构状态,并发现它们与自身免疫效应通路的关系。未来有关circRNA二级/高级结构及分子动力学的研究将是重要的发展方向。
(6)circRNA修饰谱分析;目前circRNA中仅有m6A修饰的文章报道,但RNA中有多达150种的修饰方式,绝大部分的研究依然非常有限,因此有关circRNA的秀食谱将是非常有潜力的研究方向。
(7)同一circRNA分子不同机制的研究;circRNA研究发展到今天,很多的研究都聚焦于同一个circRNA分子,因此关于同一个分子在不同组织或疾病中的功能机制将是circRNA接下来研究的重要方向,相互作用蛋白,转录后修饰以及是否可以翻译都是值得思考的功能机制方向。
参考文献:
1. Li, X., et al., A unified mechanism for intron and exon definition and back-splicing. Nature, 2019. 573(7774): p. 375-380.
2. Rei Yoshimoto, K.R., Thomas Hansen, Jørgen Kjems & Akila Mayeda, A., Biosynthesis of Circular RNA ciRS-7/CDR1as Is Mediated by Mammalian-Wide Interspersed Repeats (MIRs). BioRxiv, 2019.
3. Wu, K., et al., Circular RNA F-circSR derived from SLC34A2-ROS1 fusion gene promotes cell migration in non-small cell lung cancer. Mol Cancer, 2019. 18(1): p. 98.
4. Liang, D., et al., The Output of Protein-Coding Genes Shifts to Circular RNAs When the Pre-mRNA Processing Machinery Is Limiting. Mol Cell, 2017. 68(5): p. 940-954 e3.
5. Vo, J.N., et al., The Landscape of Circular RNA in Cancer. Cell, 2019. 176(4): p. 869-881 e13.
6. Xu Liu, X.W., Jingxin Li, Shanshan Hu, Yuqi Deng, Hao Yin, Xichen Bao, Qiangfeng Cliff Zhang, Geng Wang, Baolong Wang, Qinghua Shi, Ge Shan, The identification of mecciRNAs and their roles in mitochondrial entry of proteins. bioRxiv, 2019.
7. Chen, R.X., et al., N(6)-methyladenosine modification of circNSUN2 facilitates cytoplasmic export and stabilizes HMGA2 to promote colorectal liver metastasis. Nat Commun, 2019. 10(1): p. 4695.
8. Hua Su, L.Z., Na Li, Guowen Wang, Lingfang Wu, Xiuqing Ma, Kejing Ying, Ruifeng Zhang, Transcriptome-wide map of m6A circRNAs identified in hypoxic pulmonary hypertension rat model. BioRxiv, 2019.
9. van Heesch, S., et al., The Translational Landscape of the Human Heart. Cell, 2019. 178(1): p. 242-260 e29.
10. Mahmoudi, E., et al., Depolarization-Associated CircRNA Regulate Neural Gene Expression and in Some Cases May Function as Templates for Translation. Cells, 2019. 9(1).
11. Xia, X., et al., A novel tumor suppressor protein encoded by circular AKT3 RNA inhibits glioblastoma tumorigenicity by competing with active phosphoinositide-dependent Kinase-1. Mol Cancer, 2019. 18(1): p. 131.
12. Zheng, X., et al., A novel protein encoded by a circular RNA circPPP1R12A promotes tumor pathogenesis and metastasis of colon cancer via Hippo-YAP signaling. Mol Cancer, 2019. 18(1): p. 47.
13. Liang, W.C., et al., Translation of the circular RNA circbeta-catenin promotes liver cancer cell growth through activation of the Wnt pathway. Genome Biol, 2019. 20(1): p. 84.
14. Zhao, J., et al., Transforming activity of an oncoprotein-encoding circular RNA from human papillomavirus. Nat Commun, 2019. 10(1): p. 2300.
15. Xiaojuan Fan, Y.Y., Zefeng Wang, Pervasive translation of circular RNAs driven by short IRES-like elements. BioRxiv, 2019.
16. Chen, S., et al., Widespread and Functional RNA Circularization in Localized Prostate Cancer. Cell, 2019. 176(4): p. 831-843 e22.
17. Li, Q., et al., CircACC1 Regulates Assembly and Activation of AMPK Complex under Metabolic Stress. Cell Metab, 2019. 30(1): p. 157-173 e7.
18. Wu, N., et al., Translation of yes-associated protein (YAP) was antagonized by its circular RNA via suppressing the assembly of the translation initiation machinery. Cell Death Differ, 2019. 26(12): p. 2758-2773.
19. Park, O.H., et al., Endoribonucleolytic Cleavage of m(6)A-Containing RNAs by RNase P/MRP Complex. Mol Cell, 2019. 74(3): p. 494-507 e8.
20. Jia, R., et al., Defining an evolutionarily conserved role of GW182 in circular RNA degradation. Cell Discov, 2019. 5: p. 45.
21. Liu, C.X., et al., Structure and Degradation of Circular RNAs Regulate PKR Activation in Innate Immunity. Cell, 2019. 177(4): p. 865-880 e21.
22. Abdollahzadeh, R., et al., Competing endogenous RNA (ceRNA) cross talk and language in ceRNA regulatory networks: A new look at hallmarks of breast cancer. J Cell Physiol, 2019. 234(7): p. 10080-10100.
23. Wesselhoeft, R.A., et al., RNA Circularization Diminishes Immunogenicity and Can Extend Translation Duration In Vivo. Mol Cell, 2019. 74(3): p. 508-520 e4.
24. Chen, Y.G., et al., N6-Methyladenosine Modification Controls Circular RNA Immunity. Mol Cell, 2019. 76(1): p. 96-109 e9.
25. Chen, N., et al., A novel FLI1 exonic circular RNA promotes metastasis in breast cancer by coordinately regulating TET1 and DNMT1. Genome Biol, 2018. 19(1): p. 218.
26. Huang, S., et al., Loss of Super-Enhancer-Regulated circRNA Nfix Induces Cardiac Regeneration After Myocardial Infarction in Adult Mice. Circulation, 2019. 139(25): p. 2857-2876.
27. Wang, L., et al., Circular RNA circRHOT1 promotes hepatocellular carcinoma progression by initiation of NR2F6 expression. Mol Cancer, 2019. 18(1): p. 119.
28. Yang, F., et al., Circ-HuR suppresses HuR expression and gastric cancer progression by inhibiting CNBP transactivation. Mol Cancer, 2019. 18(1): p. 158.
29. Li, H., et al., Therapeutic targeting of circ-CUX1/EWSR1/MAZ axis inhibits glycolysis and neuroblastoma progression. EMBO Mol Med, 2019. 11(12): p. e10835.
30. Sun, Y.M., et al., circMYBL2, a circRNA from MYBL2, regulates FLT3 translation by recruiting PTBP1 to promote FLT3-ITD AML progression. Blood, 2019. 134(18): p. 1533-1546.
31. Li, Q., et al., Circular RNA MAT2B Promotes Glycolysis and Malignancy of Hepatocellular Carcinoma Through the miR-338-3p/PKM2 Axis Under Hypoxic Stress. Hepatology, 2019. 70(4): p. 1298-1316.
32. Wei, Y., et al., A Noncoding Regulatory RNAs Network Driven by Circ-CDYL Acts Specifically in the Early Stages Hepatocellular Carcinoma. Hepatology, 2019.
33. Hu, Z.Q., et al., Circular RNA Sequencing Identifies CircASAP1 as a Key Regulator in Hepatocellular Carcinoma Metastasis. Hepatology, 2019.
34. Ju, H.Q., et al., A circRNA signature predicts postoperative recurrence in stage II/III colon cancer. EMBO Mol Med, 2019. 11(10): p. e10168.
35. Hall, I.F., et al., Circ_Lrp6, a Circular RNA Enriched in Vascular Smooth Muscle Cells, Acts as a Sponge Regulating miRNA-145 Function. Circ Res, 2019. 124(4): p. 498-510.
36. Garikipati, V.N.S., et al., Circular RNA CircFndc3b modulates cardiac repair after myocardial infarction via FUS/VEGF-A axis. Nat Commun, 2019. 10(1): p. 4317.
37. Liu, C., et al., Targeting pericyte-endothelial cell crosstalk by circular RNA-cPWWP2A inhibition aggravates diabetes-induced microvascular dysfunction. Proc Natl Acad Sci U S A, 2019. 116(15): p. 7455-7464.
38. Zhang, S.B., et al., CircAnks1a in the spinal cord regulates hypersensitivity in a rodent model of neuropathic pain. Nat Commun, 2019. 10(1): p. 4119.
39. Zhu, P., et al., IL-13 secreted by ILC2s promotes the self-renewal of intestinal stem cells through circular RNA circPan3. Nat Immunol, 2019. 20(2): p. 183-194.
40. Litke, J.L. and S.R. Jaffrey, Highly efficient expression of circular RNA aptamers in cells using autocatalytic transcripts. Nat Biotechnol, 2019. 37(6): p. 667-675.
41. Xiao, M.S. and J.E. Wilusz, An improved method for circular RNA purification using RNase R that efficiently removes linear RNAs containing G-quadruplexes or structured 3′ ends. Nucleic Acids Res, 2019. 47(16): p. 8755-8769.
42. Feng, J., et al., Genome-wide identification of cancer-specific alternative splicing in circRNA. Mol Cancer, 2019. 18(1): p. 35.
43. Ji, P., et al., Expanded Expression Landscape and Prioritization of Circular RNAs in Mammals. Cell Rep, 2019. 26(12): p. 3444-3460 e5.
44. Humphreys, D.T., et al., Ularcirc: visualization and enhanced analysis of circular RNAs via back and canonical forward splicing. Nucleic Acids Res, 2019. 47(20): p. e123.
45. Ruan, H., et al., Comprehensive characterization of circular RNAs in ~ 1000 human cancer cell lines. Genome Med, 2019. 11(1): p. 55.
46. Teng, X., et al., NPInter v4.0: an integrated database of ncRNA interactions. Nucleic Acids Res, 2019.
47. Yang, Y., et al., Circular RNA profile in liver tissue of EpCAM knockout mice. Int J Mol Med, 2019. 44(3): p. 1063-1077.
48. Kristensen, L.S., et al., The biogenesis, biology and characterization of circular RNAs. Nat Rev Genet, 2019. 20(11): p. 675-691.
49. Patop, I.L., S. Wust, and S. Kadener, Past, present, and future of circRNAs. EMBO J, 2019. 38(16): p. e100836.
.png)