首先祝大家五一节快乐!在刚刚过去的四月份,环状RNA相关的研究报道总体保持稳定。四月份的研究报道中人类疾病和生理活动相关的环状RNA探索研究依然占据了绝大部分,除肿瘤外,也包含了关节系统及神经系统的研究。也有几篇文章涉及到环状RNA分子机制和进化机制的研究。四月份也有两篇文章介绍了环状RNA研究的新技术体系。下面我们一起来回顾一下四月份的环状RNA研究吧:
1. 肿瘤领域环状RNA研究持续活跃
口腔癌中circRNA_100290竞争性结合miR-29家族发挥作用
4月3日,Nature子刊Oncogene在线发表了湘雅二院L Chen为通讯作者的文章,介绍发现环状RNA circRNA_100290通过竞争性结合miR-29家族分子在口腔癌中的作用[1]。

在本中,作者通过芯片法分析了口腔癌和癌旁组织中环状RNA的表达情况,发现了环状RNA circRNA_100290与CDK6均在口腔癌中高表达。干扰circRNA_100290后CDK6表达也下降,且细胞增殖受到抑制。进一步通过荧光素酶报告系统,作者发现了circRNA_100290可以通过竞争性结合miR-29家族的分子发挥作用[1]。
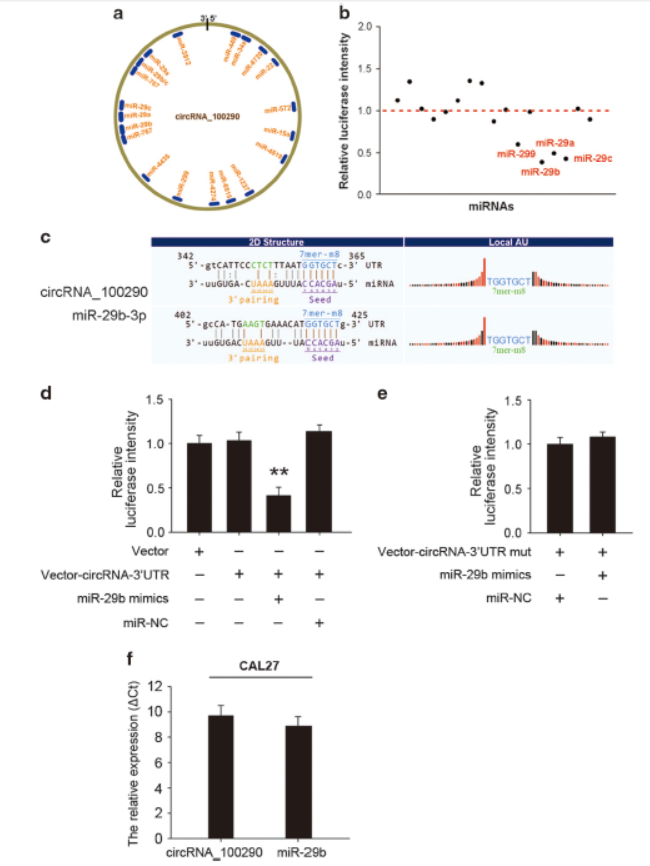
图1 circRNA_100290通过竞争性结合miR-29家族分子在口腔癌中的作用(来自[1])
环状RNA hsa_circ_0004018与肝细胞癌有关
4月6日,Oncotarget杂志在线发表了宁波大学医学院郭俊明教授和Hu Yaoren为共同通讯作者的文章,介绍发现 hsa_circ_0004018与肝细胞癌有关[2]。

文章作者通过芯片法分析发现了环状RNA hsa_circ_0004018的表达水平明显低于对应的癌旁组织,并且hsa_circ_0004018的表达水平与AFP(甲胎蛋白,肝癌的重要标志物)的表达水平存在相关性。ROC曲线(receiver operating characteristic)分析表明hsa_circ_0004018的表达与肝细胞癌分期特征的系数可达到0.848。因此作者认为hsa_circ_0004018可以作为肝细胞癌的标志物[2]。
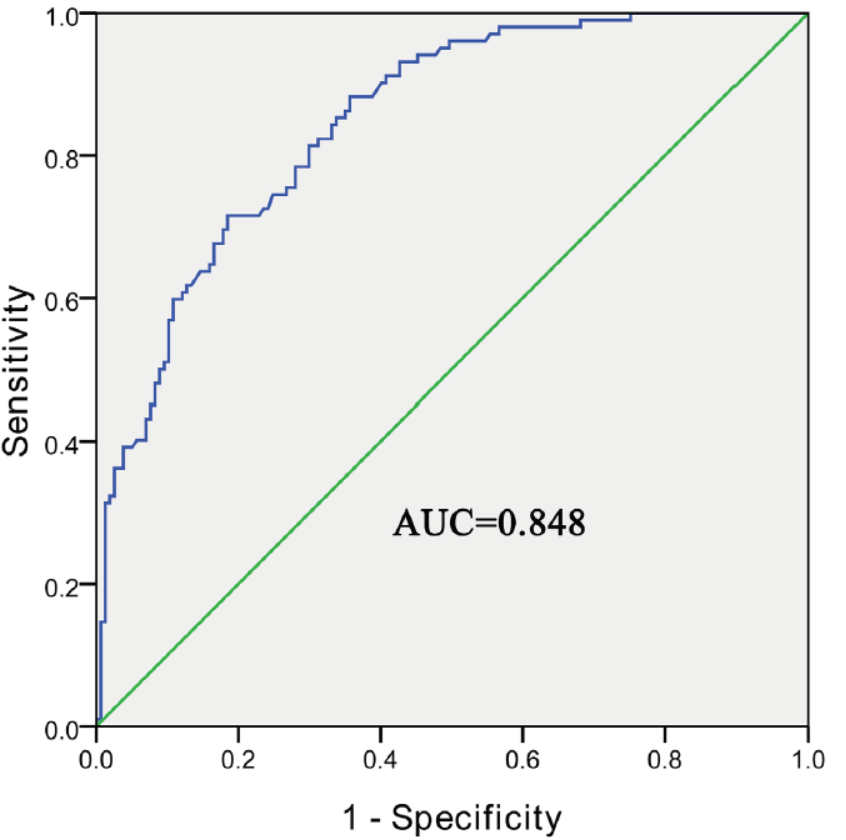
图2 hsa_circ_0004018与肝细胞癌分期有关(来自[2])
环状RNA hsa_circ_0001895在胃癌中下调
近日,Tumor Biology杂志在线发表了宁波大学医学院郭俊明教授和Ye Guoliang为共同通讯作者的文章,介绍发现环状RNA hsa_circ_0001895在胃癌中下调表达[3]。

作者首先分析了hsa_circ_0001895在胃癌细胞系中的表达情况,发现在所分析的细胞系中全部下调,然后又收集了96例临床标本,其中67例中存在下调(67%)。进一步的分析表明hsa_circ_0001895下调的表型与细胞分化,Borrmann型和组织癌胚抗原表达相关,ROC曲线分析表明AUC值可达到0.792[3]。
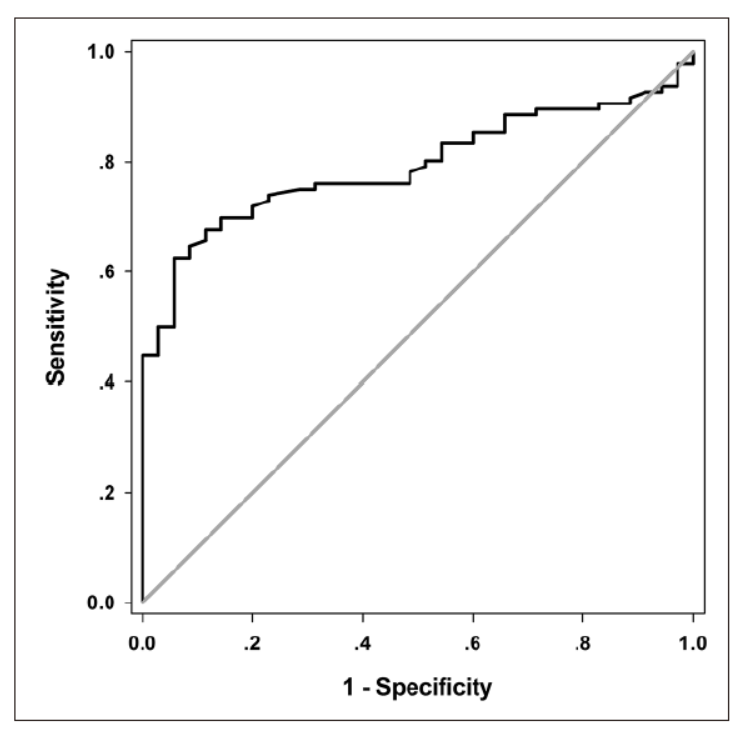
图3 hsa_circ_0001895表达与胃癌存在相关性(来自[3])
沉默 CDR1as可抑制结肠癌细胞增殖
4月7日,OncoTargets and Therapy杂志在线发表了复旦大学附属中山医院Ren Li和Xu Jianmin为共同通讯作者的文章,报道发现沉默CDR1as可抑制结肠癌细胞增殖[4]。

作者首先分析了结肠癌病例中CDR1as表达情况,40例标本中有29例存在上调。在HCT116细胞和DLD-1细胞中进行RNA干扰后发现可明显抑制细胞增殖和迁移率。由于CDR1as可竞争性结合miR-7,作者通过直接过表达miR-7也可得到类似的结果。通过QPCR筛选miR-7可能影响的基因,结果表明EGFR和IGF-1R是受到影响最明显的基因[4]。
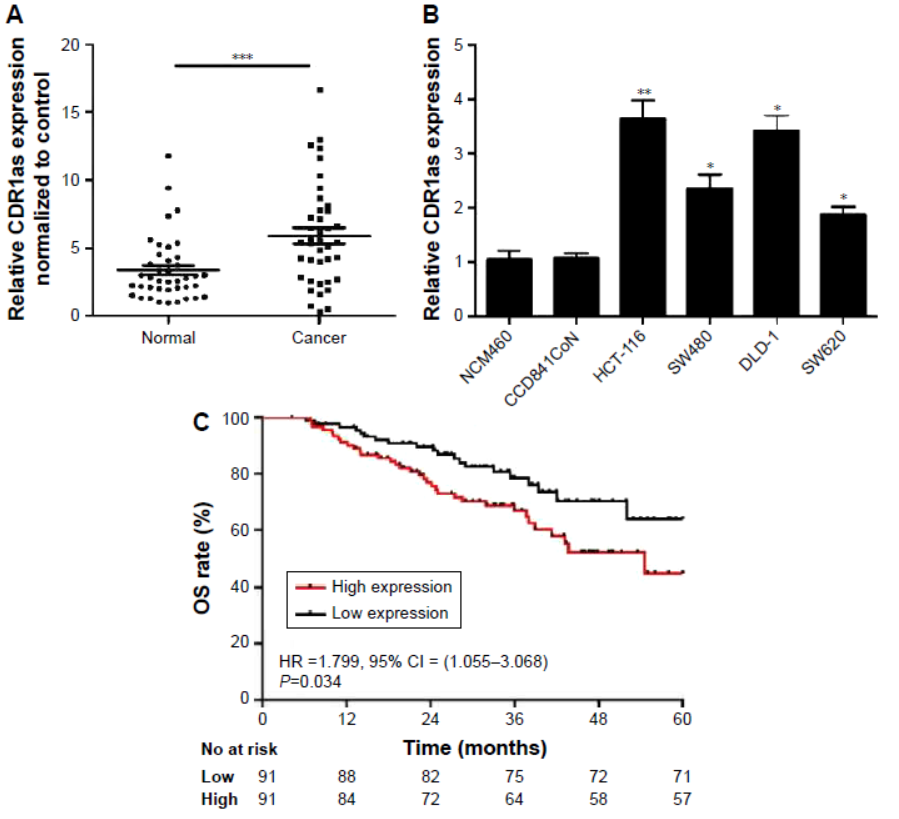
图4沉默 CDR1as可抑制结肠癌细胞增殖(来自[4])
2. 其他疾病或生理活动相关环状RNA研究报道
环状RNA circRNA_010567促进心肌纤维化
4月10日,BBRC杂志在线发表了浙江省人民医院徐鹤云主任为通讯作者的文章,介绍发现环状RNA circRNA_010567促进心肌纤维化的机制[5]。

本中作者利用芯片法分析了糖尿病db/db小鼠模型的心肌中环状RNA,筛选到43种变化的环状RNA,其中24种表达上调。其中circRNA_010567上调最明显。信息学分析后发现circRNA_010567可竞争性结合miR-141,通过荧光素酶报告系统分析后确定了这一预测结果。敲降circRNA_010567后miR-141升高,并可下调TGF-β1通路[5]。
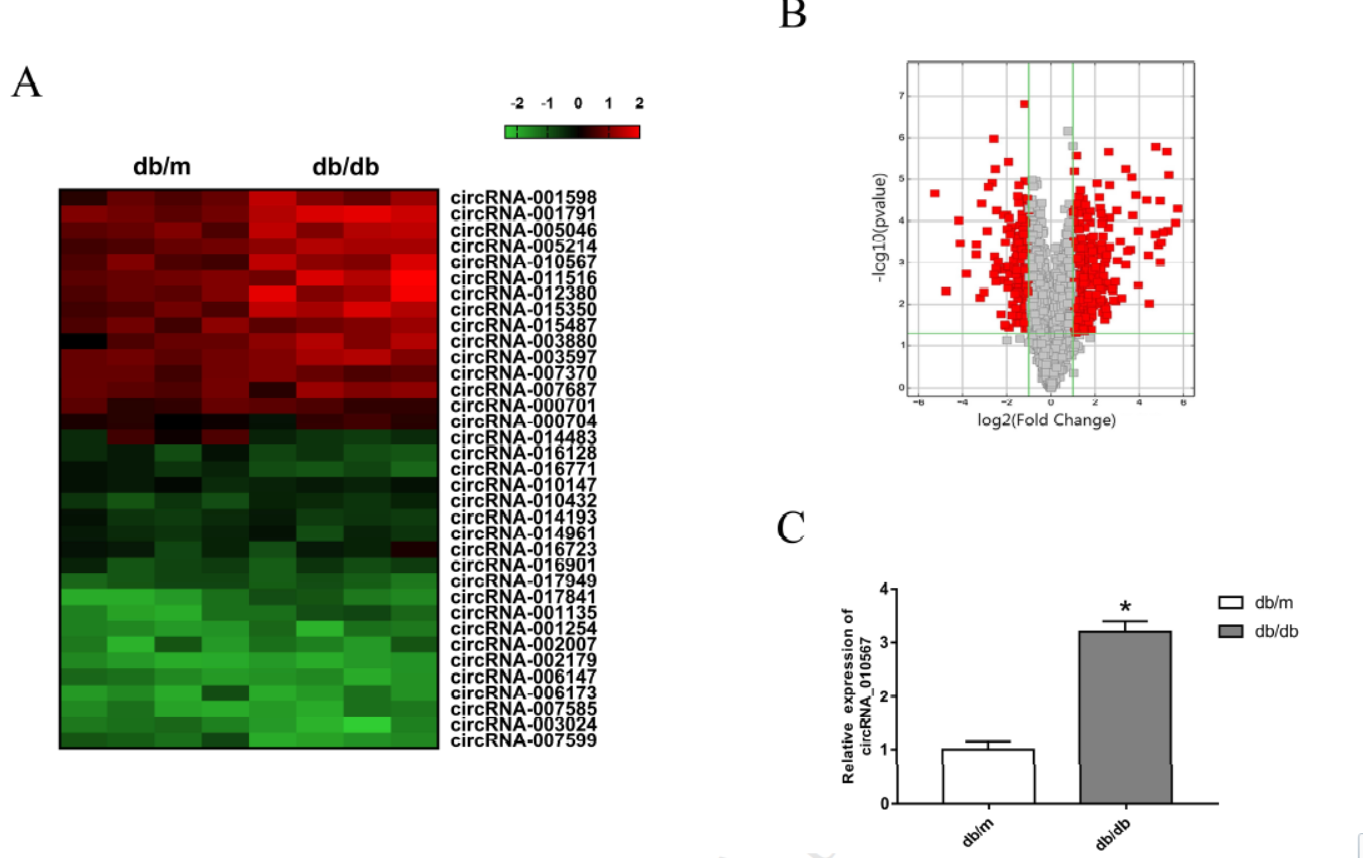
图5环状RNA circRNA_010567促进心肌纤维化(来自[5])
成骨分化中环状RNA变化研究
4月7日,Biomedicine & Pharmacotherapy杂志发表了南方医科大学Xia Hong为通讯作者的文章,介绍分析BMP2诱导MC3T3-E1细胞成骨分化过程中环状RNA变化情况[6]。

作者基于深度测序分析BMP2诱导MC3T3-E1细胞成骨分化过程中变化明显的环状RNA,共找到了158种变化明显的环状RNA,其中74种明显上调。其中circ.19142、circ.5846和circRNA.10042上调最明显。进一步通过竞争性结合miRNA预测和通路分析,作者认为circ.19142和circ.5846可能在成骨分化过程中起重要作用[6]。
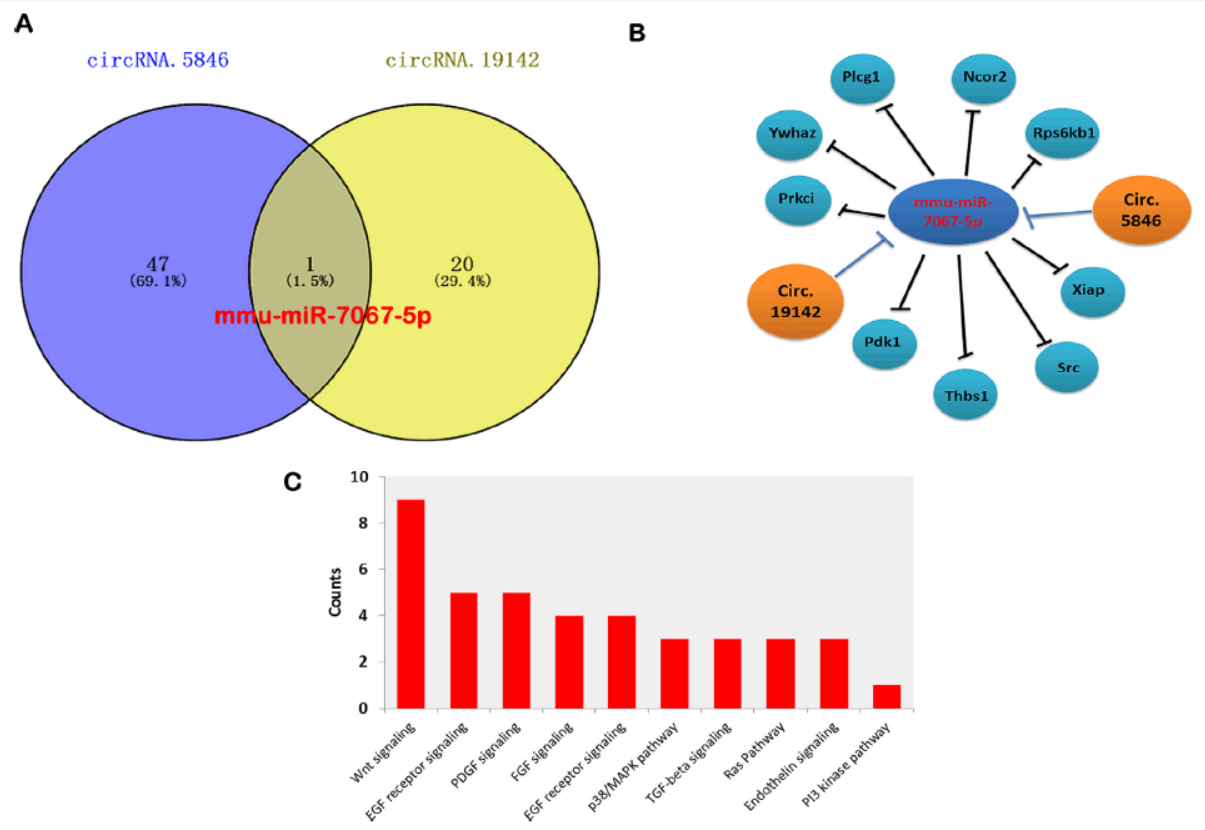
图7 circ.19142和circ.5846与成骨分化有关(来自[6])
人类软骨退变性骨关节炎中环状RNA变化分析
近日,Molecular Therapy Nucleic Acids杂志在线发表了北京大学第三附属医院Ao Yingfang为通讯作者的文章,介绍发现一项关于人类软骨退变性骨关节炎中环状RNA变化的研究工作[7]。

作者通过测序比较分析了正常和关节炎软骨中环状RNA的变化情况,共发现了104种差异的环状RNA,其中44种为过表达。RNA干扰后可促进TNFα表达并促进细胞外基质的再生。本文首次探索了关节炎相关的环状RNA,有助于环状RNA的研究和关节炎疾病机制的研究[7]。
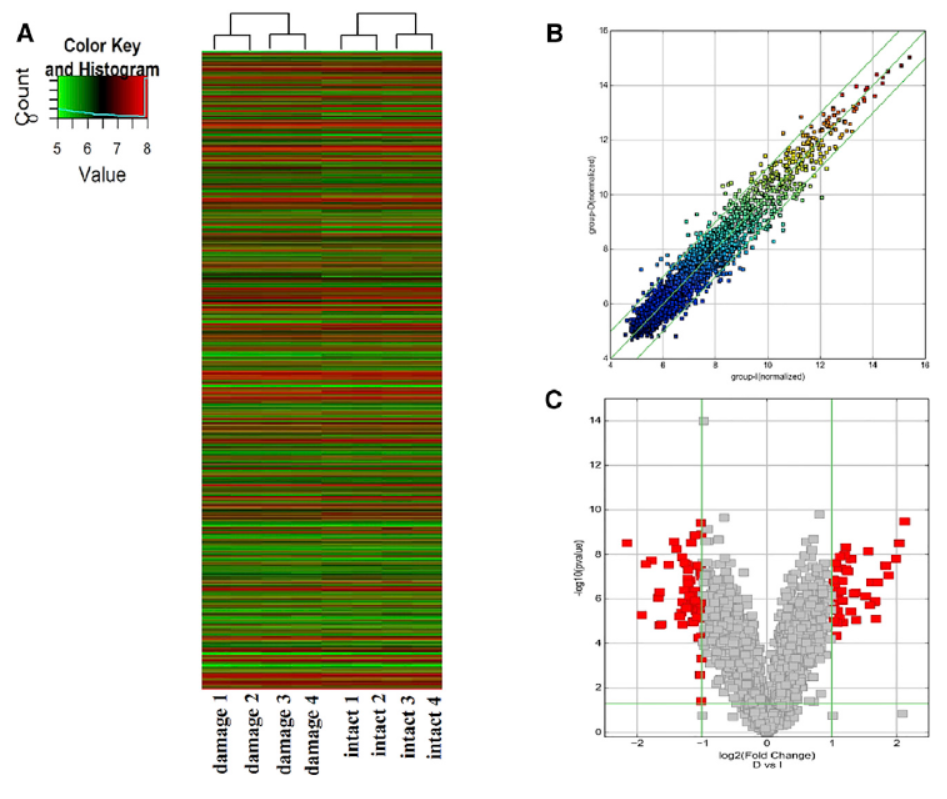
图8 关节炎相关环状RNA(来自[7])
环状RNA与神经损伤后神经性疼痛
4月3日,Frontiers in Molecular Neuroscience杂志发表了广州番禺中心医院的Fan Youling为通讯作者的文章,介绍他们在神经损伤后神经痛中探索非编码RNA的研究工作,其中也发现了一些环状RNA[8]。

本中作者分析了SD大鼠进行避免神经损伤手术(spared nerve injury surgery,SNI)术后14天的L4−5脊髓样品进行深度测序分析,作者找出了其中变化明显的RNA,包括134种lncRNA,12种miRNA,188种环状RNA和1066种mRNA。然后进行了信息学通路分析[8]。
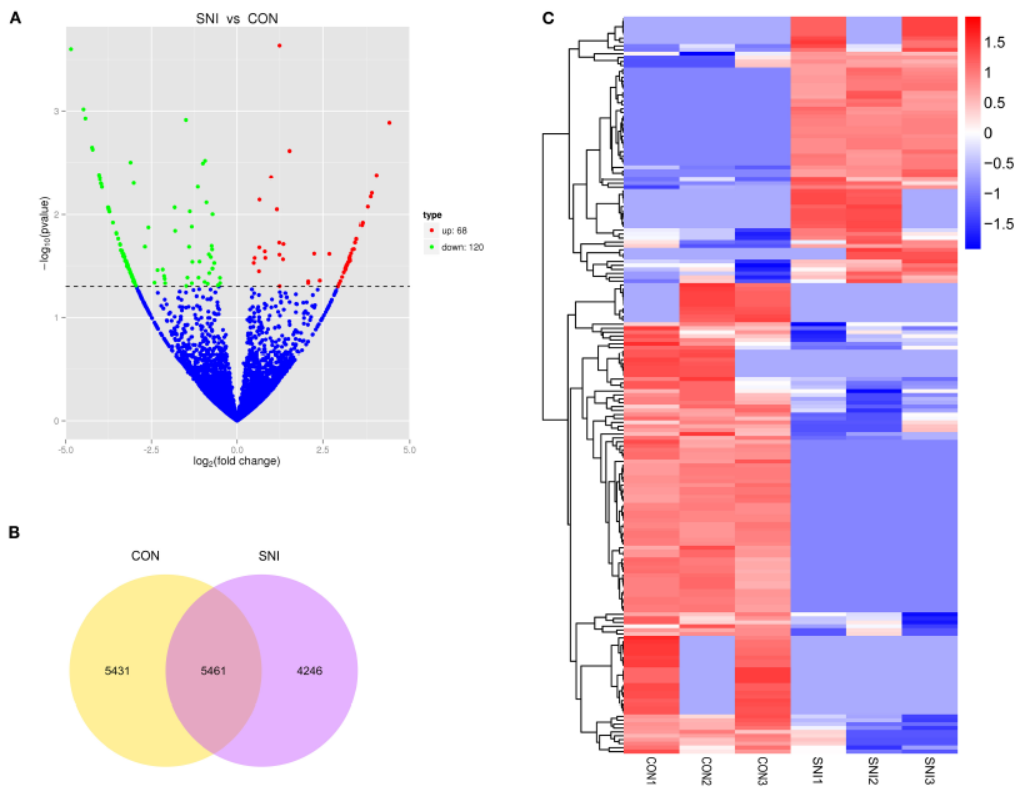
图9 神经损伤后神经性疼痛相关环状RNA(来自[8])
系统性红斑狼疮內源性竞争RNA网络研究价值
4月23日,Expert Opinion on Therapeutic Targets杂志在线发表了安徽医科大学叶冬青教授为通讯作者的综述文章,介绍作者对系统性红斑狼疮疾病中系统分析内源性竞争RNA作用网络的认识,或许有助于增进对系统性红斑狼疮疾病的认识和开发治疗方法[9]。

自噬相关非编码RNA综述
4月25日,知名杂志Autophagy在线发表了哈尔滨医科大学肿瘤医院Xu Shouping和Pang Da为共同通讯作者的综述文章,系统汇总了自噬过程中非编码RNA的作用[10]。

本中系统汇总了与自噬过程有关的非编码RNA研究进展,包括lncRNA,miRNA以及环状RNA。在此基础上基于信息学的通路分析和进化分析,形成了与自噬有关的非编码RNA作用网络[10]。
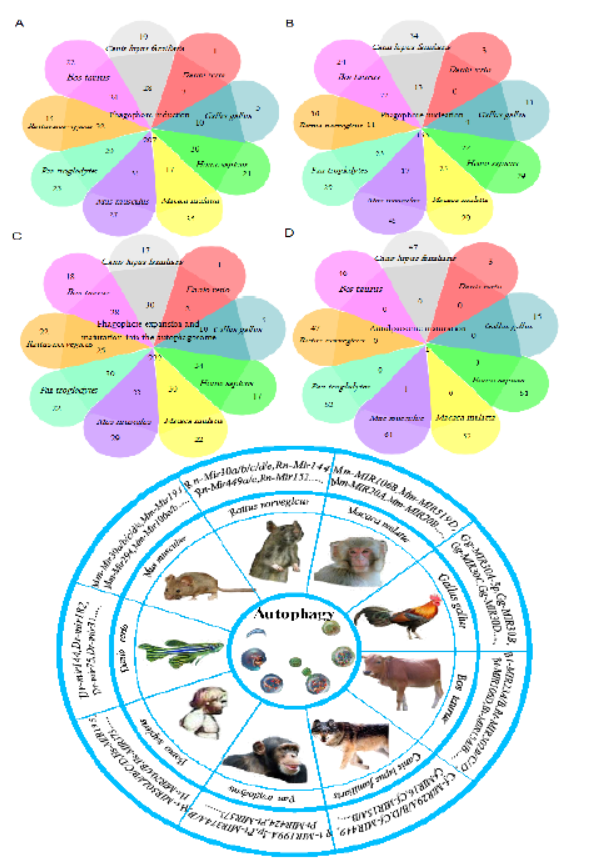
图10 不同物种自噬相关miRNA等非编码RNA作用网络(来自[10])
血液亚组份环状RNA分布研究
近日,Russian Journal of Bioorganic Chemistry杂志发表了俄罗斯科学院化学生物学与基础医学研究所A. V. Savelyeva为通讯作者的文章,介绍一项分析人类血细胞,血浆和血浆亚组份中环状RNA的研究[11]。

作者通过梯度分离血液组分,分别分析血细胞,血浆和血浆亚组份中环状RNA的情况,找到了88个环状RNA。结果表明大部分血浆中的环状RNA存在有膜脂包被的结构中,包括外泌体和microvesicles[11]。
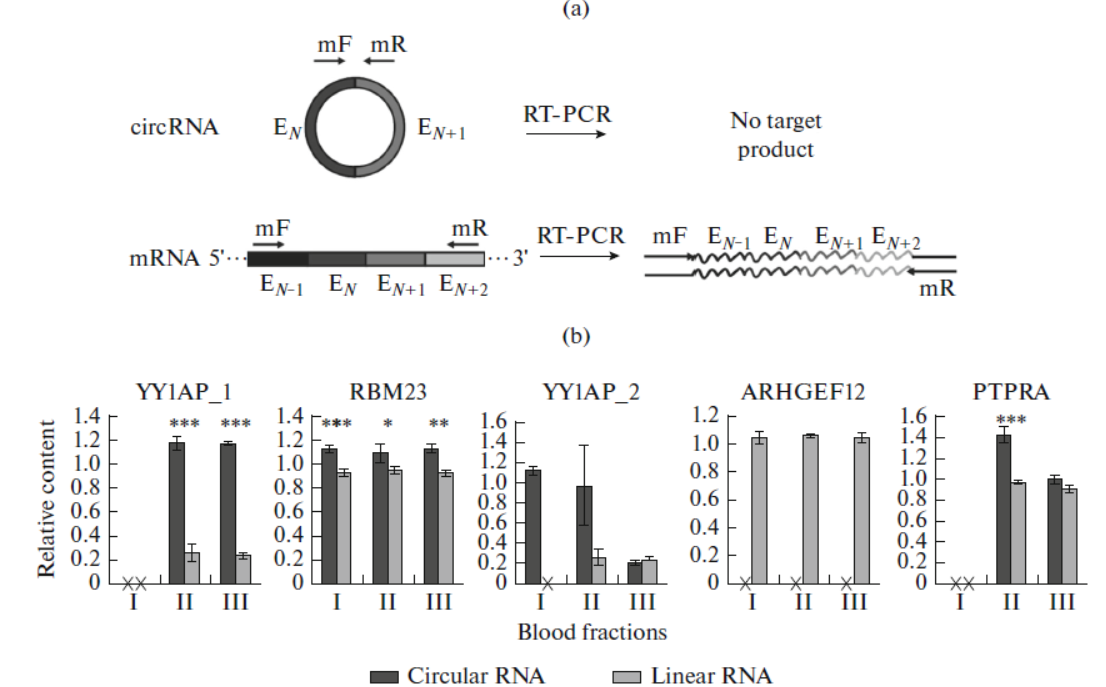
图6 大量环状RNA存在于外泌体和microvesicles中(来自[11])
I, blood cells; II, plasma particles sedimented at 16000 g; III, plasma particles binding the exoRNeasy(Qiagen) adsorbent.
3. 环状RNA机制与进化研究
拟南芥中发现环状RNA调控RNA剪接作用
4月18日,Nature Plants杂志在线发表了法国格勒诺布尔-阿尔卑斯大学Simon J. Conn教授为通讯作者的文章,介绍在拟南芥中发现SEPALLATA3(SEP3)基因来源的环状RNA形成R-loop调控同源mRNA的剪接过程[12]。

本中作者探索了拟南芥中发现SEPALLATA3(SEP3)基因第六外显子形成的环状RNA对所对应的同源mRNA剪接作用的影响,该环状RNA能够稳定结合所对应的DNA序列,形成RNA:DNA杂交体,导致转录暂停,募集RNA拼接因子,形成可变剪切[12]。
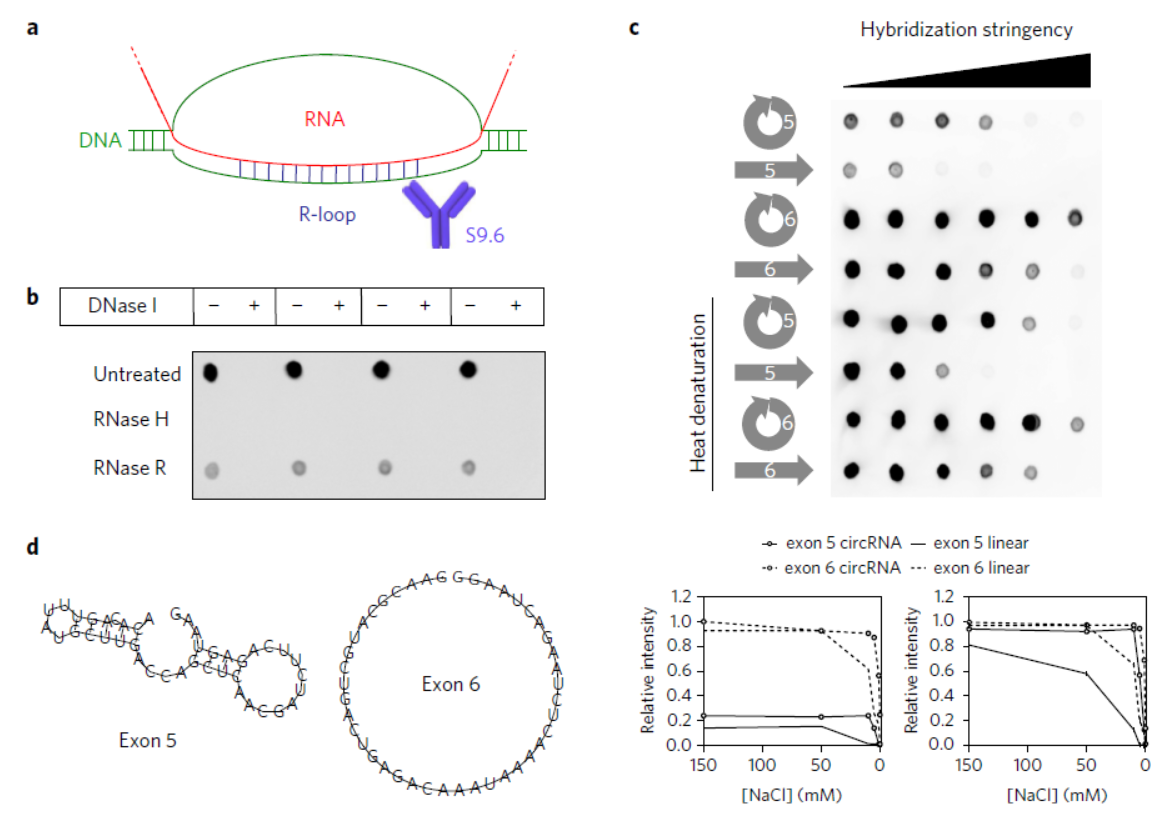
图11 SEP3基因第六外显子形成的环状RNA调控RNA拼接过程(来自[12])
具有锤头状核酶功能的环状RNA
4月28日,RNA Biology杂志在线发表了西班牙IBMCP 的Marcos de la Peña为通讯作者的综述文章,汇总分析了一类具有锤头状核酶功能的环状RNA进化研究的进展[13]。

最近在植物基因组中发现了一类具有自我切割成熟能力的反转座子,称为retrozyme。它们可以被宿主细胞转录,转录产物为非编码的环状RNA。这类环状RNA具有自我切割成熟的能力,与一些类病毒或类病毒样感染因子结构相似。基于这些发现,作者提出假设,认为这些由retrozyme基因编码的circRNA可能是由具有感染性的可自我切割的circRNA核酶进化而来。Retrozyme 基因可能来源于Penelope-like的反转座因子。这些发现为环状RNA在真核生物中的进化研究提供了新思路[13]。
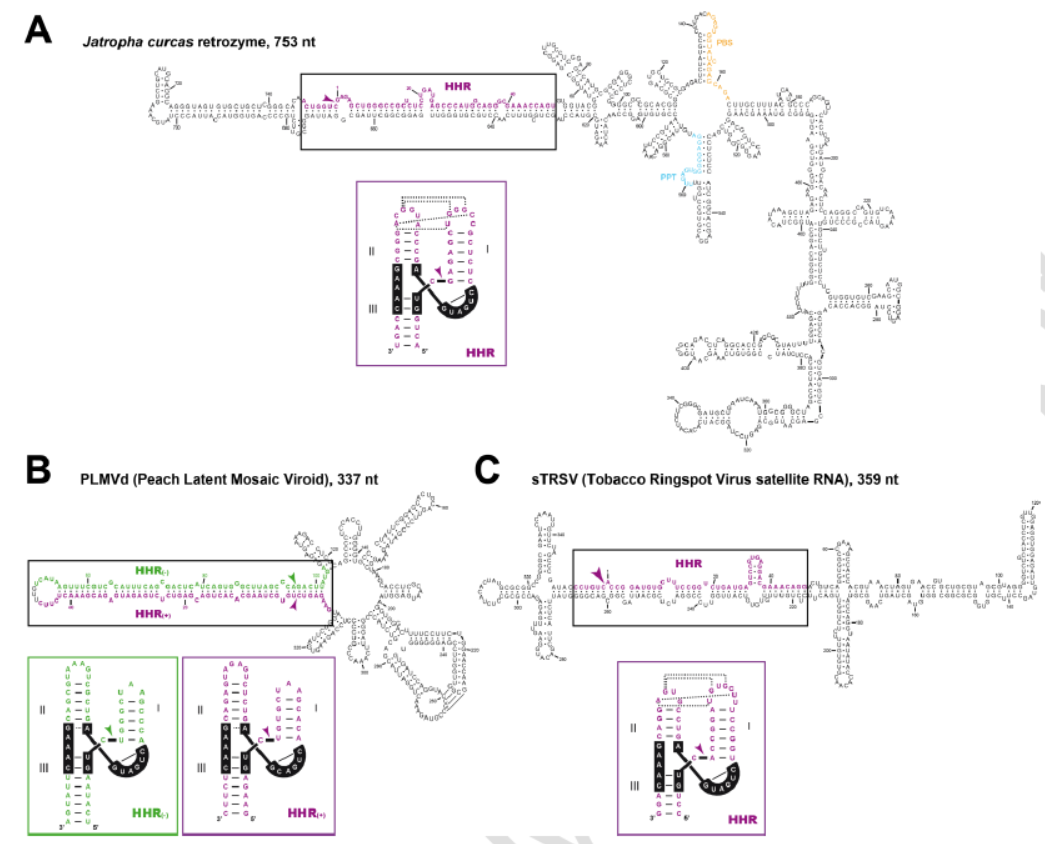
图12 具有锤头状核酶功能的环状RNA(来自[13])
4. 环状RNA研究技术体系进展
环状RNA制备新技术体系
4月11日,Nucleic Acids Research杂志在线发表了NIH 的Amaresh C. Panda和Kotb Abdelmohsen为共同通讯作者的文章,介绍开发了一种提高环状RNA获得效率的新技术[14]。

作者报道的该技术体系在常规的RNase R消化的基础上增加了去除Poly(A)的操作,可大大提高去除线性RNA的效率,作者将该技术称为(RNase R treatment followed by Polyadenylation and poly(A)+ RNA Depletion,RPAD)。基于该技术,作者发现了大量新的内含子来源的环状RNA[14]。
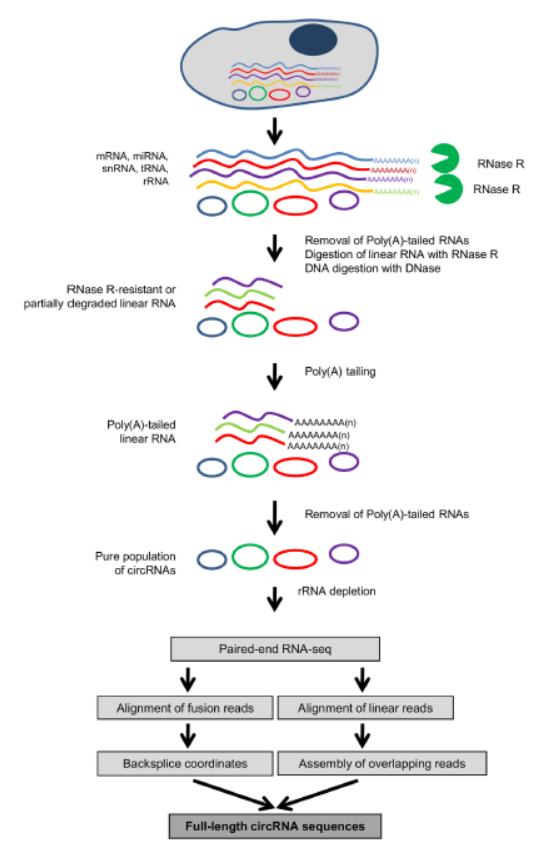
图13 RPAD技术流程(来自[14])
基于tRNA的环状RNA表达体系
4月13日,RNA Biology杂志在线发表了北卡罗莱纳大学教堂山分校A. Gregory Matera教授为通讯作者的综述文章,介绍一种基于tRNA剪接作用的环状RNA表达策略[15]。

去年A. Gregory Matera教授就曾在酶学方法杂志发表过改技术方法的文章。本文主要分析了古生物和动物中tRNA内含子来源的环状RNA,以及借助该体系开发环状RNA表达系统[15]。
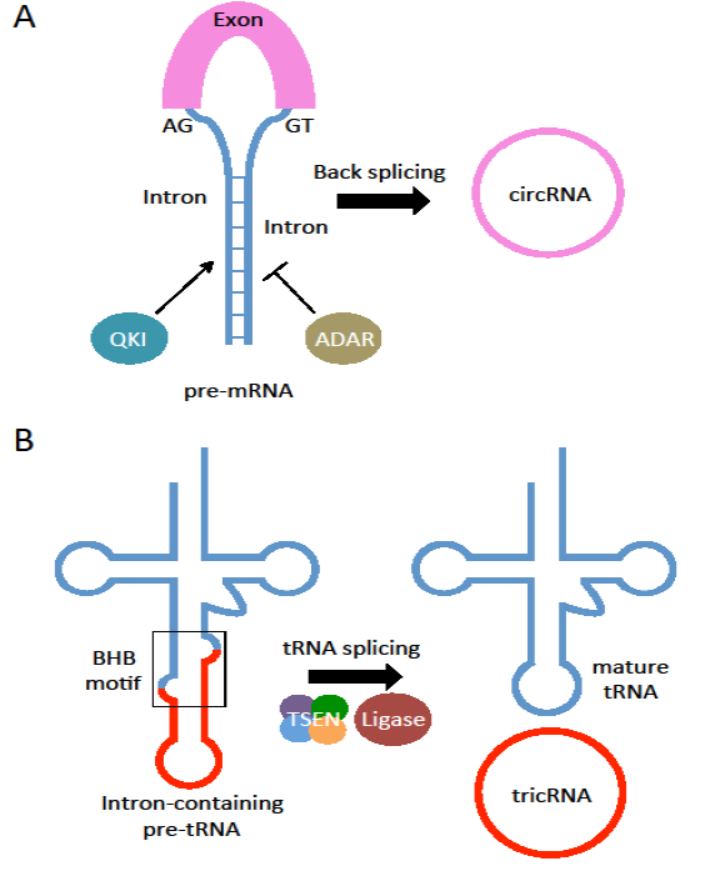
图14 两种不同的体内环状RNA表达系统(来自[15])
参考文献:
1. L Chen, S.Z., J Wu, J Cui, L Zhong, L Zeng and S Ge, circRNA_100290 plays a role in oral cancer by functioning as a sponge of the miR-29 family. Oncogene, 2017: p. 1–11.
2. Liyun Fu, T.Y., Qingqing Chen, Xiaoyan Mo, Yaoren Hu, Junming Guo, Screening differential circular RNA expression profiles reveals hsa_circ_0004018 is associated with hepatocellular carcinoma. Oncotarget, 2017: p. Online.
3. Shao, Y., et al., Decreased expression of hsa_circ_0001895 in human gastric cancer and its clinical significances. Tumour Biol, 2017. 39(4): p. 1010428317699125.
4. Wentao Tang, M.J., Guodong He, Liangliang Yang, Zhengchuan Niu, Mi Jian, Ye Wei, Li Ren, Jianmin Xu, Silencing CDR1as inhibits colorectal cancer progression through regulating microRNA -7. OncoTargets and Therapy, 2017(10): p. 2045–2056.
5. Zhou, B. and H.Y. Xu, A novel identified circular RNA, circRNA_010567, promotes myocardial fibrosis via suppressing miR-141 by targeting TGF-beta1. Biochem Biophys Res Commun, 2017.
6. Qian, D.Y., et al., Differential circRNA expression profiles during the BMP2-induced osteogenic differentiation of MC3T3-E1 cells. Biomed Pharmacother, 2017. 90: p. 492-499.
7. Qiang Liu, X.Z., Xiaoqing Hu, Lan Yuan, Jin Cheng, Yanfang Jiang, and Yingfang Ao, Emerging Roles of circRNA Related to the Mechanical Stress in Human Cartilage Degradation of Osteoarthritis. Molecular Therapy Nucleic Acids, 2017. 7: p. 223-230.
8. Zhou, J., et al., Identification of the Spinal Expression Profile of Non-coding RNAs Involved in Neuropathic Pain Following Spared Nerve Injury by Sequence Analysis. Front Mol Neurosci, 2017. 10: p. 91.
9. Li, L.J., et al., Competitive endogenous RNA network: potential implication for systemic lupus erythematosus. Expert Opin Ther Targets, 2017: p. 1-10.
10. Jian Zhang, P.W., Lin Wan, Shouping Xu & Da Pang, The Emergence of Noncoding RNAs as Heracles in Autophagy Autophagy, 2017.
11. A. V. Savelyeva, D.N.B., E. V. Kuligina, V. V. Morozov, V. A. Richter, and D. V. Semenov, Circular RNAs of Human Blood Cells, Plasma, and Plasma Subfractions. Russian Journal of Bioorganic Chemistry, 2017. 43(2): p. 115–125.
12. Vanessa M. Conn, V.H., Aditya Nayak, Stephanie A. Conos, Giovanna Capovilla, Gökhan Cildir, Agnès Jourdain, Vinay Tergaonkar, Markus Schmid, Chloe Zubieta and Simon J. Conn A circRNA from SEPALLATA3 regulates splicing of its cognate mRNA through R-loop formation. Nature Plants, 2017.
13. Cervera, M.d.l.P.A., Circular RNAs with hammerhead ribozymes encoded in eukaryotic genomes: the enemy at home. RNA Biology, 2017.
14. Panda, A.C., et al., High-purity circular RNA isolation method (RPAD) reveals vast collection of intronic circRNAs. Nucleic Acids Res, 2017.
15. Noto, J.J., C.A. Schmidt, and A.G. Matera, Engineering and expressing circular RNAs via tRNA splicing. RNA Biol, 2017: p. 0.
.png)
