环状RNA的研究进展的如火如荼,越来越多的证据表明它参与了许多关键的信号通路,而它的形成以及调控机制仍迷雾重重。然而,除了作为基因调控网络中的重要一员,circRNA的表达也是基因突变与表型改变中间的桥梁——对circRNA表达变化的理解将进一步揭露复杂性状与疾病的分子分子机制。
北京大学健康科学中心的杨恩策研究员领导的团队在遗传学著名期刊Genome Biology(IF = 13.214)发表了他们的研究成果,为环状RNA的研究提供了一个新的研究视角。

circQTL定义
数量性状位点QTL是理解遗传变异机制的一个有效的工具,例如eQTL通过确定风险SNPs是否与基因表达关联从而判定该基因是否导致疾病。
circQTL指与circRNA表达变化相关联的SNPs。文章对589例人背外侧前额叶皮质样品的circRNA进行了系统地分析,通过circRNA的表达与顺式调控 SNPs (cis-acting SNPs)关联分析,识别到了 196,255 个circQTL。

circQTL 与疾病密切关联
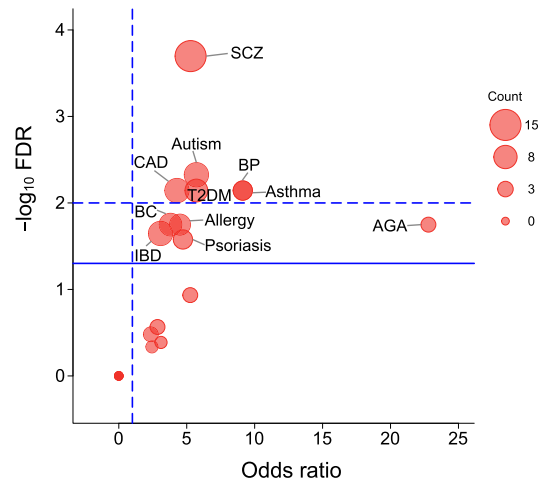
作者将所发现的circQTL SNPs 与GWAS Catlog 疾病与性状 SNP数据进行共定位,发现max-circQTL SNPs(与每个circRNA表达变化最显著关联的SNP)显著富集到了疾病相关的位点。并且与预期的一致,精神分裂症(SCZ)的关联位点最显著;其他比较显著的疾病还包括炎性肠病(IBD)和二型糖尿病(T2DM)。
circQTLs的潜在作用机制
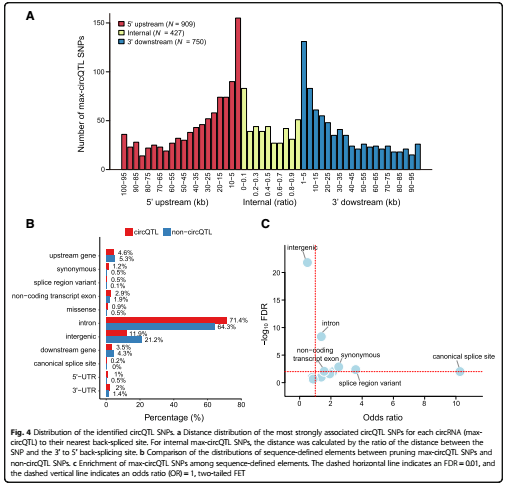
作者试图识别 circQTLs 潜在的机制,发现经典的剪切位点发生变异与更低的circRNA表达相呼应——暗示了经典剪切位点在环化中的必要性;另外还发现internal splicing sites 与external splicing sites (如图4a)有利于环状RNA的表达。同时,circQTLs 还被发现富集到了反向互补序列RCSs(如图4b,c),之前已有研究[Zhang XO et.al., Cell, 2014]表明了该元件在外显子环化中具有重要作用。
然而,虽然作者提到了这么多潜在机制,然而仍有大量circQTLs(~90%)的功能与作用机制仍需进一步探究。
分析circQTL的挑战
其中一个挑战是如何区分SNPs所调控的是circRNA表达(circQTL)还是宿主基因的表达(eQTL)。
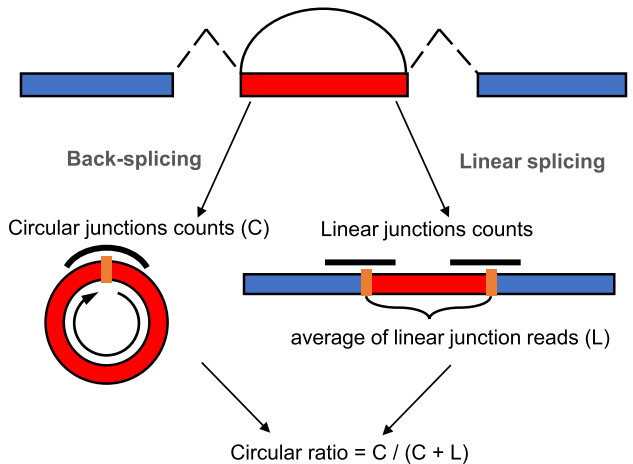
作者整合了两种方法来表示circRNA的表达
- 一种用 circRNA junction的方式
这种方式表示真实的circRNA表达水平,然而却无法区分SNP影响的是circRNA本身还是宿主基因的表达
- 一种用circRNA Ratio
这种通过相对线性RNA的表达克服了第一种方式的缺点
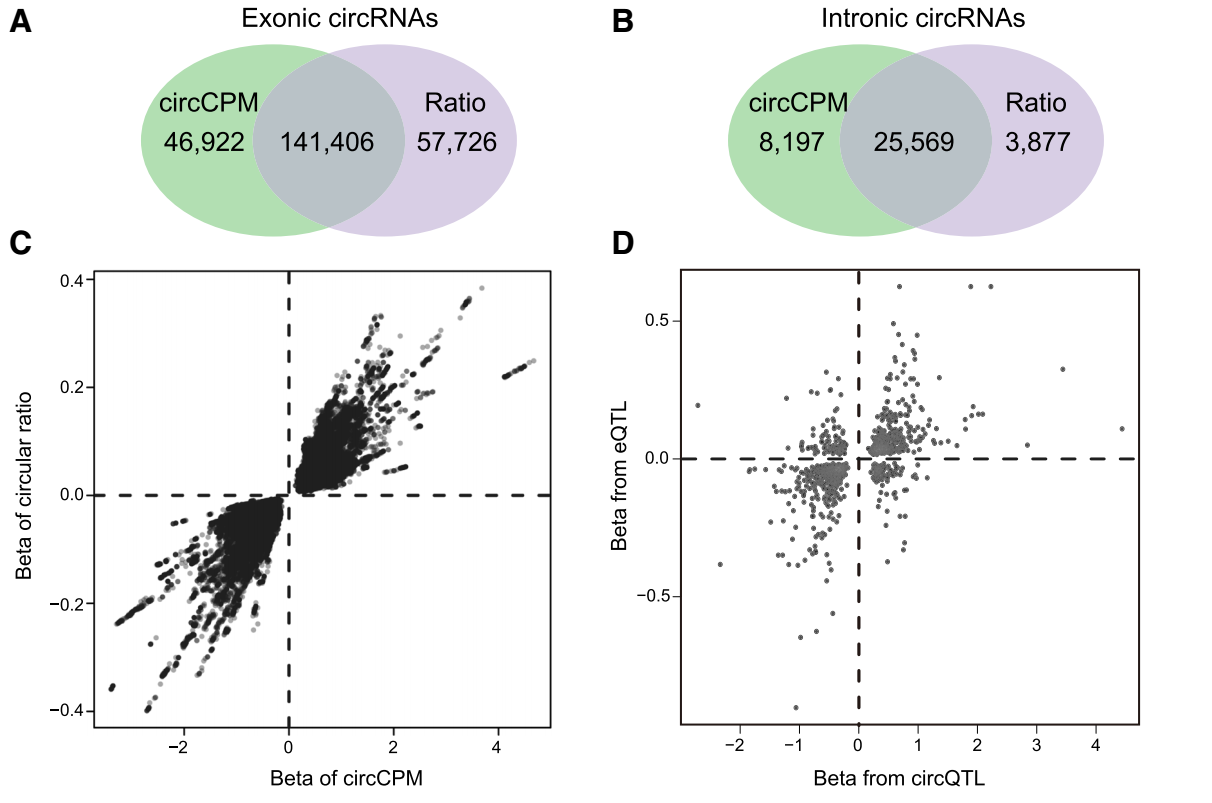
两种方式量化策略虽然不同,然而却达到了一致的检测效率(~70%)
CircRNA变化的影响因素
除了受遗传因素的影响,如circQTL,生物与技术上的因素也会改变circRNA的表达。然而,这种影响相对于线性RNA来说程度更低,这主要得益于环状RNA比线性RNA更稳定。
作者用limma包中的线性模型评估了技术性因素(Institution,文库构建批次LIB,死亡事件PMI,RNA完整性RIN值)以及生物因素(死亡年龄AOD,性别,SCZ)对circRNA表达改变的影响。
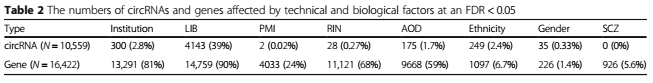
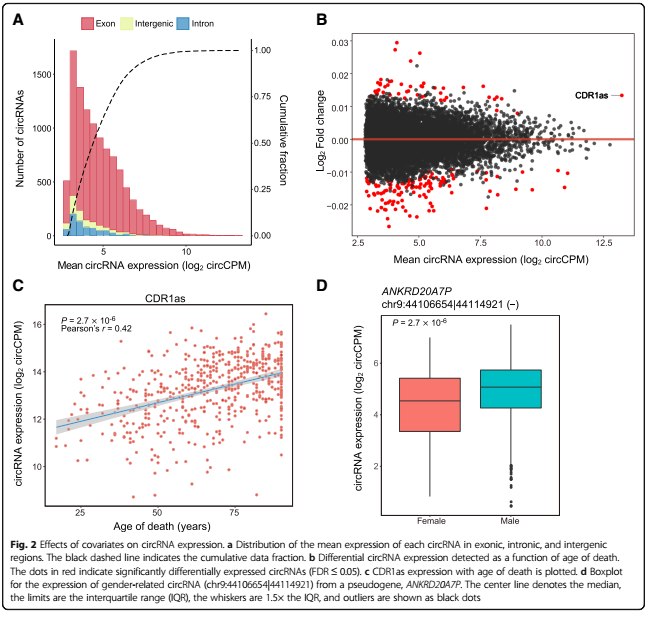
我们可以发现其中library batch即样本的文库大小对circRNA表达变化影响最大,达到了39%,说明它是一个关键的因子;另外发现了175个AOD相关的circRNA,其中有44个表达独立于宿主基因或位于基因间区,这些基因中包括了在脑中高表达的CDR1as——可能暗示了该circRNA在脑中的累积与衰老密切相关。
然而,虽然有一大堆SCZ的GWAS风险位点,但最后结果显示circRNA的表达并不受该因素影响,这种现象在之前一篇研究中[Fromer M et.al., Nat Neurosi, 2016]也被观察到——SCZ GWAS 风险基因并没有差异表达。
结论
总的来说,这篇文章通过数百个背外侧前额叶皮质样品定量了许多circRNA并集中研究了circRNA表达的改变。与线性RNA不同,环状RNA很少受到技术或生物学因素的影响。作者检测到了一些与年龄相关的circRNA并为研究衰老机制提供了有用的信息。最后,通过circQTL的分析,作者将疾病的遗传框架延伸到了环状RNA,表明了环状RNA与疾病风险密切关联;至于circRNA是如何影响病理过程的,仍需要我们去探索。
Zhang XO, Wang HB, Zhang Y, Lu X, Chen LL, Yang L. Complementary
sequence-mediated exon circularization. Cell. 2014;159:134–47.
Fromer M, Roussos P, Sieberts SK, Johnson JS, Kavanagh DH, Perumal TM,
Ruderfer DM, Oh EC, Topol A, Shah HR, et al. Gene expression elucidates
functional impact of polygenic risk for schizophrenia. Nat Neurosci. 2016;19:
1442–53
.png)
