近年来,国内研究环状RNA(circRNA)热潮越来越高,不管是circRNA还是研究circRNA的小伙伴们都如雨后春笋般涌现出来。与传统的线性RNA(linear RNA,含5’和3’末端)不同,circRNA分子呈封闭环状结构,不受RNA外切酶影响,表达更稳定,不易降解。在功能上,近年的研究表明,circRNA分子富含microRNA(miRNA)结合位点,在细胞中起到miRNA海绵( miRNA sponge)的作用,进而解除miRNA对其靶基因的抑制作用。
由于circRNA的结构特殊性,如何采用PCR技术对circRNA进行检测已成为当下一个热门话题。而想要得到好的PCR结果,设计特异的circRNA引物很关键。对于一些刚接触circRNA的小伙伴,设计circRNA引物无从下手,现在给各位小伙伴分享一下circRNA引物设计的过程。
一、circRNA数据库
首先,想要设计引物,就要有序列,要找到序列,必须要用到数据库,下面给各位小伙伴分享几个circRNA的数据库或者网站:
1、circBase,收集和整合已经发布的circRNA数据构建的数据库。
2、circRNABase,整合已发表的circRNA数据,构建miRNA与circRNA以及circRNA与RNA结合蛋白(RBP)的互作网络。
网址:http://starbase.sysu.edu.cn/mirCircRNA.php
3、deepBase,包含了大约15万的circRNA基因(人、鼠、果蝇、线虫等),构建了最全面的circRNA的表达图谱。
网址:http://deepbase.sysu.edu.cn/
4、Circ2Traits,是一个收集与疾病或性状相关的circRNA数据库。
网址:http://gyanxet-beta.com/circdb/
5、circNet,里面收录了一些circ-miRNA-gene网络,以及测序数据。
网址:http://circnet.mbc.nctu.edu.tw/
6、CSCD,癌特异环状RNA数据库
二、引物设计
1、circRNA信息查找
a、以circbase数据库为例,首先访问circbase数据库(http://www.circbase.org/),如下图所示:
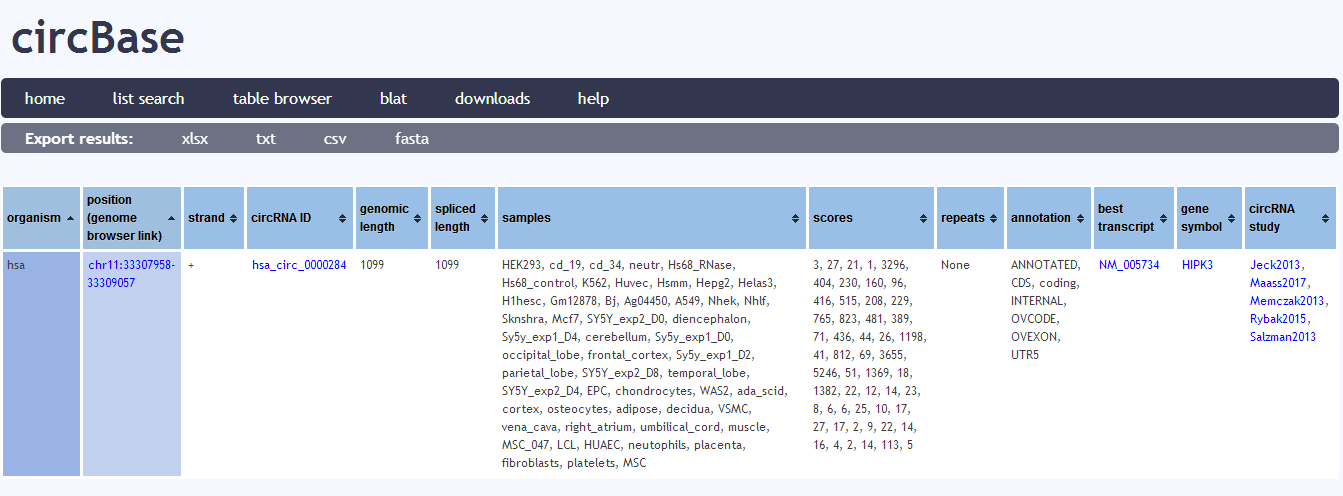
b、在搜索框中输入circRNA ID号,如:hsa_circ_0000284,然后点击Search,搜索结果如下图所示:
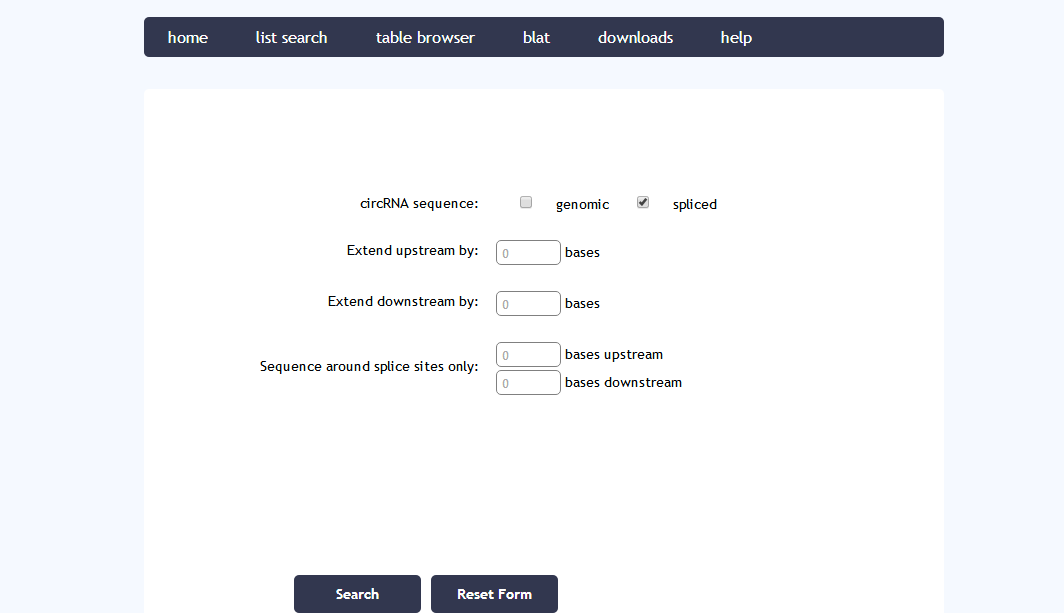
c、点击  ,显示下图界面,然后勾选spliced,点击Search,下载序列。
,显示下图界面,然后勾选spliced,点击Search,下载序列。
2、circRNA引物设计
circRNA的引物设计有以下两种方法:
- 背靠背引物
通常获取到的序列为从剪切位点(backsplice)处打开后的线性序列,因此,在设计引物前应先进行序列位置的变换,截取3´端100-200bp长度的序列置于5´端头部,形成了一个新的序列,新序列的连接处即为剪切位点处。circRNA引物设计的方法按照常规PCR引物设计的原理,跨环化位点设计,上下游引物分别在环化位点的左右两边,即最终的扩增产物必须包含环化位点,背靠背的引物设计原理图如下图所示:
F
R
b、跨环化位点引物
设计跨环化位点的引物同样按照常规PCR引物设计的原理,但是,这种方法要求上游引物(如下图a)或者下游引物(如下图b)要设计在环化位点处。
R
F
图a
R
F
图b
三、确定circRNA引物的特异性
无论是常规PCR还是circRNAPCR,引物的特异性都是很重要的。如何确定引物的特异性?很多小伙伴在设计完引物之后会用软件或者是网站来评估引物的特异性,但是,由于circRNA的结构特殊性,仅仅用软件或者网站来评估是不够的,还需要做qPCR实验来验证引物的特异性。确定circRNA引物的特异性主要有以下3步:
- 溶解曲线
溶解曲线单峰,Tm值在正常范围之内
- 电泳图
电泳条带单一,条带大小正确
- Sanger测序
测序结果单峰,环化位点正确
.png)
