值此新春佳节之际,山人给大家拜年啦!新年新气象,circRNA研究也迎来了重磅级进展:2月7日,Cell杂志同期发表了两篇circRNA研究的论文,迎来了2019年的开门红!
加拿大多伦多大学Paul C. Boutros教授和Housheng Hansen He教授为共同通讯作者的研究工作以Article形式发表在本期的Cell杂志,系统分析了局限性前列腺癌中circRNA表达特征[1]。另一篇文章通讯作者为密歇根大学Alexey I. Nesvizhskii和Arul M. Chinnaiyan1,以Resource形式发表,系统探索了人类肿瘤相关circRNA的表达情况[2]。下面我们分别概括介绍一下这两篇文章的主要内容,主要结论,以飨读者。
首先我们一起学习一下前列腺癌中circRNA表达研究的这篇文章。在这项研究工作中,作者分析了144例有详细临床资料的局限性前列腺癌标本,进行了深度RNA-seq分析,分析了其中的融合基因,线性转录产物和circRNA,统计分析其中与局限性前列腺癌密切相关的分子。基于高通量shRNA文库筛选分析了与细胞增殖有关的circRNA分子,并进一步验证了这些分子在前列腺癌细胞系增殖能力的必要性(essentiality),最后选择circCSNK1G3进行了功能验证[1]。

本项工作共鉴定到76311个circRNA分子,还包括62个融合基因来源的circRNA分子。有趣的是,作者统计发现circRNA和对应linear RNA产物的表达丰度相关性并不高,仅有3%的circRNA表达丰度与对应的linear RNA产物丰度高度相关。绝大部分circRNA的丰度低于对应的linear RNA产物,但有127种circRNA的丰度高于对应linear RNA产物。为找出对前列腺癌有必要功能(essential function)的circRNA分子,作者设计了shRNA文库进行高通量筛选。本文设计的shRNA文库分析了1507种circRNA及它们所对应的1075个host gene线性产物的shRNA,每个RNA分子至少设计2个target位点,文库感染了四种前列腺癌细胞株。shRNA文库筛选结果发现了171种对前列腺癌细胞增殖有必要功能的circRNA分子,但它们所对应的linear RNA分子绝大部分没有必要功能(90%),相关结果也借助其他实验进行了验证分析。为进一步证明这些具有必要功能的circRNA分子的功能,作者挑选了circCSNK1G3进行分析,依据主要包括:circCSNK1G3在四种前列腺癌细胞中均具有essential function,它的线性基因则没有。进一步作者发现circCSNK1G3可竞争性结合miR-181b/d[1]。
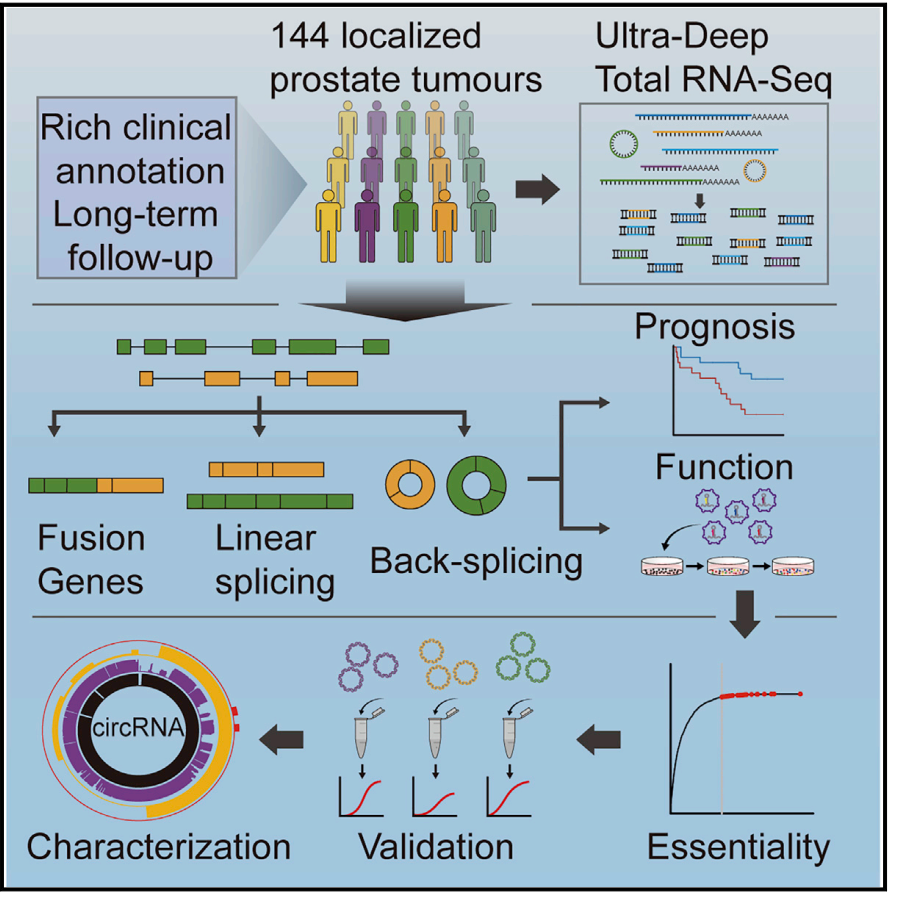
图1 局限性前列腺癌circRNA表达研究概要 (来自[1])
在另一篇文章中,系统分析了人类肿瘤相关circRNA的表达特征。在本文中,作者利用外显子捕获测序体系分析了超过2000例不同组织来源的人类肿瘤标本,分析了其中的circRNA表达谱[2]。 
人类circRNA绝大部分来源于外显子,因此作者尝试了基于已知外显子的探针捕获cDNA后进行深度测序,分析其中的circRNA,结果表明相对于传统的去除rRNA后直接测序或RNase R消化后测序的方法,基于外显子捕获法能得到更多的circRNA信息,且能完整的保留circRNA与对应linear RNA的表达量信息。利用这一体系,作者分析了超过2000例,来源于十几种不同类型肿瘤的标本的circRNA表达情况,汇总形成了MiOncoCirc数据库。该数据库提供了下载链接,包含了circRNA对应的基因座位信息,表达量和基因组结构信息,以及所对应的临床样品信息。有趣的是,本文还发现了一类由于转录通读造成的特殊circRNA分子,称为rt-circRNA (read-through circular RNA),即基因组中上游基因与下游基因的外显子反向拼接形成的circRNA,这与之前报道的融合基因来源的circRNA有所不同,rt-circRNA所对应的基因组并未发生任何序列变化。与上一篇文章的结论类似,本文的作者也发现circRNA的表达量与所对应的linear RNA产物的表达量并不完全对应,并且,在肿瘤中circRNA的表达量普遍低于健康对照组织[2]。
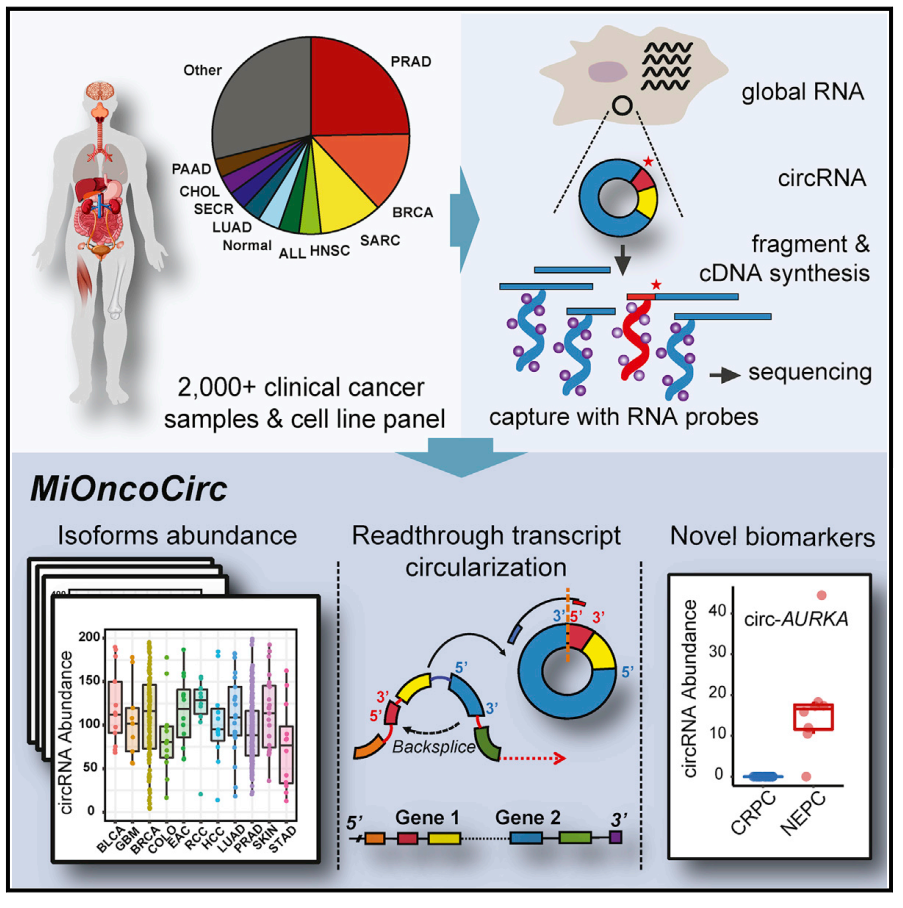
图2 人类肿瘤circRNA表达谱分析 (来自[2])
两篇Cell文章的发表为2019年circRNA研究开了个好头,也暗示circRNA逐渐被国际同行认可,未来必将有更多的国际同行加入该领域。
参考文献:
1. Sujun Chen, V.H., Xin Xu, Julie Livingstone, Fraser Soares, Jouhyun Jeon, Yong Zeng, Junjie Tony Hua, Jessica Petricca, Haiyang Guo, Miranda Wang, Fouad Yousif, Yuzhe Zhang, Nilgun Donmez, Musaddeque Ahmed, Stas Volik, Anna Lapuk, Melvin L.K. Chua, Lawrence E. Heisler, Adrien Foucal, Natalie S. Fox, Michael Fraser, Vinayak Bhandari, Yu-Jia Shiah, Jiansheng Guan, Jixi Li, Miche` le Orain, Vale´ rie Picard, He´ le` ne Hovington, Alain Bergeron, Louis Lacombe, Yves Fradet, Bernard Teˆ tu, Stanley Liu, Felix Feng, Xue Wu, Yang W. Shao, Malgorzata A. Komor, Cenk Sahinalp, Colin Collins, Youri Hoogstrate, Mark de Jong, Remond J.A. Fijneman, Teng Fei, Guido Jenster, Theodorus van der Kwast, Robert G. Bristow, Paul C. Boutros and Housheng Hansen He, Widespread and Functional RNA Circularization in Localized Prostate Cancer. Cell, 2019.
2. Josh N. Vo, M.C., Yajia Zhang, Sudhanshu Shukla, Lanbo Xiao, Yuping Zhang, Yi-Mi Wu, Saravana M. Dhanasekaran, Carl G. Engelke, Xuhong Cao, Dan R. Robinson, Alexey I. Nesvizhskii and Arul M. Chinnaiyan, The Landscape of Circular RNA in Cancer. Cell, 2019.
.png)
