欢迎各位来到“circRNA研究报道汇总”栏目,本期从pubmed中检索收集最新发布的circRNA文献23篇,下面我们一起来看看circRNA研究有哪些新进展。
- 迷人的环:环状RNA研究中的冲突和争议
论文标题:Intriguing circles: Conflicts and controversies in circular RNA research杂志:Wiley Interdiscip Rev RNA. 影响因子:5.844
通讯作者单位:华中科技大学同济医学院
环状RNA(circRNA)是共价闭合的单链环形RNA分子,没有5’帽或3’尾。自2013年具有里程碑意义的ciRS-7 / CDR1作为miR-7海绵发挥作用发现以来,circRNA已成为RNA研究的热门话题。已发现CircRNA在癌症,心血管疾病,神经障碍和许多其他疾病中发挥积极作用。它们可以作为microRNA(miRNA)海绵,蛋白质支架,甚至翻译模板。然而,随着circRNA研究的扩大,出现了许多不同的观点。例如,大多数circRNA能够作为miRNA海绵吗?什么样的circRNA最有可能使miRNA结合?除了作为miRNA海绵体,大多数circRNA的功能是什么?可翻译的circRNA有哪些特征?许多研究声称,circRNA含量丰富,稳定,保守和特异性高,在作为生物标志物方面具有很大的潜力。然而,circRNA丰度是可变的,并非所有的circRNA都是高丰度,它们的稳定性和保存性可能因环境而异。此外,尚不清楚circRNA形成是否更可能受RNA结合蛋白或转录因子的调节。所有这些都是悬而未决的问题,仍有待这些领域的研究人员解答。讨论和研究这些问题将促进对这类新分子的理解,并可能推动启发未来研究的新思路。该综述文章系统全面底探讨上述circRNA相关问题,对深入认识和理解circRNA的功能机制有很好的参考借鉴意义。
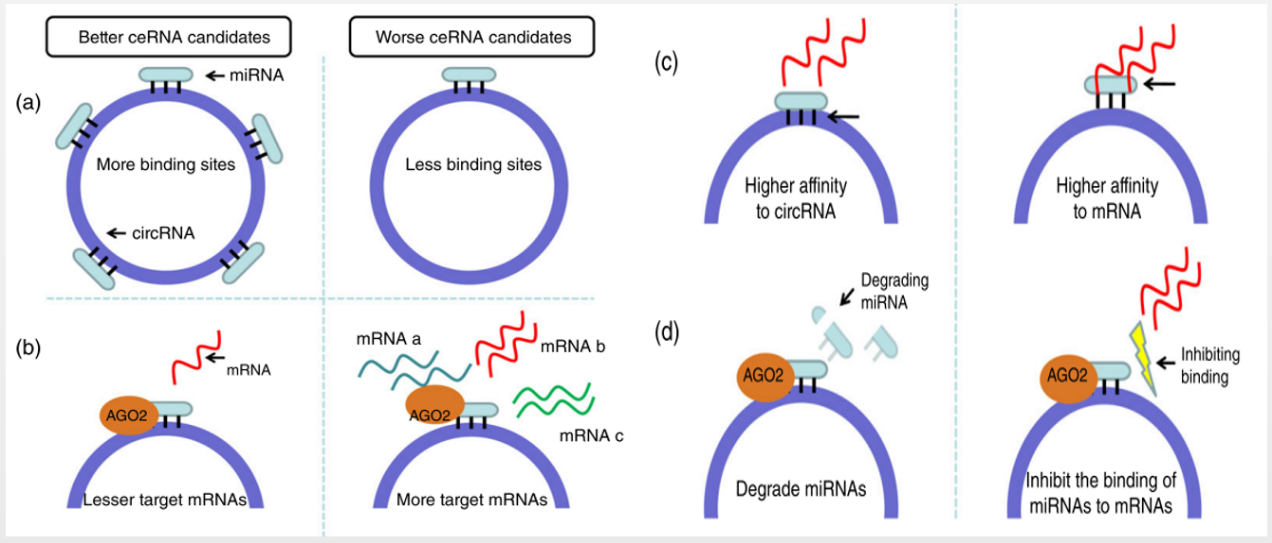
图注:ceRNA形成的先决条件示意图,a:circRNA含有miRNA多个结合位点好过少个结合位点;b:miRNA靶向较少种类mRNA;c:circRNA跟miRNA的亲和力要高于miRNA跟mRNA的亲和力;d:circRNA绑定miRNA后可引起miRNA降解或抑制miRNA-mRNA结合。
- 环状RNA circCHFR通过miR-370/ FOXO1/Cyclin D1途径促进血管平滑肌的增殖和迁移
论文标题:Circular RNA circCHFR Facilitates the Proliferation and Migration of Vascular Smooth Muscle via miR-370/FOXO1/Cyclin D1 Pathway
杂志:Mol Ther Nucleic Acids. 影响因子:5.66
通讯作者单位:西安医学院
动脉粥样硬化(AS)是一种最常见的血管疾病,是脑梗塞,脑卒中和脑梗塞缺血再灌注损伤的主要原因,其特征在于脂质代谢紊乱并且起源于内膜。在AS的发病机制中,伴随着胶原蛋白纤维生成和脂质的积累,血管平滑肌细胞(VSMCs)的增殖是典型的高概率事件。在血管病变期间,来自中膜的VSMC的异常增殖和迁移进入内皮下层的介质引发血管重塑。此外,VSMC暴露于复杂多样的微环境中,导致VSMC的可变性。因此,阐明VSMC参与的未知分子机制AS对于有效治疗至关重要。环状RNA(circRNA)在脑血管疾病的动脉粥样硬化中起重要作用。然而,circRNA调节血管平滑肌增殖和迁移的深层机制仍然难以捉摸。在这项研究中,作者鉴定了一种新的circRNA——circCHFR被证实在ox-LDL诱导的血管平滑肌细胞(VSMC)中异常过表达。功能上,通过转染siRNA沉默circCHFR后,抑制了VSMC的增殖和迁移能力。机制上,生物信息学预测和荧光素酶报告基因测定表明circCHFR充当miR-370的海绵,并且miR-370靶向转录因子FOXO1的3’UTR。此外,转录因子FOXO1可以与CCND1 mRNA的启动子区域结合并促进细胞周期蛋白D1的表达。这一研究表明circCHFR/miR-370/FOXO1/Cyclin D1轴对平滑肌细胞和动脉粥样硬化的重要作用。
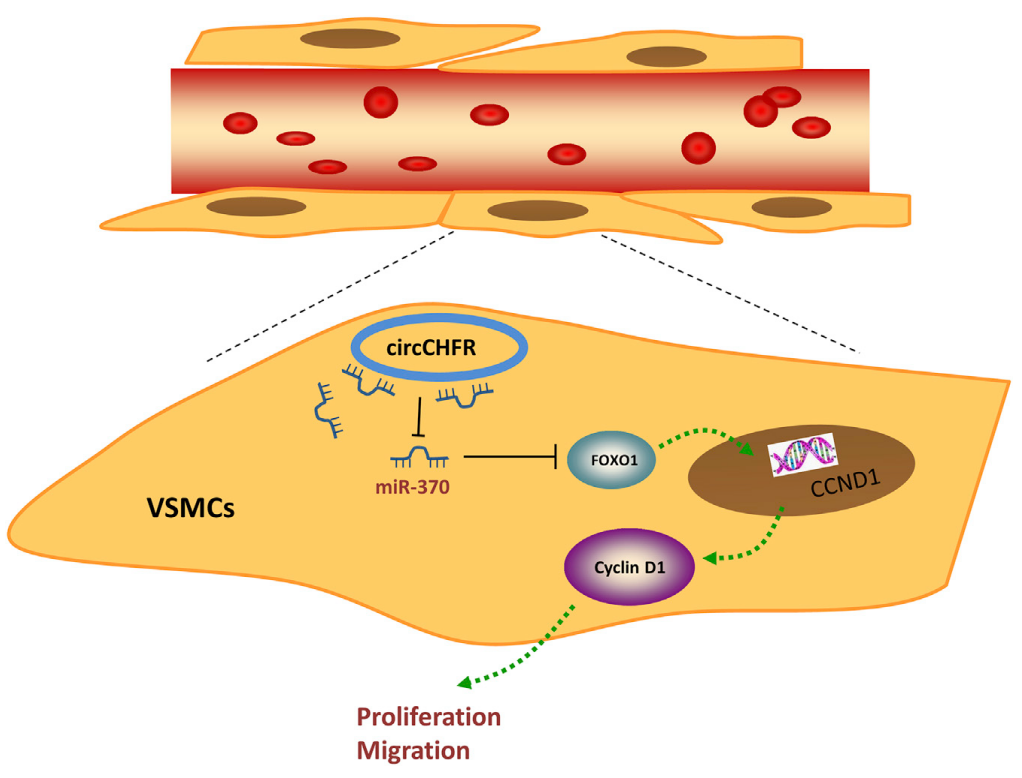
图注:circCHFR通过circCHFR/miR-370/FOXO1/Cyclin D1轴促进平滑肌细胞增殖和迁移
- 环状RNA SNX29海绵吸附miR-744通过激活Wnt5a/Ca2 +信号通路来调节成肌细胞的增殖和分化
论文标题:Circular RNA SNX29 Sponges miR-744 to Regulate Proliferation and Differentiation of Myoblasts by Activating the Wnt5a/Ca2+ Signaling Pathway
杂志:Mol Ther Nucleic Acids. 影响因子:5.66
通讯作者单位:西北农林科技大学
肌生成是一个复杂而精确的协调过程,受到多种非编码RNA和信号通路的高度调节。环状RNA(circRNA)在真核生物的转录后调控中发挥重要作用,但精确的分子作用机制仍未知。该研究中,研究者通过先前的牛骨骼肌的circRNA测序数据筛选到源自SNX29基因的circRNA——circSNX29,并进一步表征其在肌肉发育期间的调节和功能。 circSNX29的过表达促进成肌细胞分化并抑制细胞增殖。使用RNAhybrid预测显示circSNX29可能具有9个miR-744潜在结合位点,并通过荧光素酶筛选实验验证,发现circSNX29直接与miR-744相互作用,miR-744的下调有效地逆转了其对Wnt5a和CaMKIIδ的抑制。进一步,通过KEGG富集分析,Fluo-4、AM、细胞渗透-钙离子荧光探针和WB蛋白质印迹分析,发现Wnt5a和circSNX29的过表达激活了非经典Wnt5a/Ca2+途径。该研究的证据阐明了circSNX29在牛原代成肌细胞中作为miRNA-744的海绵起作用的调节机制,有助于我们理解circRNA-miRNA在肌生成中的作用。
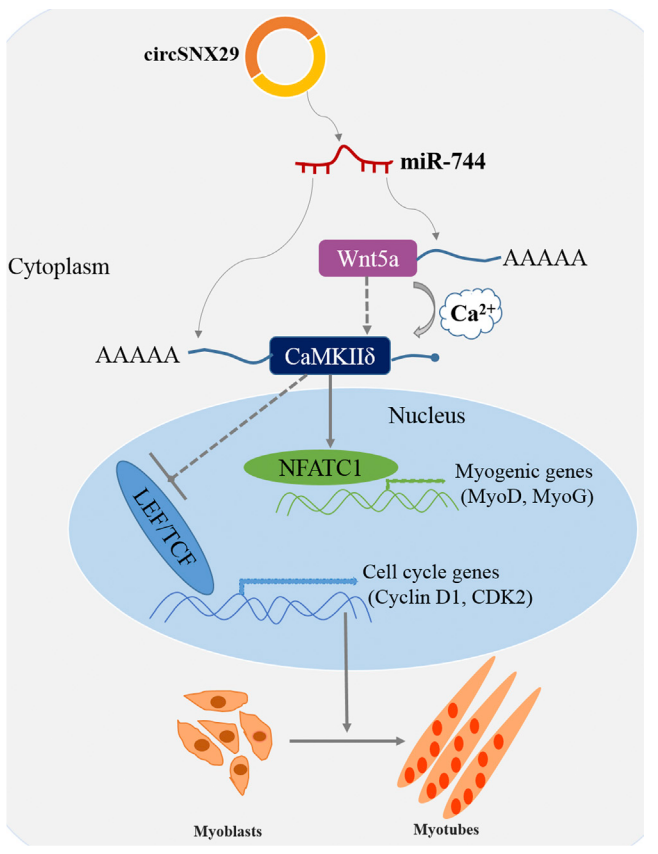
图注:circSNX29作为竞争性海绵吸附miR-744介导成肌细胞分化作用模式图
4、环状RNA形成中的反向剪接编码深度学习
论文标题:Deep Learning of the Back-splicing Code for Circular RNA Formation
杂志:Bioinformatics. 影响因子:5.481
通讯作者单位:克莱姆森大学
环状RNA(circRNA)是广泛存在的一类新型内源RNA。在RNA剪接前期,外显子的5’和3’末端可以通过反向剪接(头对尾剪接)共价连接以形成circRNA。CircRNAs可以跨物种保守,显示组织和发育阶段特异性表达模式,与人类疾病密切相关。尽管有部分研究表明一些序列特征影响反向剪接,但circRNA形成的机制仍然不清楚。
在这项研究中,作者通过应用最先进的机器学习技术,开发了第一个深度学习模型DeepCirCode, DeepCirCode利用以核苷酸序列为输入的卷积神经网络(CNN),以预测人类circRNA形成的反向剪接。并且表现出优于传统机器学习算法的优越性能,例如支持向量机(SVM)和随机森林(RF)。通过DeepCirCode训练学习得到的相关特征序列作为序列基序,其中一些与人类已知RNA剪接,转录或翻译中涉及的基序相匹配。对这些基序的分析表明它们在RNA序列中的分布对于反向剪接是重要的。此外,一些人类基序在小鼠和果蝇中是保守的。这些发现为circRNA形成的反向剪接提供了新的见解。DeepCirCod模型构建的所有数据集和源代码网址 https://github.com/BioDataLearning/DeepCirCode。
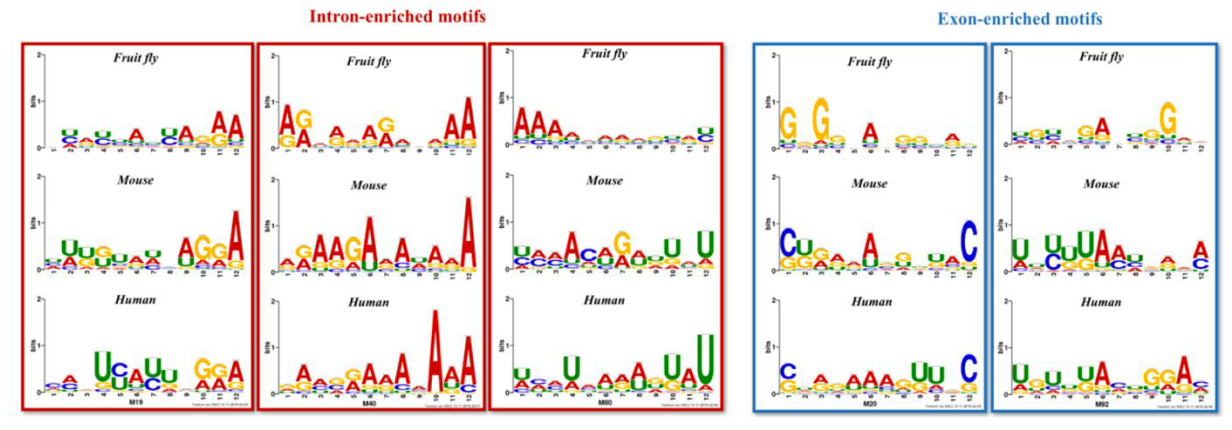
图注:DeepCirCod分析人、小鼠和果蝇中部分circRNA反向剪切位点附近保守的motif基序
参考文献列表
| 序号 | 主要内容 | 论文标题 | 杂志 | IF | 单位 |
| 1 | 迷人的环:环状RNA研究中的冲突和争议 | Intriguing circles: Conflicts and controversies in circular RNA research | Wiley Interdiscip Rev RNA. | 5.844 | 华中科技大学同济医学院 |
| 2 | 环状RNA circCHFR通过miR-370 / FOXO1 / Cyclin D1途径促进血管平滑肌的增殖和迁移 | Circular RNA circCHFR Facilitates the Proliferation and Migration of Vascular Smooth Muscle via miR-370/FOXO1/Cyclin D1 Pathway | Mol Ther Nucleic Acids. | 5.66 | 西安医学院 |
| 3 | 环状RNA SNX29海绵吸附miR-744通过激活Wnt5a/Ca2 +信号通路来调节成肌细胞的增殖和分化 | Circular RNA SNX29 Sponges miR-744 to Regulate Proliferation and Differentiation of Myoblasts by Activating the Wnt5a/Ca2+ Signaling Pathway | Mol Ther Nucleic Acids. | 5.66 | 西北农林科技大学 |
| 4 | 环状RNA形成中的反向剪接编码深度学习 | Deep Learning of the Back-splicing Code for Circular RNA Formation | Bioinformatics. | 5.481 | 克莱姆森大学 |
| 5 | 非编码RNA在肺癌肿瘤发生中的作用 | The Function of Non-Coding RNAs in Lung Cancer Tumorigenesis | Cancers (Basel). | 5.326 | 尤柳·哈蒂耶加努医学和药学大学 |
| 6 | 环状RNA:系统性红斑狼疮疾病的新型生物标志物? | Circular RNAS: novel biomarkers of disease activity in systemic lupus erythematosus? | Clin Sci (Lond). | 5.22 | 西班牙瓦伦西亚临床医学院 |
| 7 | CropCircDB:针对作物应对非生物胁迫的环状RNA资源库 | CropCircDB: a comprehensive circular RNA resource for crops in response to abiotic stress | Database (Oxford). | 3.978 | 南京农业大学 |
| 8 | ALK阳性间变性大细胞淋巴瘤中的非编码RNA网络 | Non-Coding RNA Networks in ALK-Positive Anaplastic-Large Cell Lymphoma | Int J Mol Sci. | 3.687 | 柏林夏里特医学院 |
| 9 | 增加上游染色质长程相互作用可能有利于LysoPC激活的人主动脉内皮细胞中环状RNA的诱导 | Increasing Upstream Chromatin Long-Range Interactions May Favor Induction of Circular RNAs in LysoPC-Activated Human Aortic Endothelial Cells | Front Physiol. | 3.394 | 费城坦普尔大学 |
| 10 | Hsa_circ_0005379通过EGFR途径调节口腔鳞状细胞癌的恶性行为 | Hsa_circ_0005379 regulates malignant behavior of oral squamous cell carcinoma through the EGFR pathway | BMC Cancer. | 3.288 | 北京大学深圳医院 |
| 11 | 在心血管疾病中长的非编码RNA /环状RNA-miRNA-mRNA轴研究综述 | Long noncoding RNA/circular noncoding RNA-miRNA-mRNA axes in cardiovascular diseases | Life Sci. | 3.234 | 吉林大学第二医院 |
| 12 | 分析山羊子宫内膜从着床前阶段到着床阶段发展过程中的circRNA表达谱 | Analyses of circRNA profiling during the development from pre-receptive to receptive phases in the goat endometrium. | J Anim Sci Biotechnol. | 3.205 | 西北农林科技大学 |
| 13 | 环状RNA hsa_circ_0075828通过激活CREB1促进膀胱癌细胞增殖 | Circular RNA hsa_circ_0075828 promotes bladder cancer cell proliferation through activation of CREB1 | BMB Rep. | 3.085 | 北京大学深圳医院 |
| 14 | CircRNAwrap-用于circRNA鉴定,转录物预测和丰度估计的分析流程 | CircRNAwrap-a flexible pipeline for circRNA identification, transcript prediction, and abundance estimation | FEBS Lett. | 2.999 | 中国科学院计算技术研究所 |
| 15 | 过表达circ_0005198海绵吸附miR-1294调节神经胶质瘤细胞增殖,凋亡,迁移和侵袭 | Overexpression of circ_0005198 sponges miR-1294 to regulate cell proliferation, apoptosis, migration, and invasion in glioma | J Cell Biochem. | 2.959 | 齐齐哈尔医科大学附属第二医院 |
| 16 | 乳腺癌中环状RNA circ_0103552预测预后不良,通过海绵吸附miR-1236促进乳腺癌细胞增殖和侵袭 | Circular RNA circ_0103552 forecasts dismal prognosis and promotes breast cancer cell proliferation and invasion by sponging miR-1236 | J Cell Biochem. | 2.959 | 齐齐哈尔医科大学附属第三医院 |
| 17 | 失调的circRNA_100876通过靶向microRNA-136抑制骨肉瘤癌细胞的增殖 | Dysregulated circRNA_100876 suppresses proliferation of osteosarcoma cancer cells by targeting microRNA-136 | J Cell Biochem. | 2.959 | 南方医科大学附属第三医院 |
| 18 | 环状RNA rno_circ_0004002通过靶向miR-342-5p和Wnt3a在肛门直肠畸形中调节细胞增殖,凋亡和上皮 – 间质转化 | Circular RNA rno_circ_0004002 regulates cell proliferation, apoptosis, and epithelial-mesenchymal transition through targeting miR-342-5p and Wnt3a in anorectal malformations | J Cell Biochem. | 2.959 | 中国医科大学附属盛京医院 |
| 19 | 鉴定环状RNA相关竞争内源RNA网络在腭裂发展中作用研究 | Identification of circular RNA-associated competing endogenous RNA network in the development of cleft palate | J Cell Biochem. | 2.959 | 汕头大学医学院附属第二医院 |
| 20 | 骨肉瘤中circRNA的差异表达和生物信息学分析 | Differential expression and bioinformatics analysis of circRNA in osteosarcoma | Biosci Rep. | 2.899 | 广西医科大学附属第一医院 |
| 21 | 电离辐射HEK 293T细胞环状RNA表达谱分析 | Analysis of Circular RNA Expression Profile in HEK 293T Cells Exposed to Ionizing Radiation | Dose Response. | 2.435 | 中国医学科学院北京协和医学院 |
| 22 | 人椎间盘退变中差异表达环状RNA及生物信息学分析 | Profiling and bioinformatics analysis of differentially expressed circular RNAs in human intervertebral disc degeneration | Acta Biochim Biophys Sin (Shanghai). | 2.224 | 第二军医大学长征医院 |
| 23 | SpliceV:线性和环状RNA剪接,表达和调控的分析工具 | SpliceV: analysis and publication quality printing of linear and circular RNA splicing, expression and regulation | BMC Bioinformatics. | 2.213 | 新奥尔良杜兰大学医学院 |
.png)
