8月26日,Genome Medicine杂志在线发表了德州大学休斯顿健康科学中心韩冷教授为通讯作者的文章,报道基于在线数据分析了近1000种肿瘤细胞系中circRNA的表达谱[1]。
总体情况
作者分析所涉及的在线数据主要由以下几个地方下载:
(1)NCI’s Genomic Data Commons (https://portal.gdc.cancer.gov/legacy-archive)
(2)CCLE gene expression data(https://ocg.cancer.gov/programs/ctd2/data-portal)
(3)mutation data from CCLE data portal (https://portals.broadinstitute.org/ccle)
(4)MD Anderson Cell Lines Project (http://tcpaportal.org/mclp)
除了这些细胞系表达与突变相关信息,作者还分析了药物基因组学相关的信息:
(5)Genomics of Drug Sensitivity in Cancer ( http://www.cancerrxgene.org/)
(6)Cancer Therapeutics Response Portal ( https://portals.broadinstitute.org/ctrp/)
(7)CCLE (https://portals.broadinstitute.org/ccle)
作者总共收集到935种肿瘤细胞系的数据,利用多种circRNA分析工具分析整理这些细胞系中circRNA的数据,最终获得了92589种circRNA分子,并根据这些细胞系的来源组织,分析了组织特异性circRNA的表达情况。
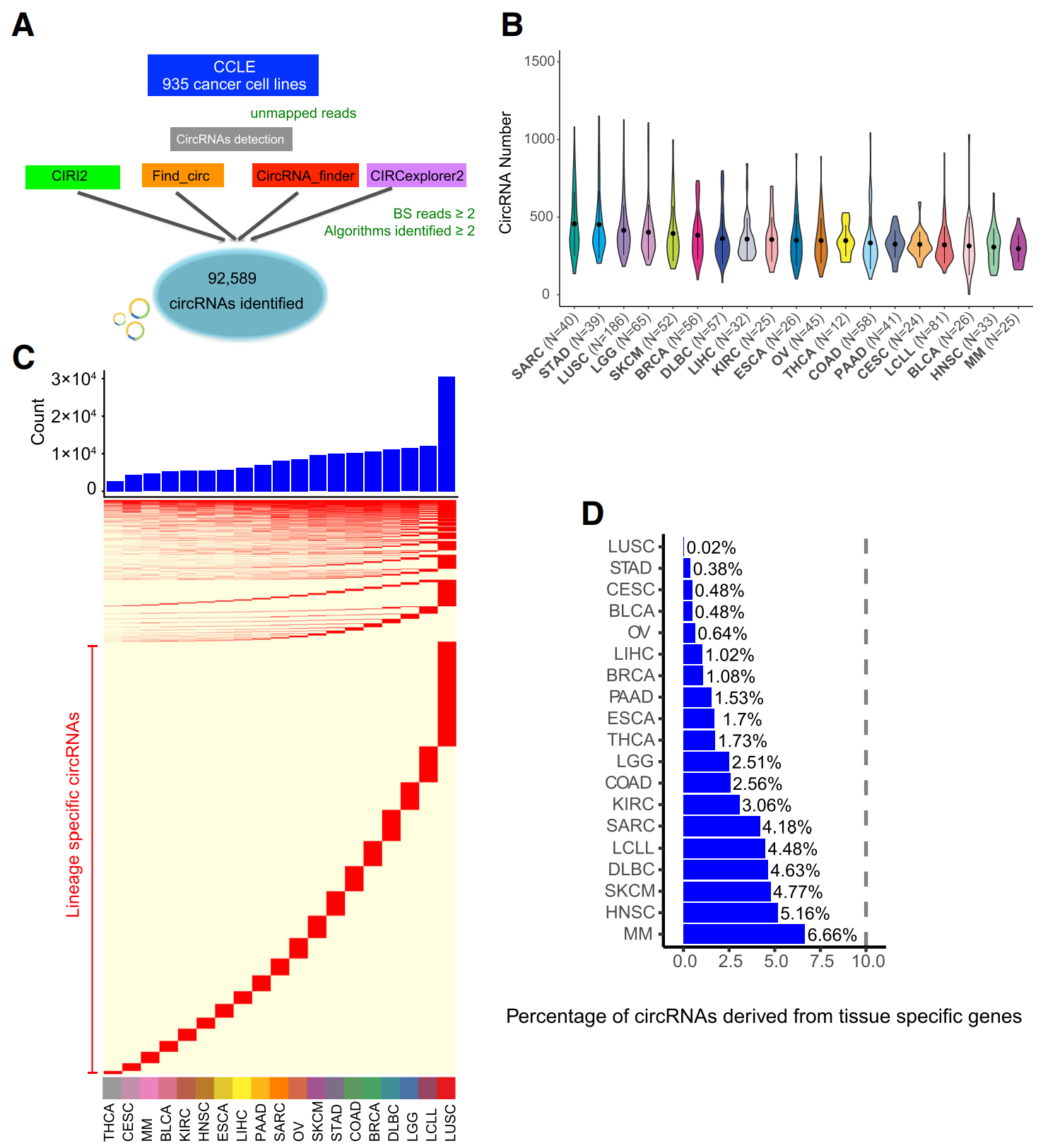
图1肿瘤细胞系circRNA表达谱 ([1])
调控circRNA生成的因素分析
基于上述数据,作者首先通过表达相关性统计分析了可能参与调控circRNA生成的各种因子,包括51种RNA剪切相关的基因,22种3’加工相关的基因,71种RNA解螺旋酶和104种RNA结合蛋白。分析这些基因的表达与circRNA表达的相关性。结果表明,9个基因与反向剪切比例呈现高度正关性,作者也分析了显著负相关的基因,与此前的一项研究报道一致。除了单个基因的相关性,作者还分析了与circRNA生成有关的通路。GSEA分析表明有16种信号通路与circRNA的生成是高度相关的,比如EMT通路,TGFβ通路等等。其中TGFβ通路此前没有报道与circRNA的形成有直接相关性,本文发现该通路在携带较多circRNA的细胞谱系(lineage)中有较强的富集。
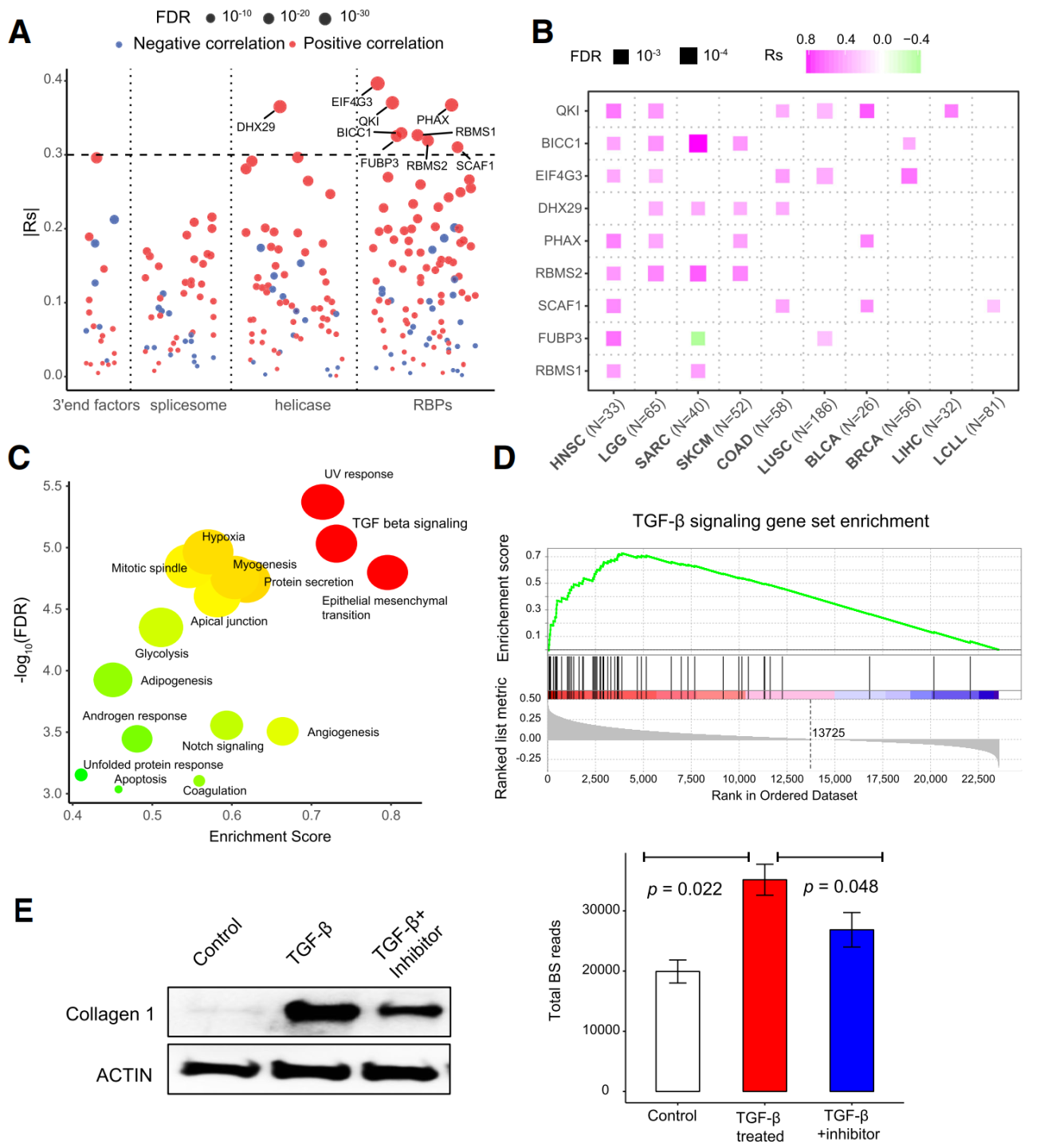
图2 调控circRNA生成的基因和通路分析 ([1])
肿瘤治疗靶点基因相关circRNA在不同细胞系中表达特征分析
已获得FDA批准可以作为肿瘤治疗靶点的基因(Clinically actionable genes)在肿瘤研究中具有特殊重要的意义,作者分析发现其中59个基因的circRNA可以在两种以上细胞系中同时存在,于是作者分析了这些基因来源的circRNA在不同肿瘤中出现的频率,并根据细胞分化谱系进行了划分。Clinically actionable genes生成circRNA的比例偏高,且其中也有更多的RBP结合位点。
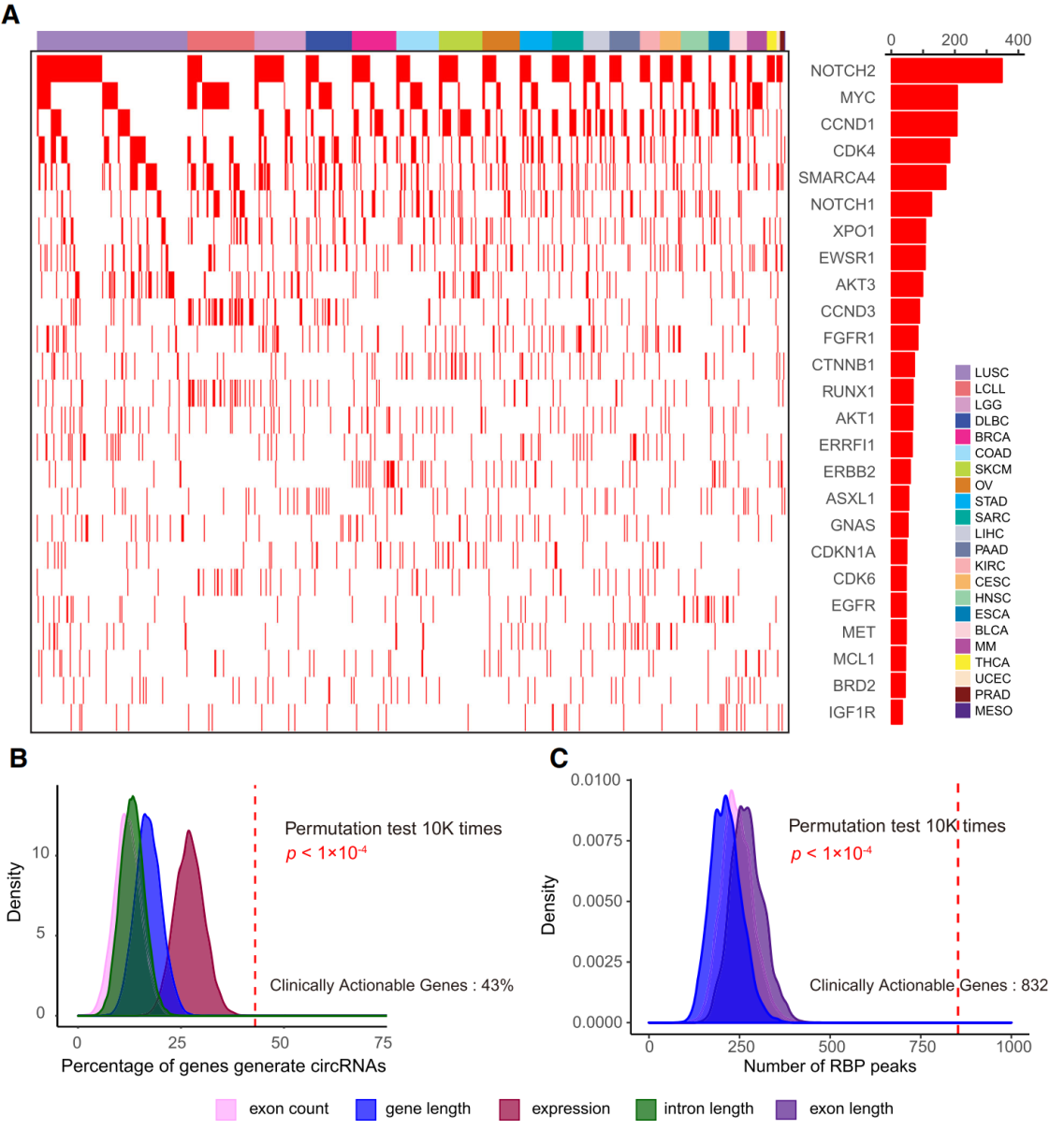
图3 clinically actionable gene来源circRNA分析 ([1])
Myc来源的circRNA(circMyc)与药物作用的关系
从上述clinically actionable gene来源circRNA的分析中,作者发现Myc来源的circRNA (circMyc)在多达208种细胞系中均有表达。circMyc高表达的细胞系中Myc的mRNA和蛋白的表达水平均偏高。MDA-MB-231细胞中过表达circMyc可以促进Myc mRNA的表达增高。过表达circMyc也可以促进细胞增殖。药物敏感性相关性分析表明circMyc的表达与多种药物的敏感性有关。比如HDAC抑制剂Belinostat,circMyc高表达的细胞系对Belinostat的敏感性显著降低。另一种HDAC抑制剂Vorinostat也有类似的线性。这说明circMyc的表达状态与HDAC抑制剂等药物的效果有一定的相关性。
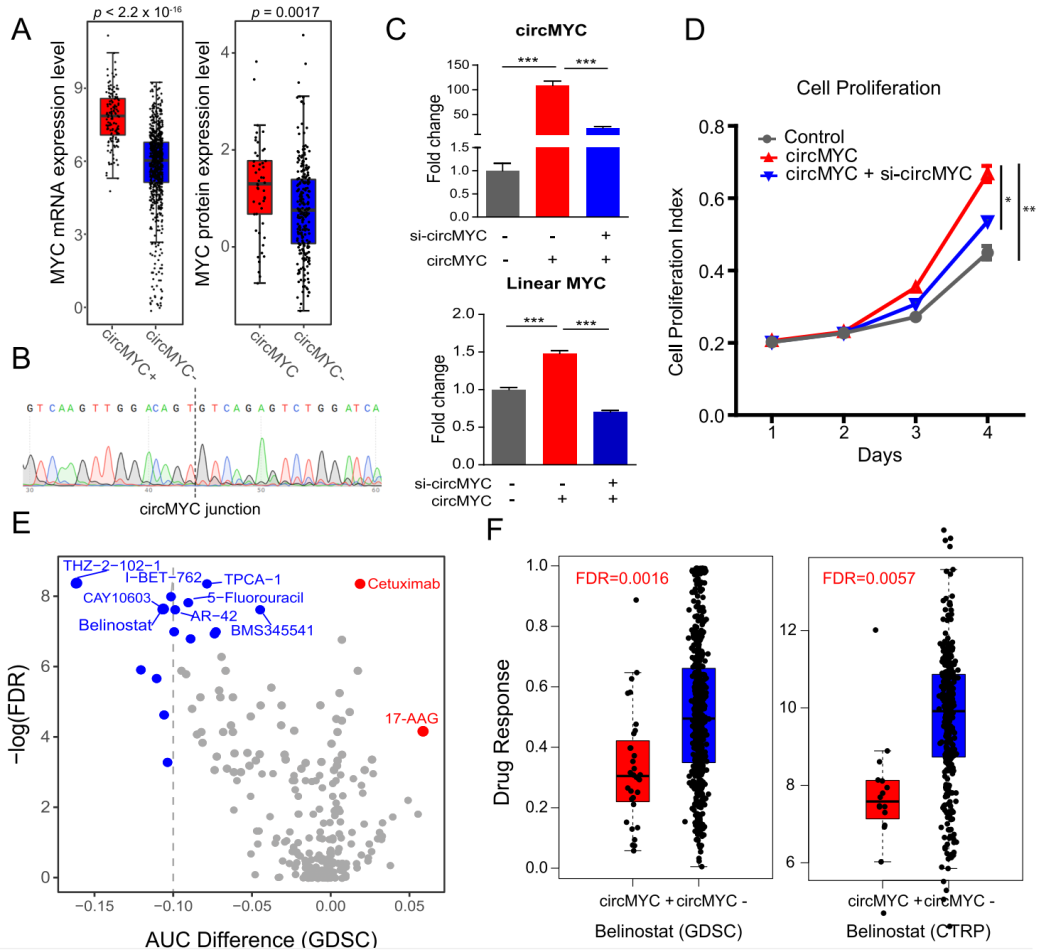
图4 circMyc功能分析 ([1])
药效相关性circRNA分析
作者系统分析了临床药物治疗效果与circRNA表达谱的相关性,共发现4564种circRNA分子的表达与临床用药效果存在显著的相关性。
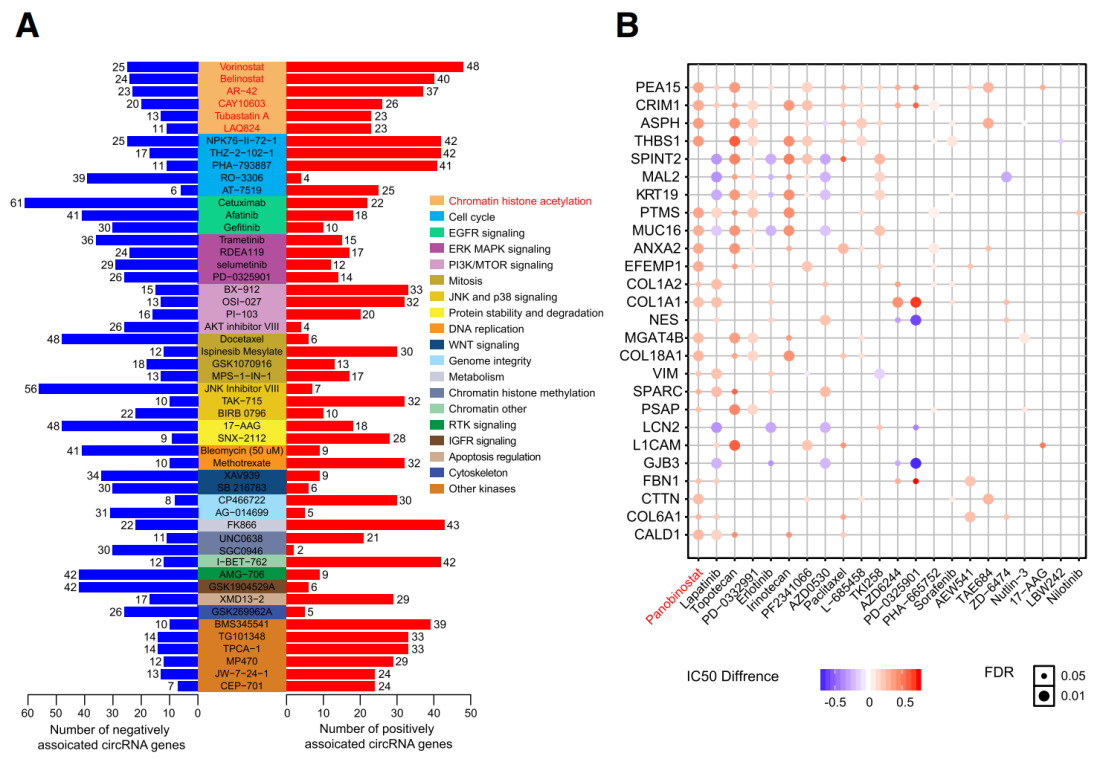
图5 临床药物敏感性相关circRNA分析 ([1])
CircRiC数据库
作者将所分析的接近1000种细胞系的circRNA表达与各种药物敏感性相关的信息整理形成了一个数据库:CircRiC(网址:https://hanlab.uth.edu/cRic)。感兴趣的同行可以去数据库检索使用。
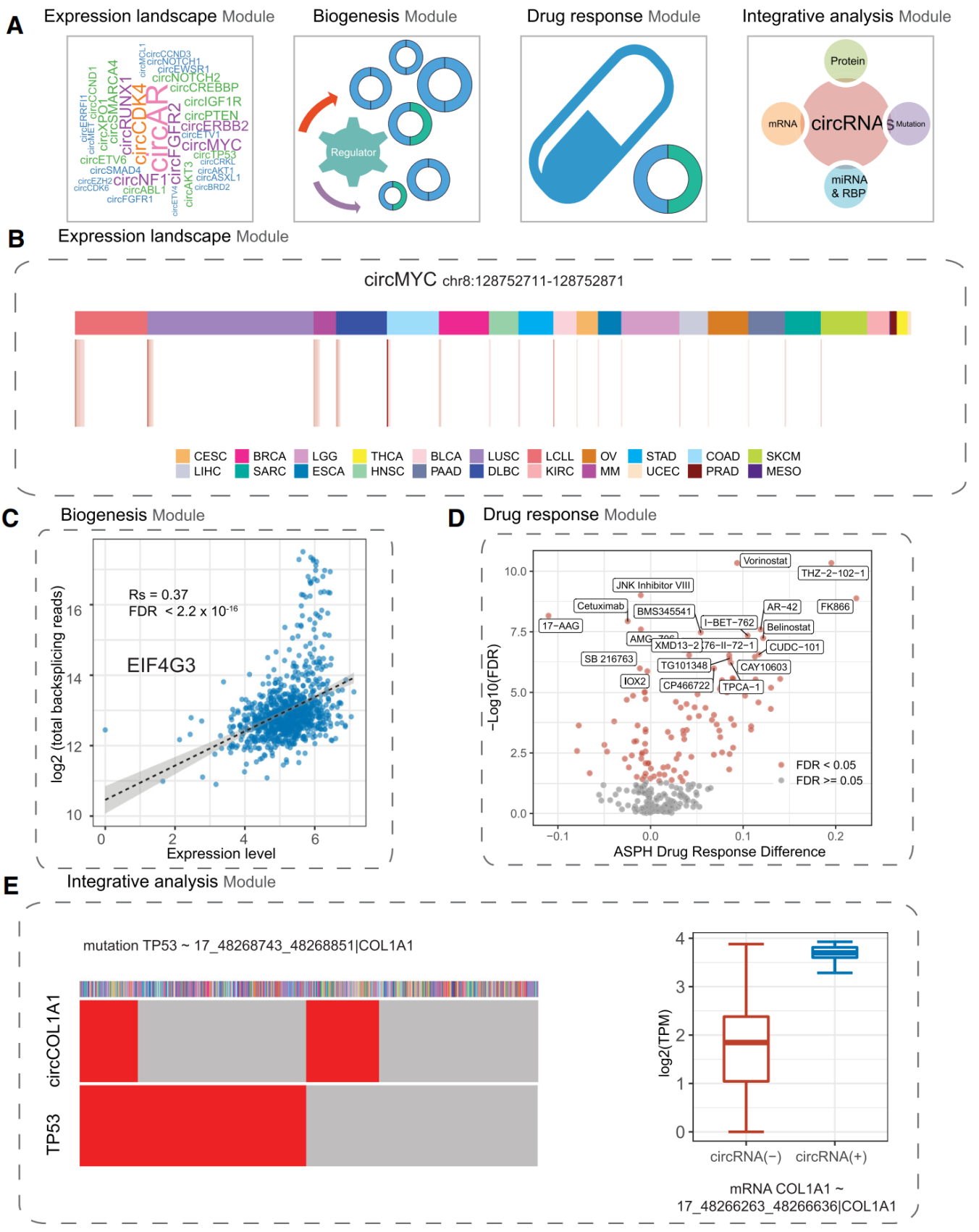
图6 CircRiC数据库 ([1])
参考文献
1. Ruan, H., et al., Comprehensive characterization of circular RNAs in ~ 1000 human cancer cell lines. Genome Med, 2019. 11(1): p. 55.
.png)
