10月16日, Nature Communications杂志在线发表了一项circRNA m6A修饰的文章,报道发现m6A修饰的circNSUN2可结合YTHDC1并促进出核,进一步结合IGF2BP2促进HMGA2 mRNA的稳定,最终促进大肠癌的肝转移增强[1]。文章的通讯作者是中山大学附属肿瘤医院的谢丹教授,徐瑞华教授和Wang Fengwei。

以m6A为代表的RNA修饰是近几年的热点研究方向,circRNA目前已有多篇文章报道可携带m6A的修饰。推荐阅读:
BioRxiv:HPH模型中circRNA的m6A修饰
m6A介导mRNA和circRNA降解
重大发现:circRNA存在细胞特异性的m6A修饰
重磅:m6A修饰促进环状RNA翻译
本文的机制表明m6A修饰可调控circRNA的出核,是circRNA修饰研究的重要进展。下面就让我们一起学习一下这篇文章:
如何找到circNSUN2分子的?
作者通过芯片法分析了两对大肠癌(CRC)与癌旁的circRNA表达谱,共找到13167种circRNA分子。基因拷贝数异常(CNV)的区域经常是肿瘤驱动基因的位置,作者提出是否该区域的circRNA与肿瘤发生发展有关?于是作者选择了CRC最常见的CNV 位点(拷贝数增加的位点:5p15.31,8q24.21,8q24.3,13q12.13:拷贝数降低的位点:5q21,18q21.1),芯片筛选得到的circRNA中比对到这些位点的circRNA有104种,其中与对应位点的增高或降低趋势一致的为38种,9种circRNA的变化趋势在两例肿瘤标本中是一致的。为进一步分析筛选有价值的分子,作者在97例CRC的标本库中分析了这9个circRNA的表达情况,其中4种circRNA在超过75%的标本中呈现出高表达的趋势。这其中circNSUN2在20例原发灶和对应肝转移的标本中表现为肝转移组表达的显著增高,并且伴随着肝转移程度的增加,circNSUN2的表达也呈现显著的增高趋势。在97例标本群体中,circNSUN2高表达的组生存状况显著低于低表达组。基于这些数据,本文选择circNSUN2作为研究的目标分子。
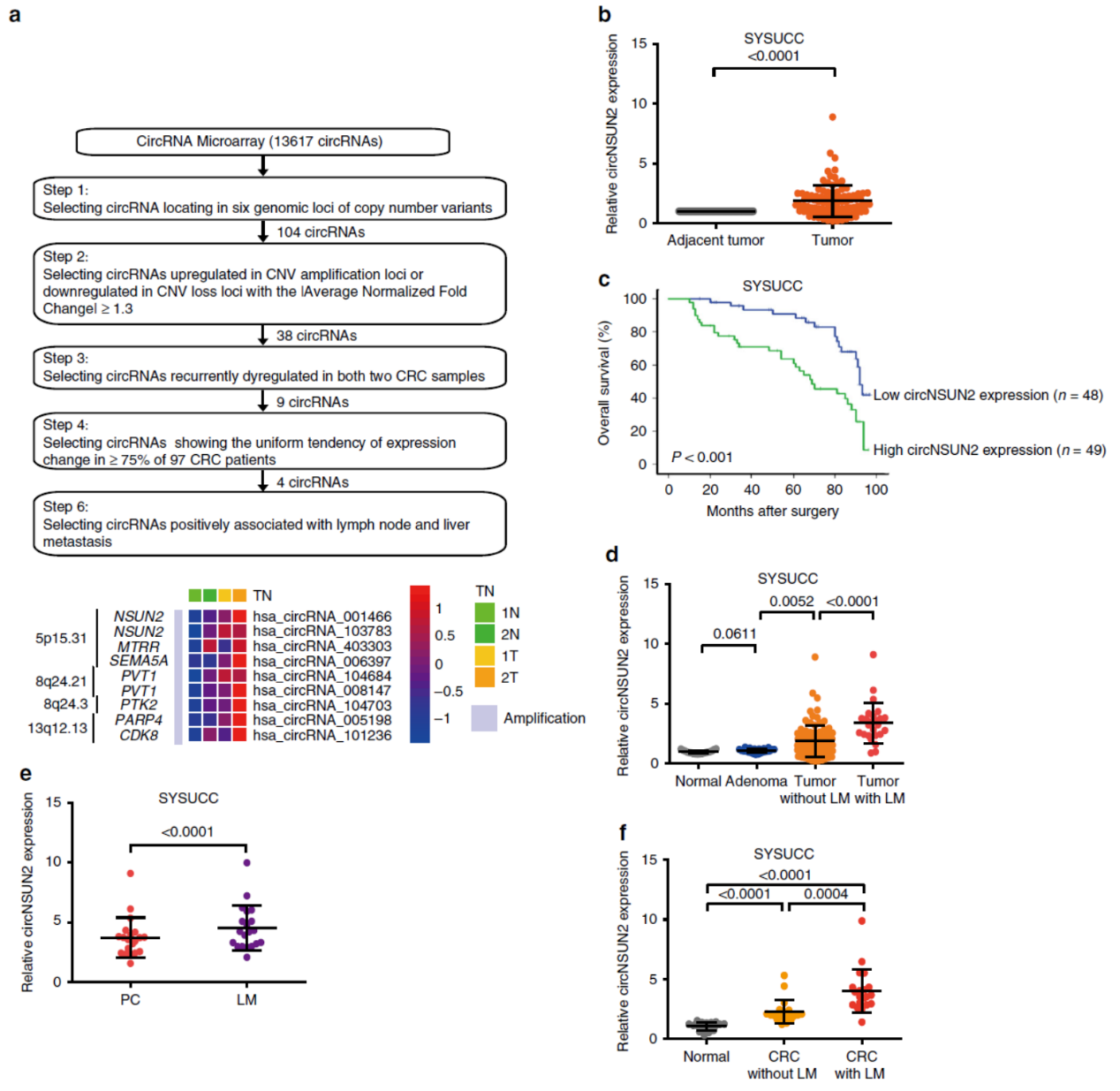
图1 circNSUN2在CRC中高表达,与肝转移相关 ([1])
circNSUN2的鉴定
Sanger测序,反向引物设计,RNase R消化,Northern blot,放线菌素D处理后稳定性分析, FISH和亚细胞组分分析实验鉴定了circNSUN2的疾病情况。circNSUN2主要定位于胞质中,
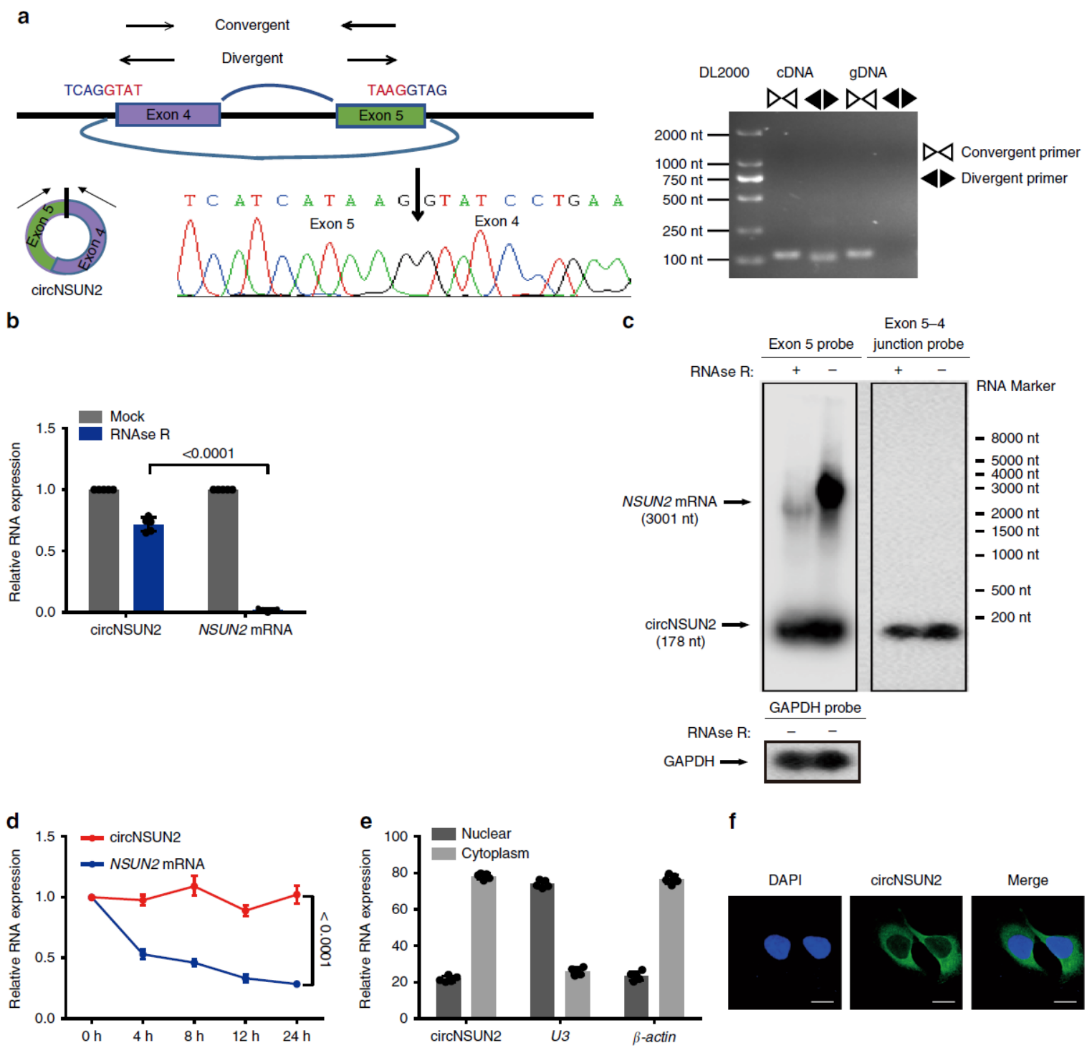
图2 circNSUN2的鉴定 ([1])
体内实验证明circNSUN2与CRC的肝转移相关
TC17细胞或PDX模型的细胞做shRNA稳定敲降或过表达circNSUN2,分析肝转移效率的影响情况。结果均表明干扰circNSUN2可以降低CRC的肝转移而过表达circNSUN2则促进肝转移。
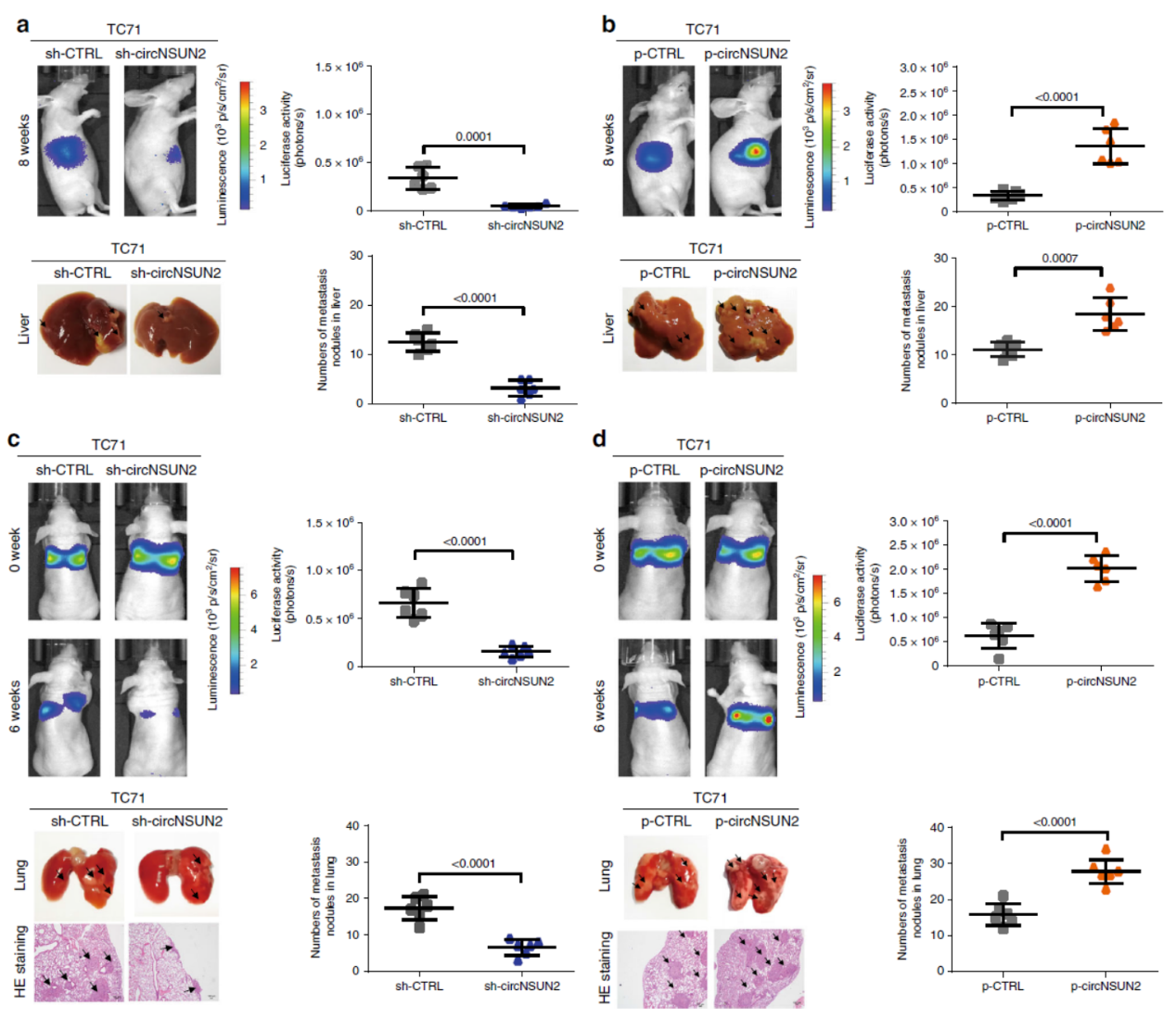
图3 动物模型实验证明circNSUN2促进CRC的肝转移 ([1])
circNSUN2与YTHDC1结合后促进出核
为探索circNSUN2的分子机制,作者通过RNA pull-down分析了相互作用的蛋白,差异条带质谱分析后发现了YTHDC1和IGF2BP2。pull-down产物Western检测,anti- YTHDC1的RIP实验均证明了circNSUN2与YTHDC1的相互作用。YTHDC1是已知的RNA m6A的Reader分子之一,上述结果证明了circNSUN2与YTHDC1的相互作用,是否意味着circNSUN2也可能携带了m6A修饰?meRIP实验证明了circNSUN2确实可以被m6A的抗体富集。序列分析预测到了circNSUN2中存在GAACU motif(已知的m6A修饰位点),设计突变实验,将其中的A突变为G,EMSA实验分析突变前后与YTHDC1的相互作用情况,结果表明突变后不再与YTHDC1相互作用。这就证明了circNSUN2的确存在m6A修饰,该修饰方式介道了与YTHDC1的相互作用。干扰YTHDC1后发现circNSUN2的亚细胞分布发生了变化,干扰后核内circNSUN2的丰度增高了。FISH定位分析也验证了这一现象。
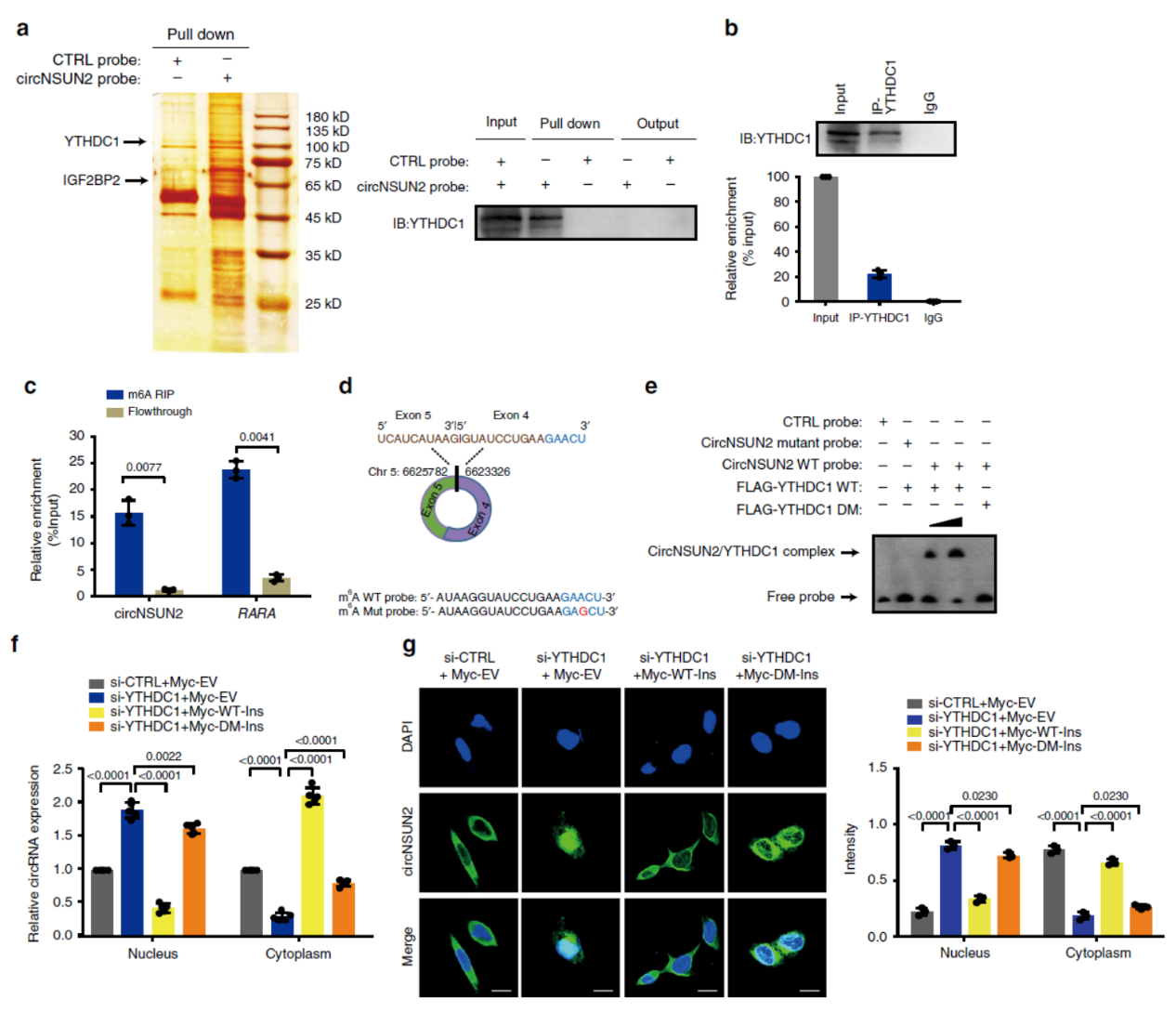
图4 circNSUN2与YTHDC1结合后促进出核 ([1])
circNSUN2与IGF2BP2结合
pull-down实验中还发现了circNSUN2可结合IGF2BP2,作者也对该蛋白进行了类似的分析,包括pull-down后Western分析,RIP实验。免疫荧光-FIH共定位分析也证明了circNSUN2与IGF2BP2的相互作用。IGF2BP2是一种RNA结合蛋白,与mRNA的稳定性有关,已知在CRC中与侵袭有关。作者首先个截短突变体实验分析了IGF2BP2结合circNSUN2的结构域位置,结果表明KH3-4 di-domain是特异性结合circNSUN2的结构域。之前已报道IGF2BP2结合RNA的motif是CAUH (H = A, U or C),在circNSUN2中有该基序存在,于是基于位点突变前后EMSA分析,确定了circNSUN2与IGF2BP2结合。
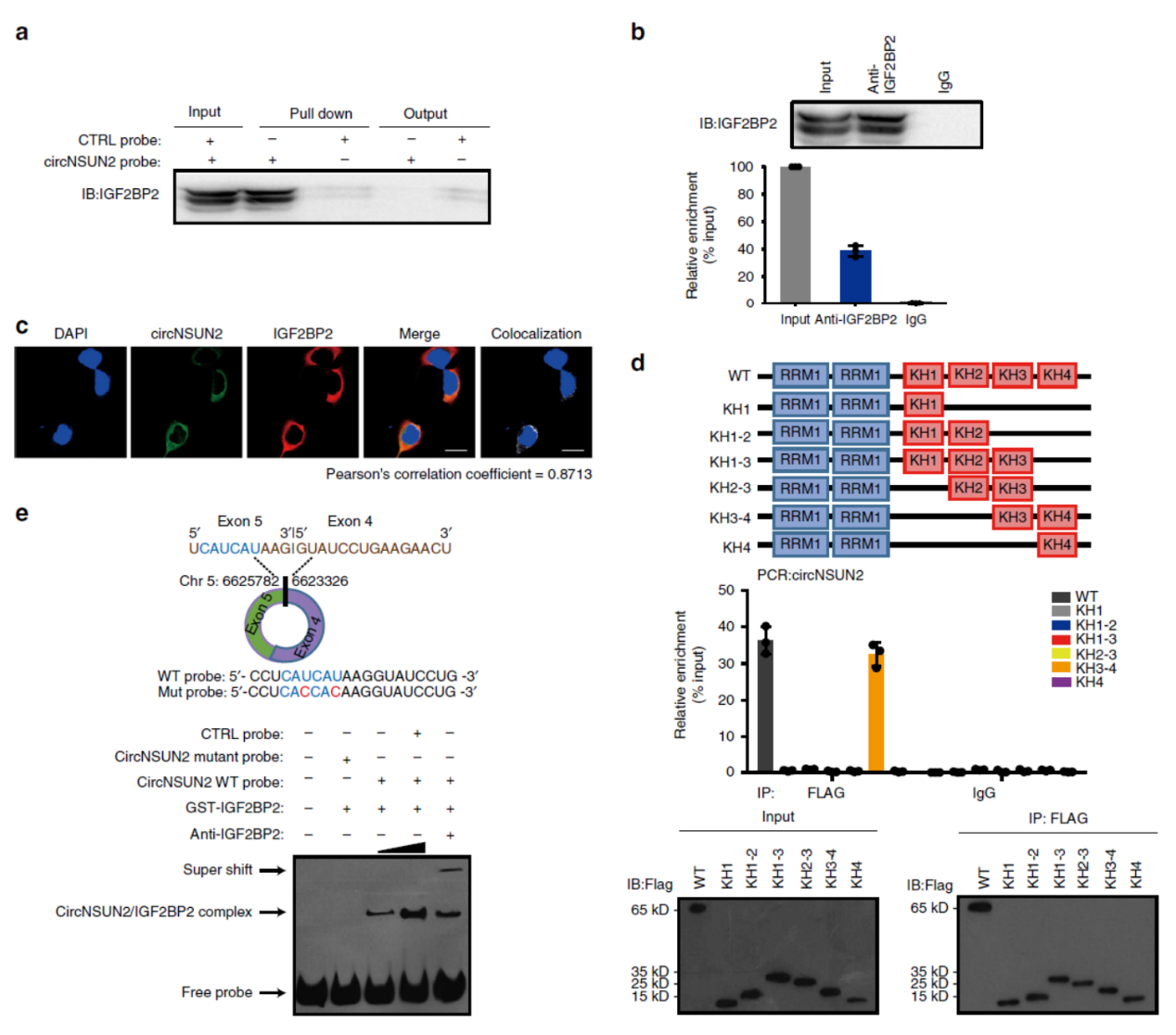
图5 circNSUN2与IGF2BP2结合 ([1])
circNSUN2/ YTHDC1/IGF2BP2复合体促进HMGA2 mRNA稳定
已知IGF2BP2与mRNA的稳定性有关,那么这里发现的circNSUN2与IGF2BP2的相互作用是否会影响一些mRNA的稳定性?为弄清楚这个问题,作者设计了干扰circNSUN2前后分析转录组的差异情况,结果有644种mRNA在干扰circNSUN2丰富发生显著变化。IGF2BP2通常结合mRNA 的3’UTR区富含AU的序列,基于这一规律,作者基于已发表的CLIP数据分析了这644种mRNA的情况,其中有21种mRNA一直是可以结合IGF2BP2的。circNSUN2与CRC肝转移有关,结合QPCR和Western检测的数据,最终选择HMGA2作为靶分子。RNA pull-down实验证明了circNSUN2的确可以捕获HMGA2 mRNA。干扰circNSUN2后HMGA2 mRNA的稳定性显著降低。荧光素酶报告基因实验,免疫荧光-FISH共定位分析证明了circNSUN2/ YTHDC1/IGF2BP2复合体与HMGA2 mRNA的结合。IGF2BP2分段突变和circNSUN2干扰等实验进一步证明了他们相互作用的位点和序列信息。
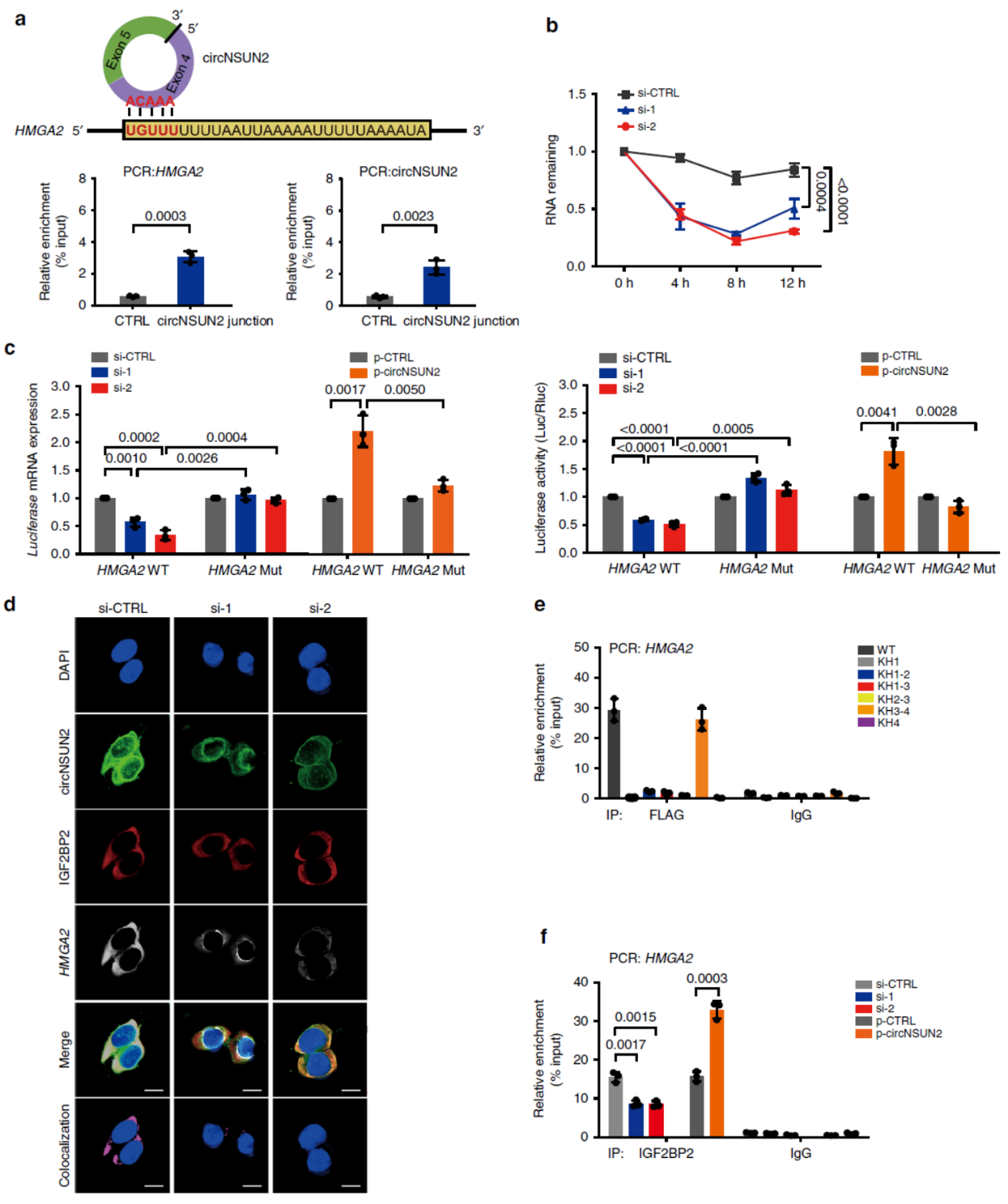
图6 circNSUN2/ YTHDC1/IGF2BP2复合体功能 ([1])
体内实验证明HMGA2是circNSUN2调控CRC肝转移的介导者
最后,作者在体内模型中验证了上述分子模型与CRC肝转移的相关性。单独干扰circNSUN2可抑制肝转移,但如果同时过表达HMGA2则促进肝转移。
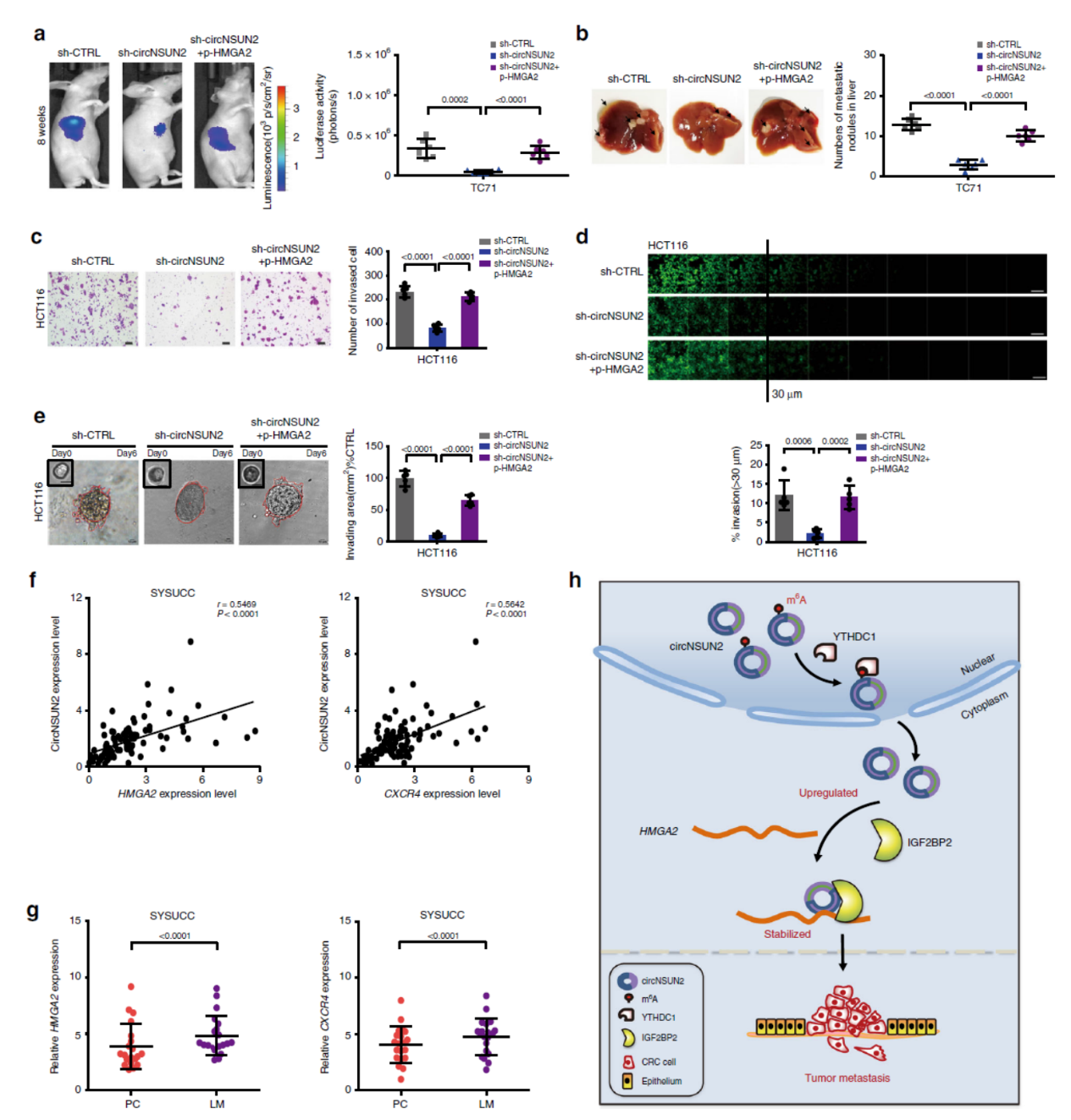
图2 体内模型证明circNSUN2通过HMGA2促进CRC肝转移 ([1])
本文的故事非常精彩,有几个值得学习的思路总结如下:
1. 从已知的疾病相关CNV位点(很可能是肿瘤的驱动基因所在的位置)出发收缩circRNA筛选的目标,这样可能有助于找到对疾病发生发展关系更紧密的分子。
2. RNA Pull-down找到了circRNA相互作用的蛋白,是揭示circNSUN2分子功能的关键一步。
3. 通过circNSUN2的相互作用蛋白的捕获,发现了m6A的Reader蛋白YTHDC1,进一步发现了circNSUN2出核的机制。本文最初并不是要分析circRNA的m6A修饰的,互作蛋白的捕获帮助作者发现了circNSUN2的m6A修饰对它出核的作用。
总之,本文的工作非常精彩,从小样本临床标本的分析入手,找到CRC肝转移相关的circRNA分子,然后基于RNA Pull-down分析,发现了circNSUN2的相互作用蛋白YTHDC1和IGF2BP2,进而从干扰circNSUN2前后转录组差异分析结合作者所发现的现象找到HMGA2 mRNA是circNSUN2促进CRC肝转移的介导分子,故事前后逻辑衔接非常通顺,是circRNA机制研究的重要参考工作。
参考文献
1. Ri-Xin Chen, X.C., Liang-Ping Xia, Jia-Xing Zhang, Zhi-Zhong Pan, Xiao-Dan Ma, Kai Han, Jie-Wei Chen, Jean-Gabrie Judde, Olivier Deas, Feng Wang, Ning-Fang Ma, Xinyuan Guan, Jing-Ping Yun, Feng-Wei Wang, Rui-Hua Xu, Dan Xie N6-methyladenosine modification of circNSUN2 facilitates cytoplasmic export and stabilizes HMGA2 to promote colorectal liver metastasis. Nature Communications, 2019.
怎样找到有功能的circRNA分子?
是否希望更准确的发现circRNA的相互作用分子?
如何研究circRNA的m6A修饰?
circRNA 研究思路与技术培训班干货满满,你所关心的问题都能找到答案,名额有限,抓紧时间报名啦:
.png)