环状RNA研究越来越受到国内外研究学者的青睐,有关环状RNA的一些基本科学问题也不断取得新的认知。近日,知名预印本杂志BioRxiv发表了一项最新的circRNA研究进展,报道发现一种新的CDR1as的生成机制,由哺乳动物的散在重复元件(Mammalian-Wide Interspersed Repeats,MIR)介导[1]。文章的通讯作者是日本藤田健康大学的Akila Mayeda,著名circRNA研究专家Jørgen Kjems也是本文的共同作者。

如何发现MIR与CDR1as生成的相关?
作者分析了人和小鼠CDR1as两侧的基因组序列,发现在人的基因组序列中只有上游存在两个Alu序列,下游没有,因此不能简单的用反向互补Alu序列解释CDR1as的形成。有趣的是,作者在人和小鼠的相应基因组序列中发现了另外一种短散在重复元件(SINE),称为哺乳动物的散在重复元件(Mammalian-Wide Interspersed Repeats,MIR)。在CDR1as的两侧,MIR元件是反向排列的。
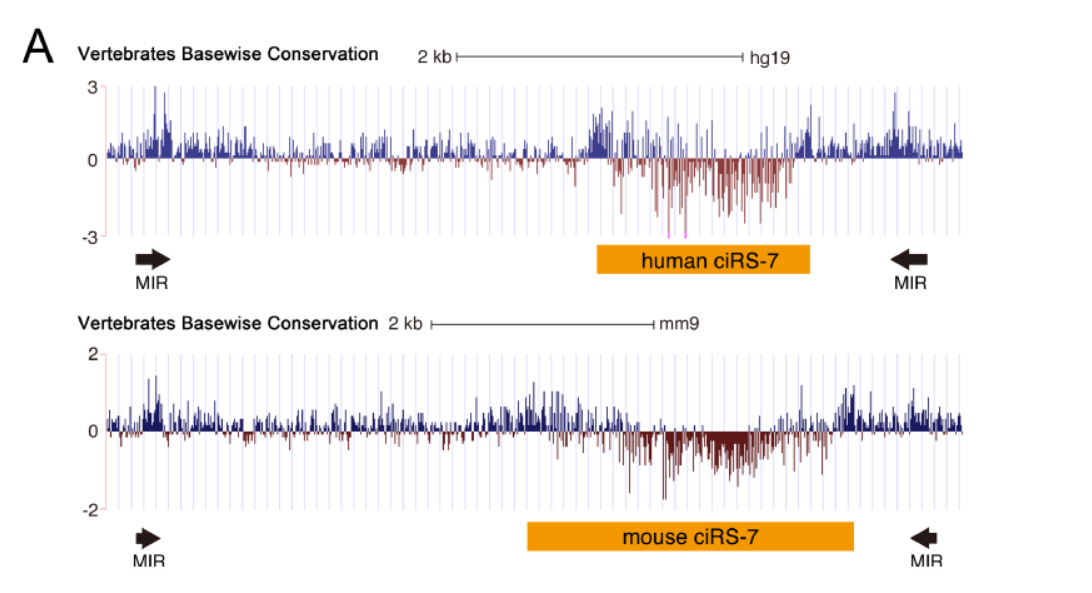
人与小鼠CDR1as两侧存在反向MIR元件([1])
如何验证MIR介导CDR1as的形成?
基于上述发现,作者构建了模拟CDR1as生成的mini基因载体,该载体基于四环素诱导系统,当添加强力霉素(Dox,一种四环素的类似物,广泛用于Tet诱导体系)后,可诱导下游序列的转录表达。前体mRNA剪切抑制剂Spliceostatin A用于抑制转录后RNA的剪切作用。通过Northern杂交,作者验证了这一载体系统的有效性。
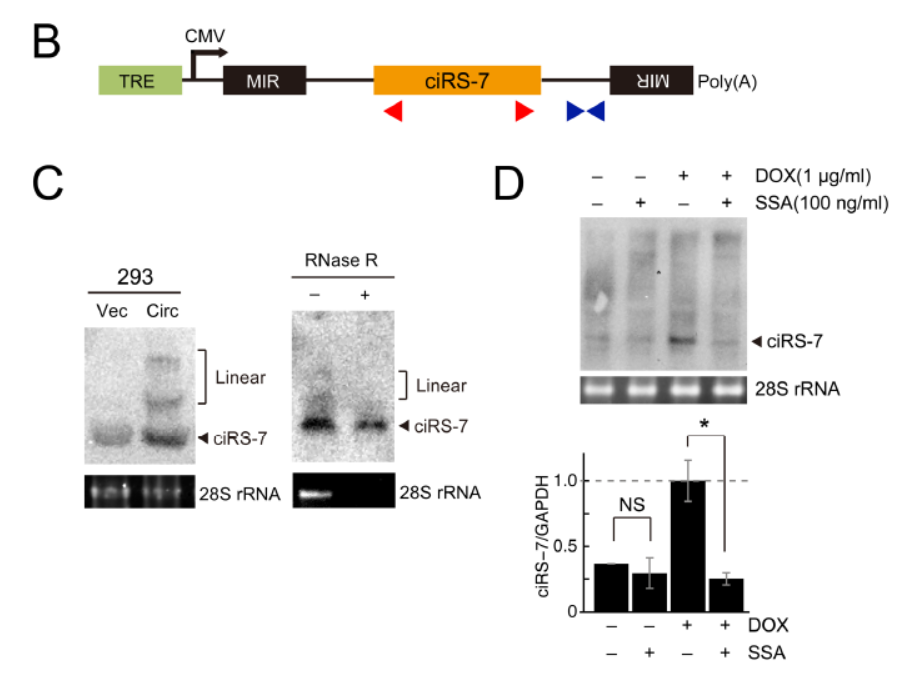
Mini基因分析CDR1as 形成 ([1])
在这一载体系统的基础上,作者依次构建了上游MIR,下游MIR及两者共同删除的突变体,以验证MIR对CDR1as形成的作用。结果证明,无论删除上游还是下游MIR均能显著降低CDR1as的生成效率。进一步,作者还在SH-SY5Y细胞中利用Cas9打靶,精确敲除上游或下游的MIR元件,然后QPCR分析敲除株中CDR1as表达量。结果证明基因组中敲除上游或下游MIR均可显著抑制CDR1as的生成。
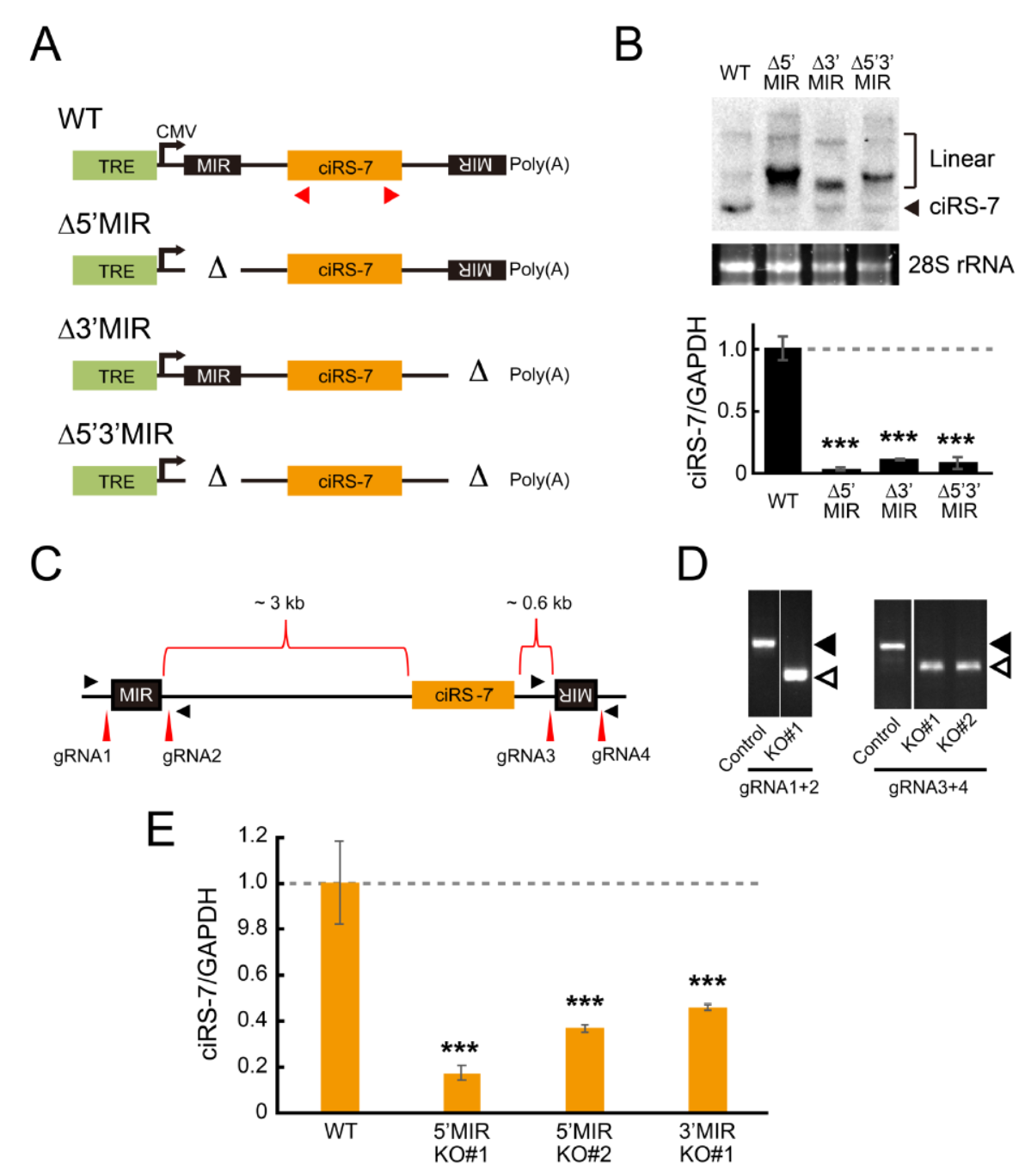
删除上下游MIR均抑制CDR1as 的生成 ([1])
其他受MIR调控生成的circRNA
在hg19基因组序列中,作者共分析到595094种MIR元件,与小鼠mm9基因组分析的结果比对后,104074种MIR是保守的。进一步,作者发现了有171种circRNA的上下游1000bp范围内同时存在反向互补MIR元件。这些circRNA中有一些circRNA的两侧序列非常长,难以构建报告载体验证,作者挑选了5种circrNA进行了mini基因载体的构建和验证:circCDK8, circSPNS1, circTMEM109, circZYMND8和circSRGAP2C。验证结果表明删除这些circRNA的上下游反向互补MIR可显著影响它们的生成效率。
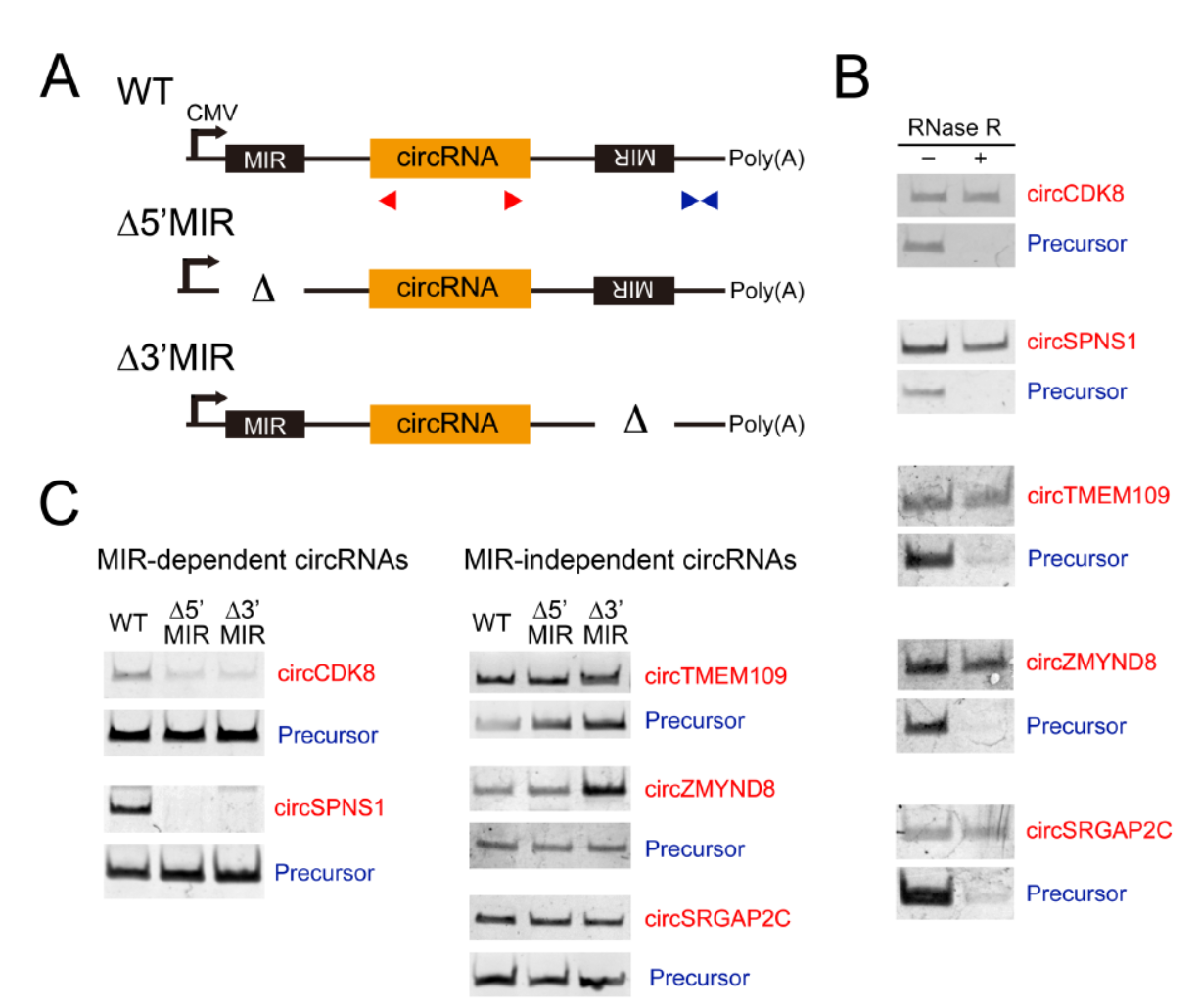
受MIR调控生成的circRNA验证 ([1])
调控circRNA生成的MIR元件特征
MIR在进化中比Alu元件更古老,很多MIR元件因此变得短了。如果是这样的,那么在不是MIR依赖生成的circRNA两侧的MIR元件势必没有依赖MIR生成的circRNA两侧的MIR元件保守性好。为验证这一假设,作者分析了MIR依赖和不依赖的circRNA两侧序列中MIR元件的Smith-Waterman值,结果确实证明MIR依赖的circRNA两侧SW值明显高,且MIR元件的长度也更高,更完整。在所发现的171种circRNA中,SW值大于250的有118种,SW值大于500的有19种,随着SW值的增加,对应的circRNA两侧的MIR元件的热力学稳定性也越高。
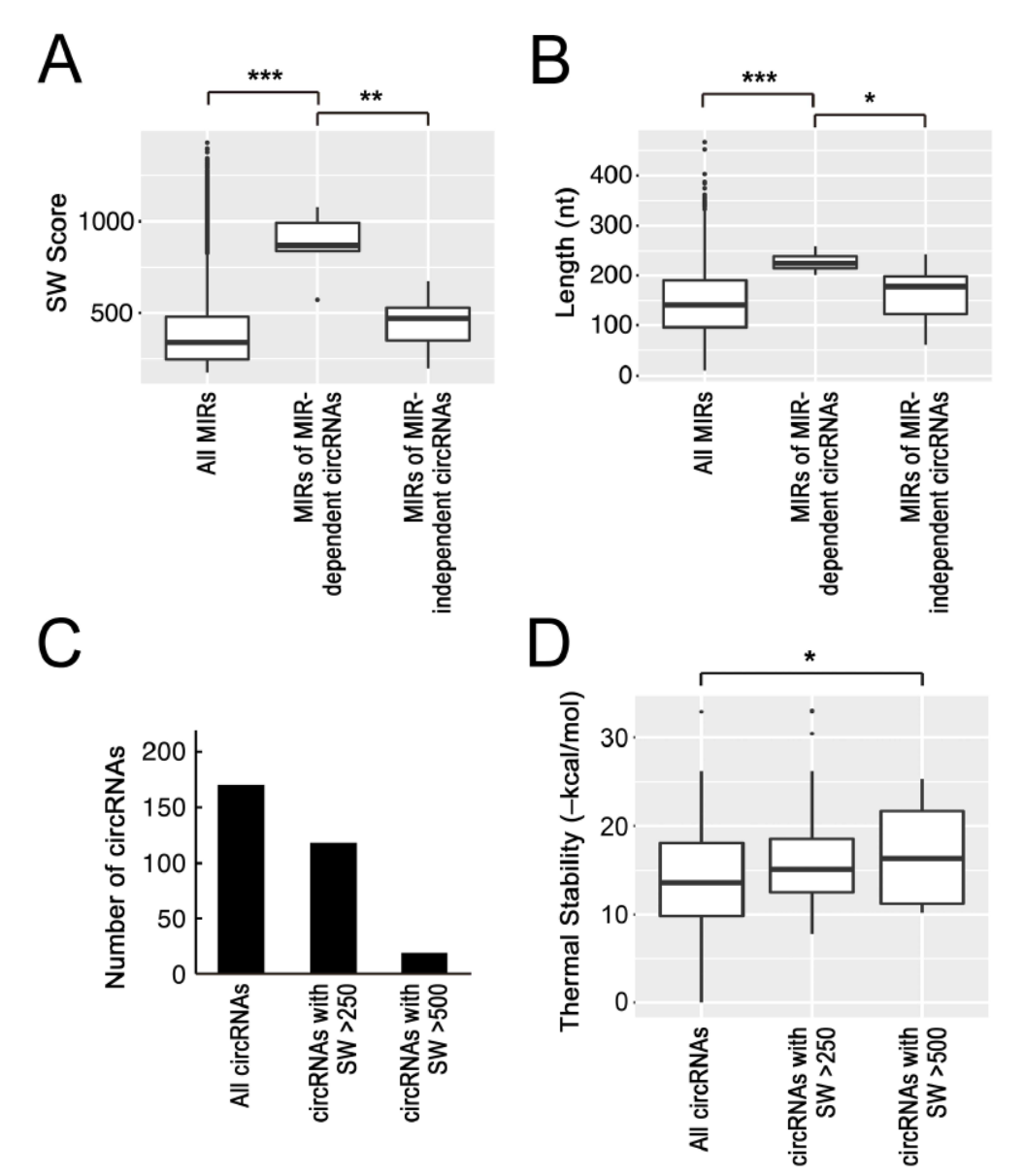
介导circRNA生成的MIR特性分析 ([1])
最后,作者还分析了反向互补MIR或Alu序列的热力学稳定性情况,结果表明反向互补MIR元件的热力学稳定性不如Alu元件的高。计算热力学稳定性采用的是RNAup工具。
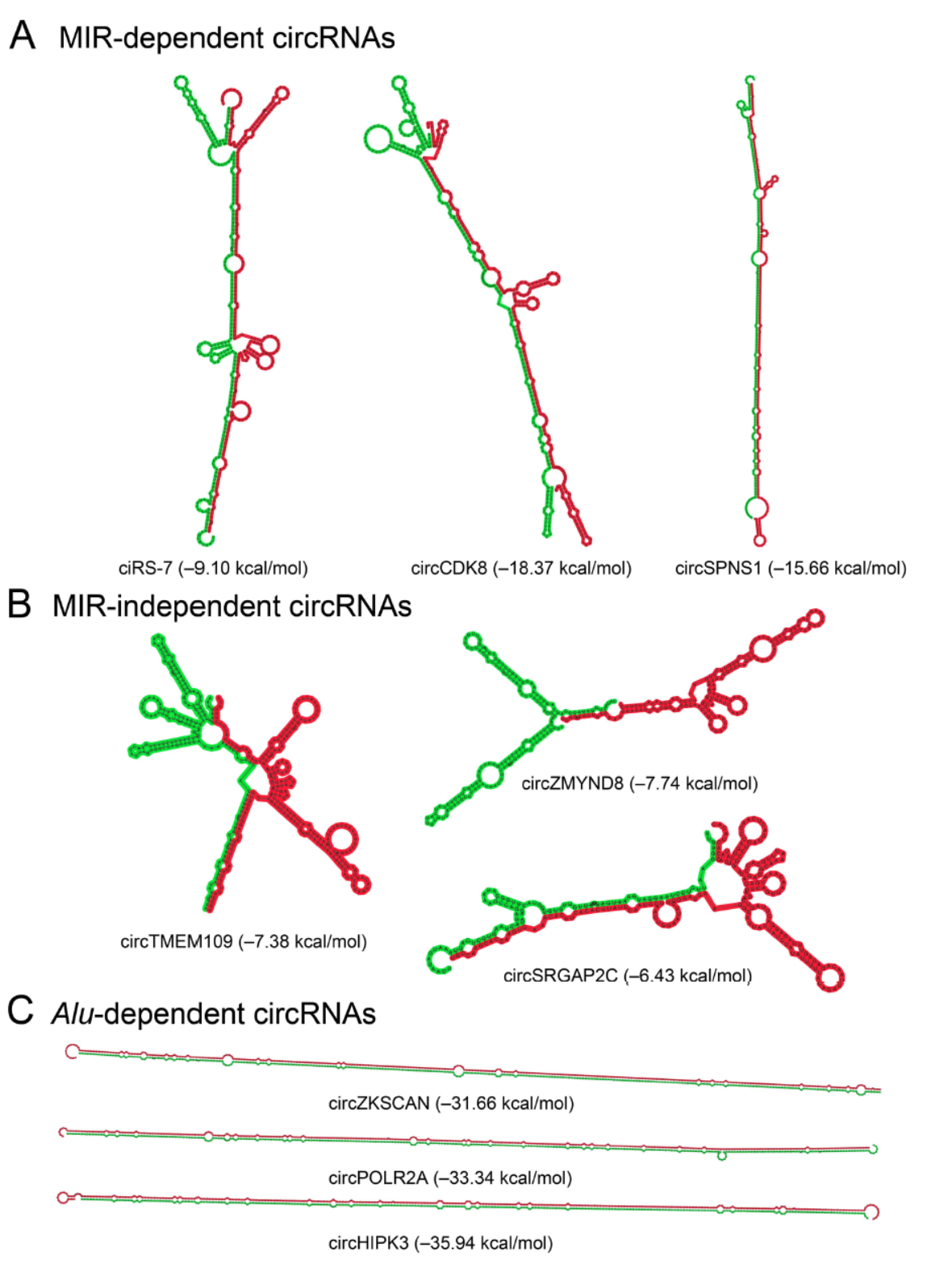
反向互补MIR或Alu热力学稳定性预测分析 ([1])
参考文献
1. Rei Yoshimoto, K.R., Thomas Hansen, Jørgen Kjems & Akila Mayeda, A., Biosynthesis of Circular RNA ciRS-7/CDR1as Is Mediated by Mammalian-Wide Interspersed Repeats (MIRs). BioRxiv, 2019.
.png)
