现在对PubMed查询结果进行汇总分析的工具有很多,有不少分析工具是收费的,现在向大家介绍一个免费分析的工具——PubReMiner(或许有些人用过这个工具),其不仅可以对PubMed的查询结果进行高效率的汇总分析,还会对分析结果按照年份、期刊、作者、关键字、文献类型等分类分析。
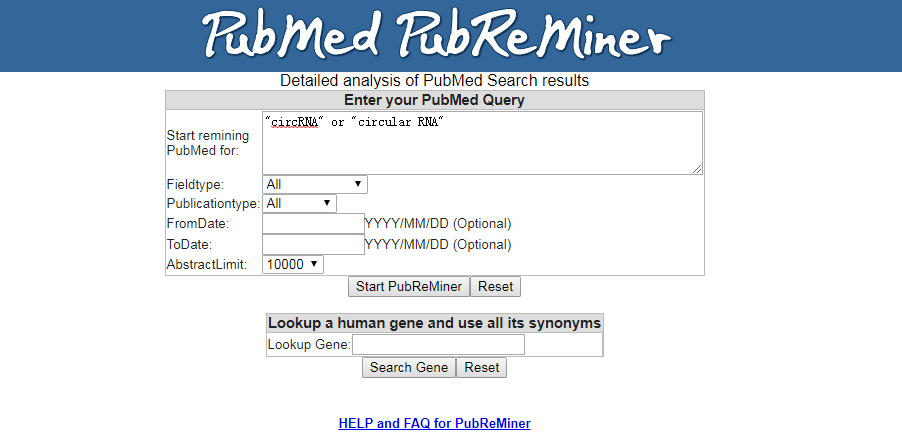
1. 首先进入https://hgserver2.amc.nl/cgi-bin/miner/miner2.cgi网站,如下界面,输入检索关键字或者pubmed的高级检索式,根据需要修改出版日期,同时一定要将Abstractlimit设为10000,才可以进行检索。
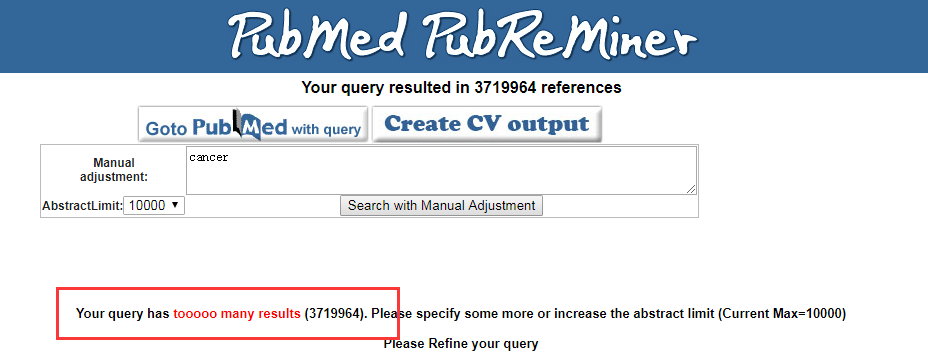
2. 有些关键字如cancer检索结果会出现以下情况,这个时候得先修改出版日期,可改为最近三个月或者更短时间(这不会影响到检索结果,因为有助于筛查出更适合自己的期刊),同时一定要将Abstractlimit设为10000,重新检索;
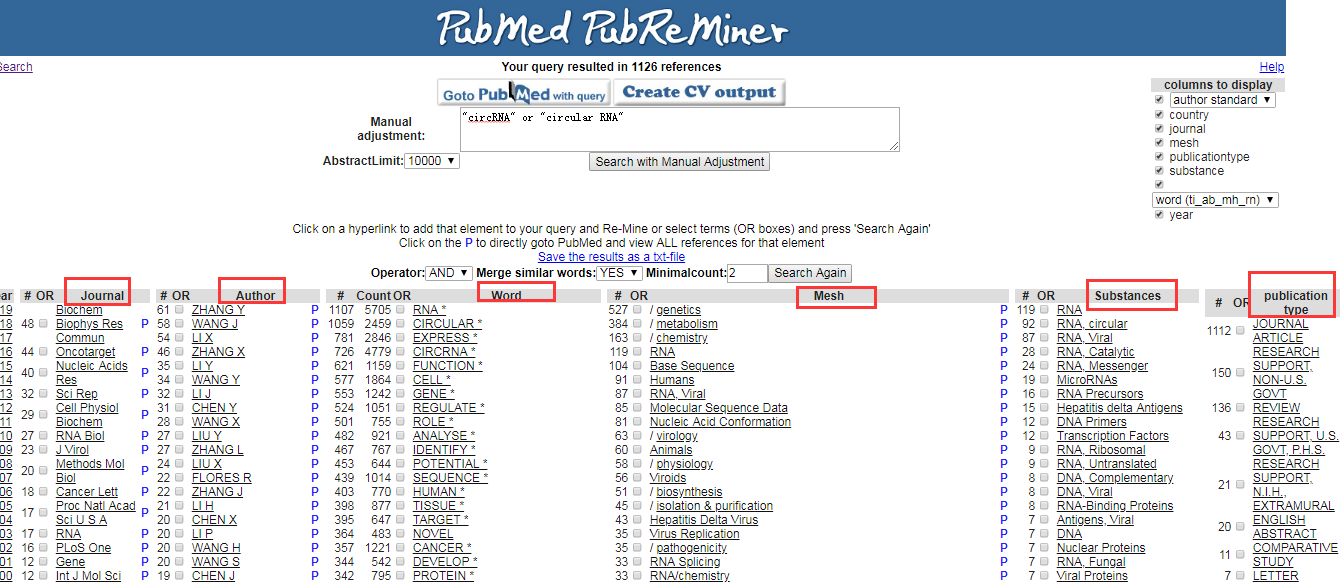
3. 关键字”circRNA” or “circular RNA”的检索结果:按年份、期刊、作者、涉及关键字、主题词和文献类型等分类,“#”指的是对应项目的文献数量;同时我们也可以对右上角的选项勾选,选择展示的内容;根据关键字检索,可以看到2016年到2018年circRNA相关文献发表数量递增;可以看到自己想投的期刊出版有关circRNA研究的情况;
4. 我们可以对不同期刊的出版数量进行统计分析,选择适合投的期刊,例如其中高分期刊Nucleic Acids Res发表有关circRNA研究数量为40篇,点击蓝色的“P”,可以链接到pubmed的检索结果,但是这个链接需要在此基础上加上(“circRNA” or “circular RNA”)一起检索,才可以看到该期刊发表过的有关circRNA的研究;
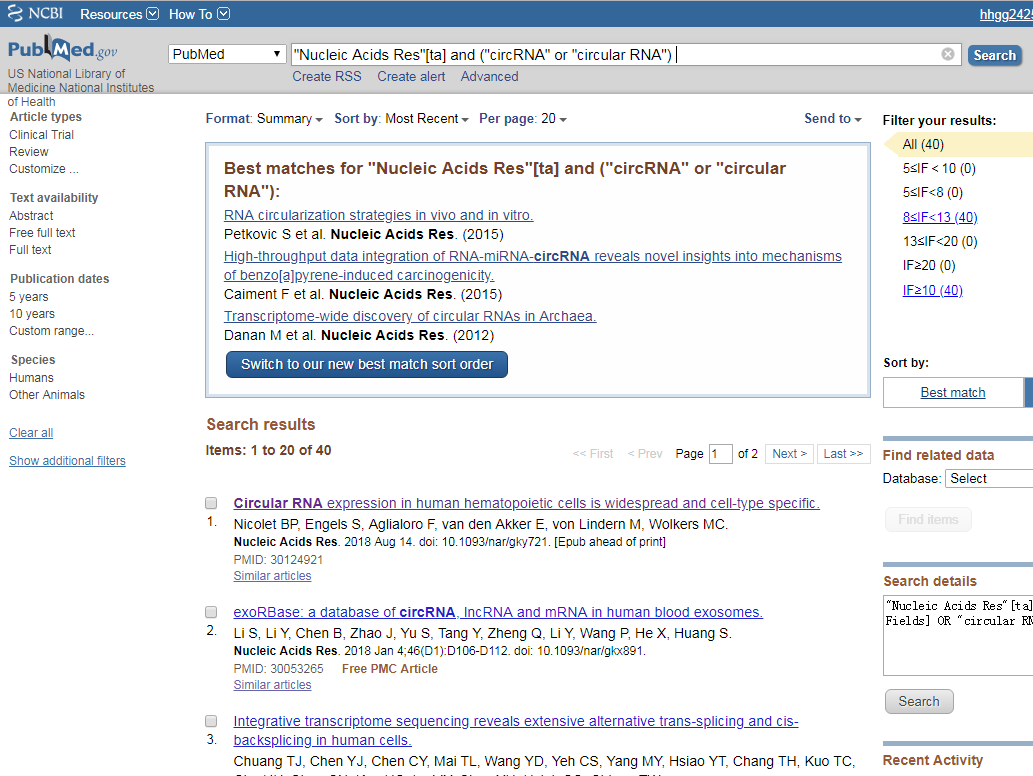
5. 对于author分类项,点击作者那栏的“P”后,也可以链接到pubmed,再加上(”circRNA” or “circular RNA”)一起检索,可以发现对应作者发表的具体文章,但有个缺点就是作者不一定就是文章的通讯作者或者第一作者。
6. 通过分析检索结果中的word和Mesh,我们也可以发现circRNA研究涉及的内容和研究方向,可以为研究人员提供一定课题方向和思路;同时可以对不同的文献类型的数量进行统计。
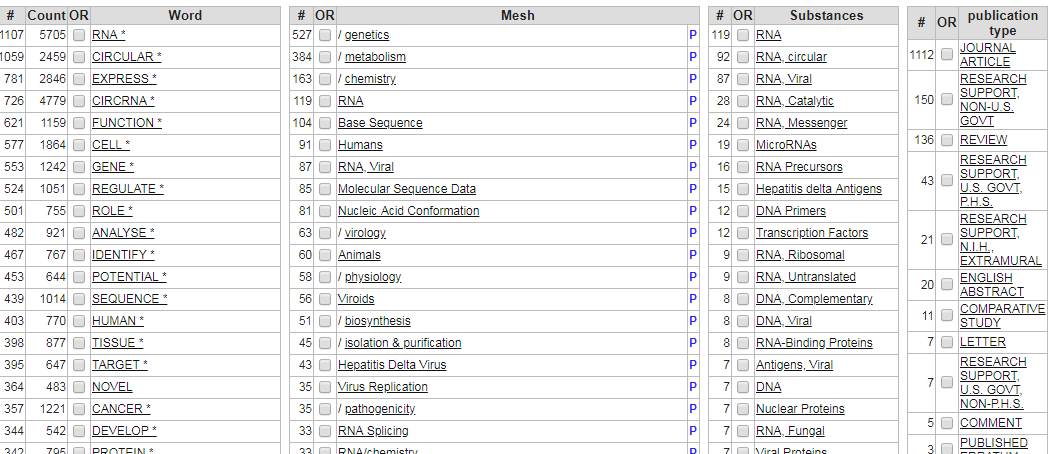
总结: PubReMiner这个汇总分析网站不仅可以对pubmed的检索结果进行初步过滤和分类汇总,更重要的方面还在于为研究者提供研究趋势的预测,根据年份汇总结果分析文章发表趋势,根据期刊文章发表数量预测投刊的命中率,根据发表文章涉及的关键字可以预测研究热点,根据作者发表文章的结果了解该研究领域的大牛,有一定的辅助功能,有兴趣的同学可以了解一下。
.png)
