说起互作网络图,大家都不陌生。看文献的时候,经常看见相互作用网络图展示了circRNA-miRNA的靶向关系或者ceRNA竞争性网络关系。我们先来看几个图:
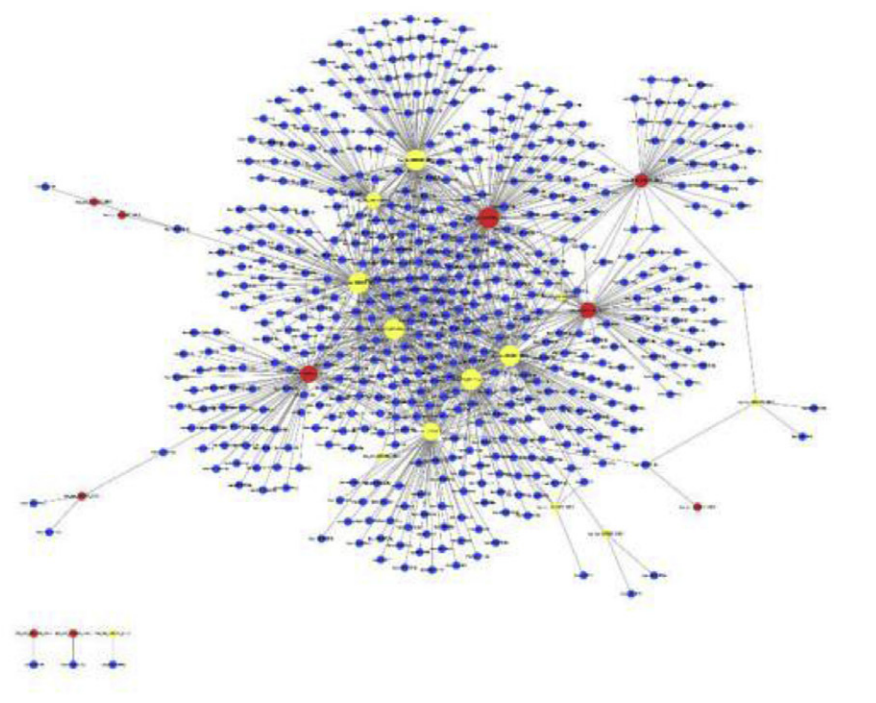
CircRNA-miRNA network maps the interaction relationship between circRNAs and miRNAs [1]
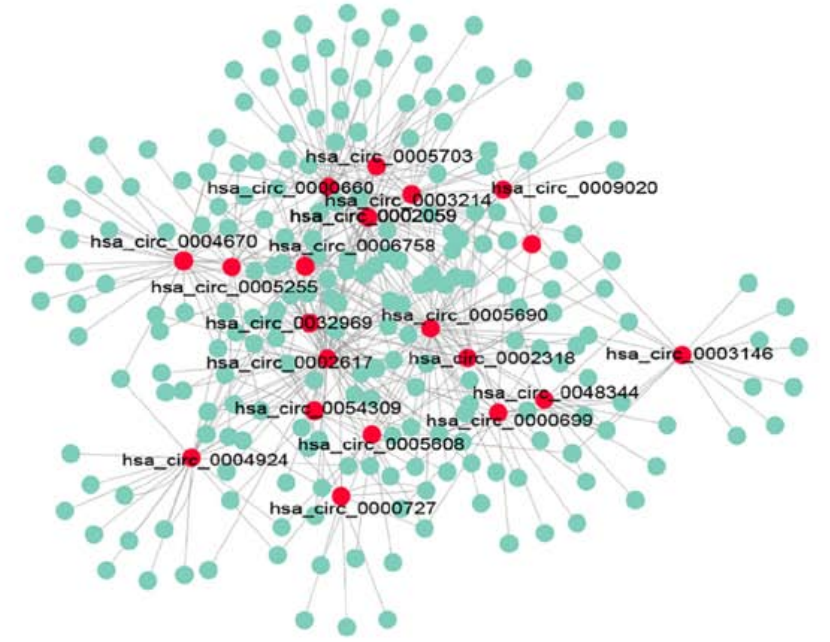
miRNA-ceRNA network. Upregulated circRNAs/miRNAs ceRNA network. [2]
网络图把他们之间的联系进行可视化的展现,为文章增加了几分高大上。这些漂亮而又明了的网络图都是由cytoscape这个神奇的软件绘制而成。大家不要以为网络图看着这么复杂,软件使用也会很复杂,下面就给大家简单介绍一下,如何使用cytoscape把分析数据变成网络图。
步骤如下:
第一,安装cytoscape软件。软件安装比较简单,这里就不展开赘述了。
第二,把要画图的数据进行整理。假设,我在吉赛生物做了circRNA测序,筛选到了一些表达差异的circRNA分子,想要把要研究的circRNA及其靶向miRNA,挑选出来做网络图。
我们会拿到2个表格,circRNA-miRNA线属性(netedges)和点(nodes)属性的表:
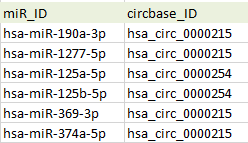
netedges表格示例图
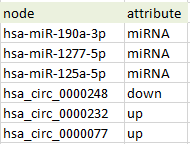
nodes表格示例图
第三,先把靶向关系netedges表格导入cytoscape:
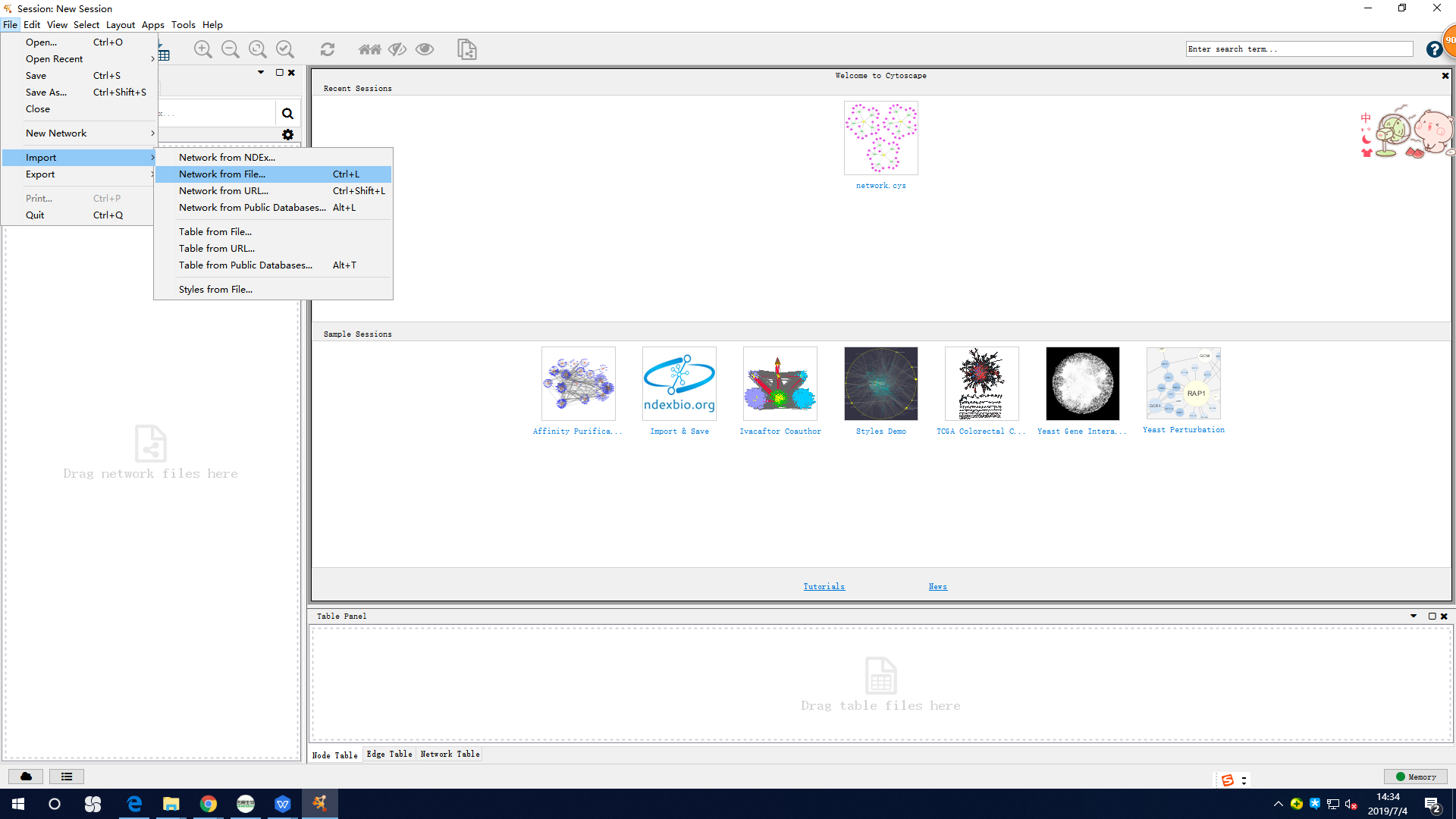
再把设为miRNA源(sourse)节点,circRNA设为靶(target)节点:
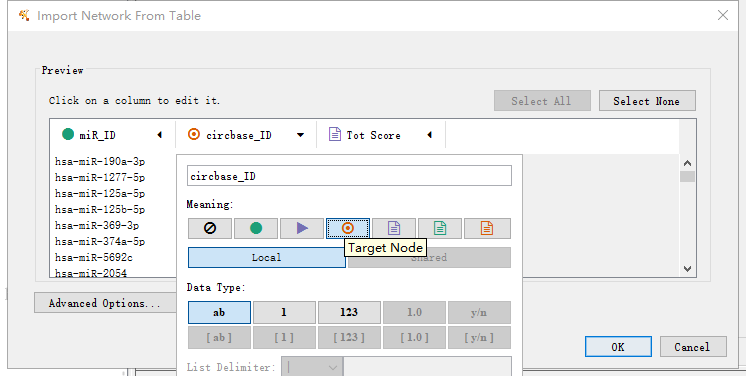
然后点击OK,导入数据,生成初步的网络图:
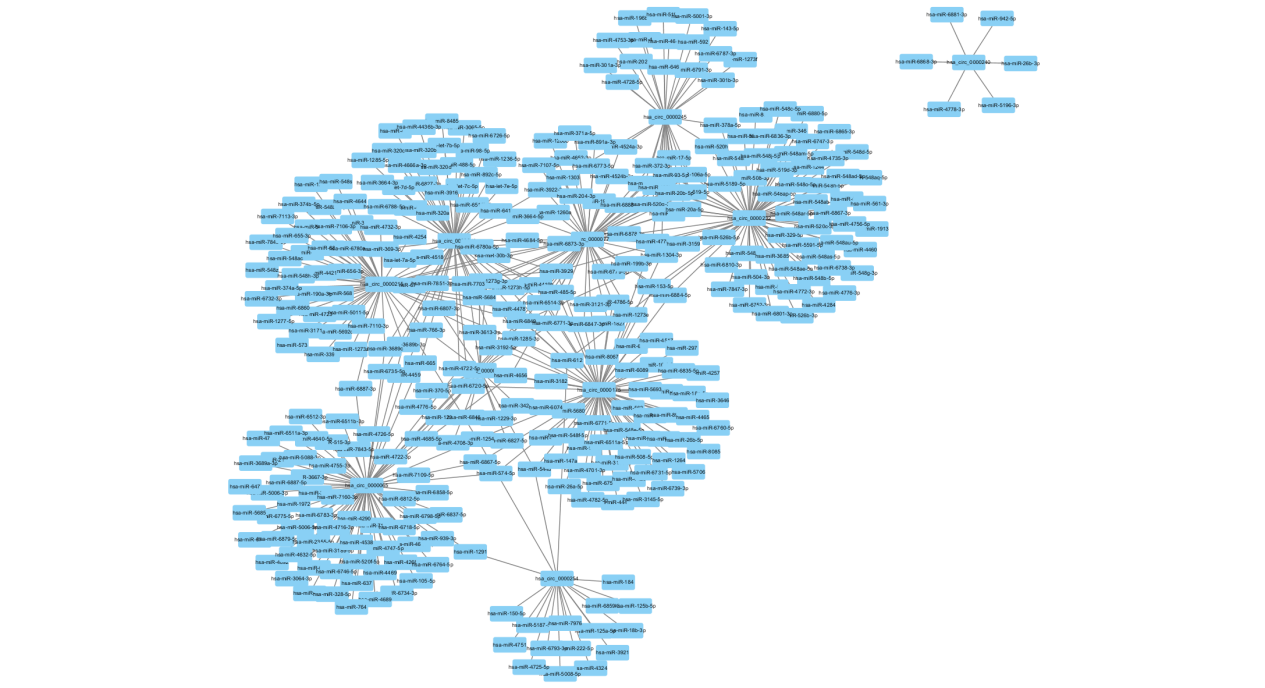
接下来,大家就可以对它进行美化了。一般是通过设置点的不同颜色和形状、大小等来标注circRNA的表达情况,和与哪些miRNA结合。
第四,先把nodes表格作为Table导入,把各个点的属性导入:
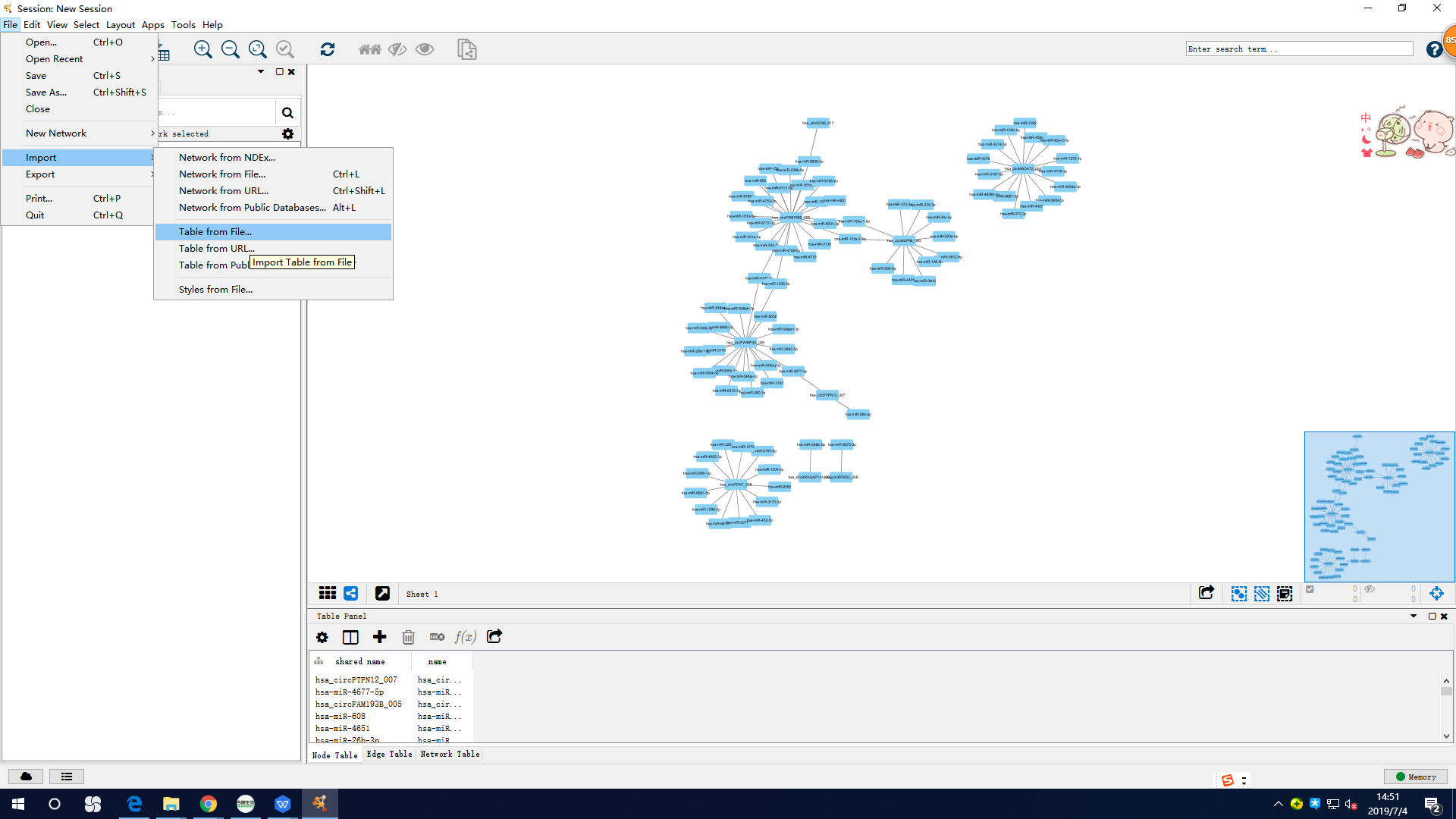
然后,在Style选项卡设置想要的风格,如把表达上调的circRNA标为红色,表达下调的标为绿色,miRNA是预测的,用蓝色标识;则在Fill Color按照attribute进行调整。或者是可以点选某个点进行调整。
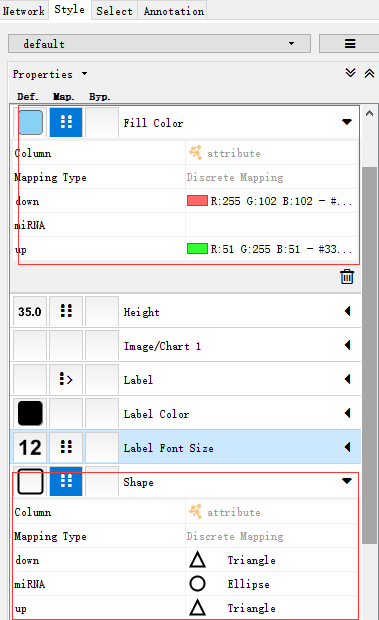
经过一番上色、整形,网络图就变成了这样:
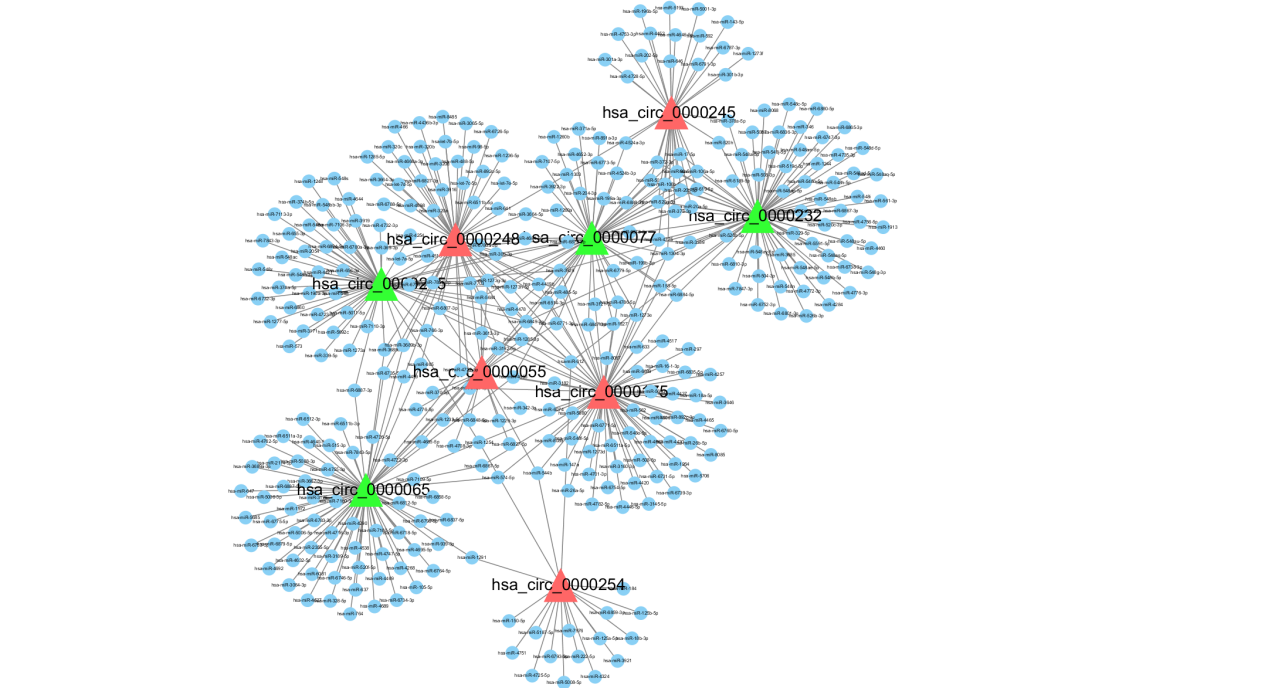
这样的图看起来也还可以,但是如果想要把图形风格进行调整,弄得跟开篇的图一样的,可以通过Layout选项卡进行改变。
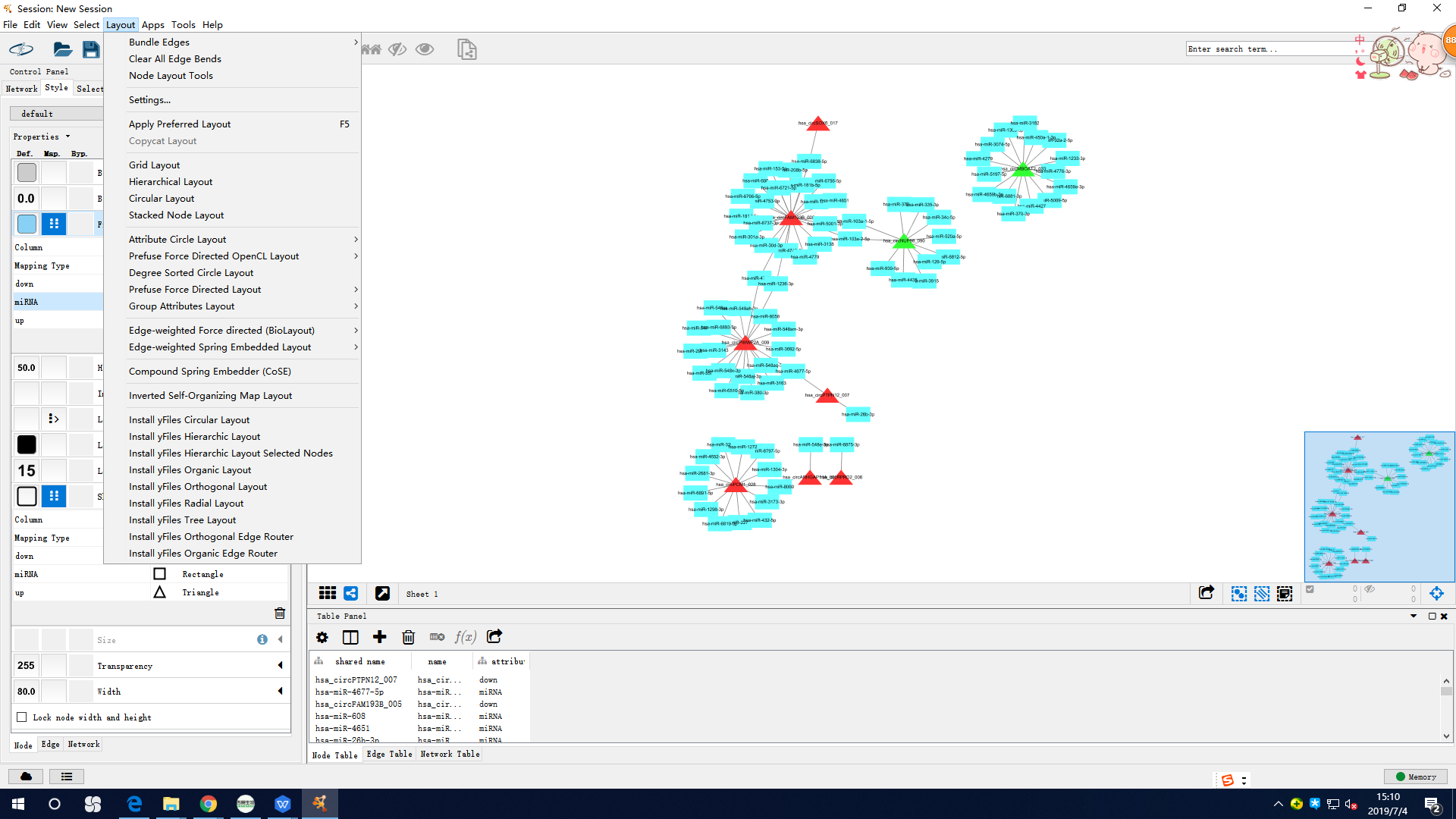
最后得到一个漂亮的circRNA-miRNA网络图:
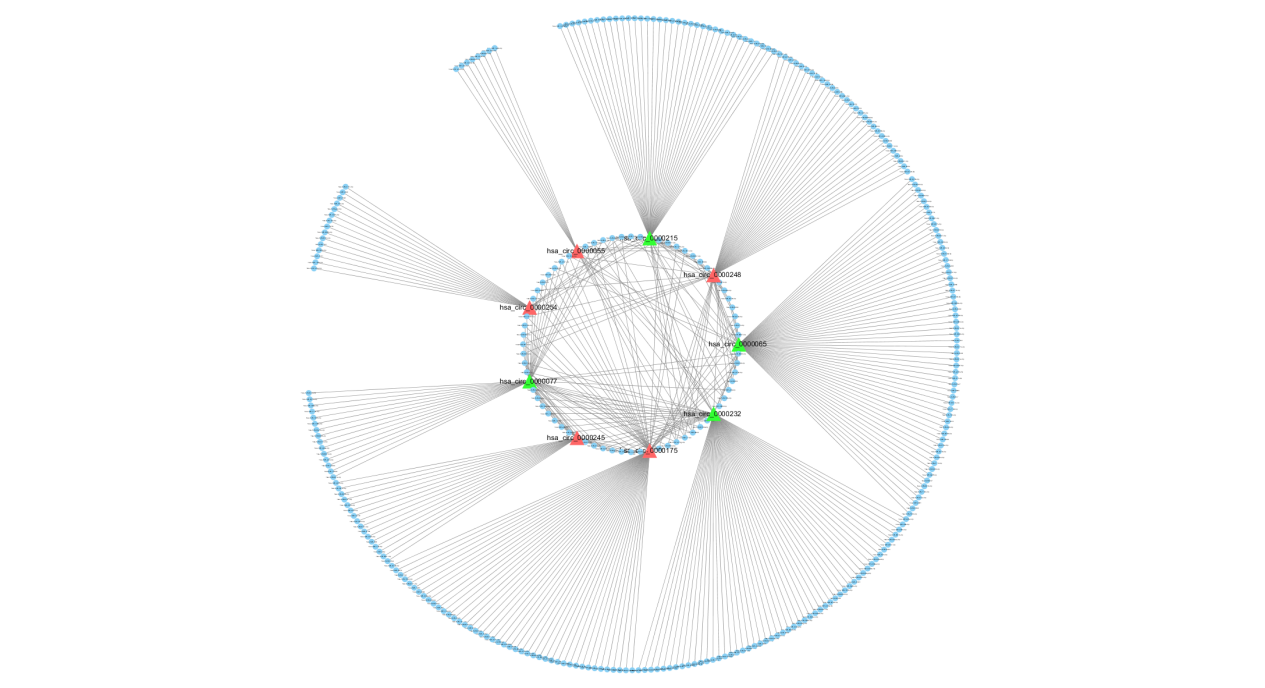
上面就给大家简单介绍了Cytoscape的部分基础功能,主要通过Edges连接Nodes构成Network,通过导入Table对Network中的Nodes进行注释,从而得到我们想要的效果。当然miRNA-mRNA网络图,蛋白-蛋白相互作用网络图(PPI),也可以通过该软件进行可视化处理。如果想研究比较热门的circRNA-miRNA-mRNA 竞争性网络的,也可以把预测到的靶向关系画成网络图。再根据ceRNA竞争性网络的原理,miRNA与靶向ceRNA间表达量负相关,以及ceRNA对之间表达量的正相关的关系;结合实验测到的结果,删去不符合ceRNA竞争性网络的原理的关系,完善circRNA-miRNA-mRNA网络。
另外,Cytoscape软件还有一些小插件,可以构建更庞大更明确的网络图,这个软件远远不止本文中介绍的功能。学海无涯,生信无边,期待与您一起探索。
参考文献:
[1] Xingjie Bao , Shuying Zheng , Shuqi Mao , Tianlun Gu , Shike Liu , Jihan Sun , Lina Zhang.A potential risk factor of essential hypertension in case-control study_ Circular RNA hsa_circ_0037911. Biochemical and Biophysical Research Communications, March 2018; DOI:10.1016/j.bbrc.2018.03.059.
[2] CHUN FENG, YUXIAO LI, YAN LIN, XIANBAO CAO, DONGDONG LI, HONGLEI ZHANG and XIAOGUANG HE. CircRNA-associated ceRNA network reveals ErbB and Hippo signaling pathways in hypopharyngeal cancer. INTERNATIONAL JOURNAL OF MOlecular medicine 43: 127-142, 2019; DOI: 10.3892/ijmm.2018.3942.
.png)
