食道癌是一种全球性的致命癌症,5年生存率为18%[1]。食道癌的发病率、死亡率和组织病理学在不同的地理区域存在差异。腺癌和鳞癌是两种主要的组织学亚型,占食道癌的95%以上。在过去的几十年里,对这种癌症的治疗研究已经有了一些进展。目前,针对食道癌的现状,还需开发新的治疗方法。
环状RNAs(CircRNAs)作为内源性ncRNAs的一个亚类,在肿瘤的细胞周期、凋亡、增殖、侵袭和迁移中发挥着重要的调节作用。它们是由pre-mRNAs的反向剪接产生的,形成一个没有5‘-帽子或3’- poly(A)尾的共价闭合环[2],由于它们广泛表达,相对较高的稳定性,以及在唾液、血液中大量存在,因此有可能成为癌症诊断和预后的生物标志物。
2022年7月15日,宁波市医疗中心李惠利医院心胸外科沈韦羽教授与南昌大学第一附属医院心胸外科喻本桐教授共同通讯在Molecular Cancer发表文章CircBCAR3 accelerates esophageal cancer tumorigenesis and metastasis via sponging miR-27a-3p,研究结果表明,剪接因子QKI促进了CircBCAR3的生物发生,从而通过与miR-27a-3p结合上调TNPO1来加速食道癌的发生,提示CircBCAR3是食道癌治疗的潜在靶点。

首先,作者为了了解CircBCAR3在食管癌中的表达,从GEO数据库中分析了CircRNAs在食管癌中的表达,分析中发现有五个CircRNA在食道癌中差异表达(图1)。为了探讨这些异常表达的CircRNAs在低氧条件下能否在食管癌中发生改变,作者检测了低氧处理后EC109细胞中这些CircRNA的表达水平,发现hsa_circ_0007624(CircBCAR3)在低氧处理后表达上调。随后在EC109和KYSE150细胞中进行了RNA-FISH,表明CircBCAR3主要存在于食道癌细胞的细胞质中。随后,为了研究CircBCAR3在食道癌发生发展中的作用,设计了两个shRNA来沉默EC109和KYSE150细胞。CCK-8、克隆形成试验和EDU试验表明,沉默CircBCAR3抑制EC109和KYSE150细胞的活力和增殖。划痕试验和迁移和侵袭试验表明,CircBCAR3基因的敲除抑制了食道癌细胞的迁移和侵袭能力。
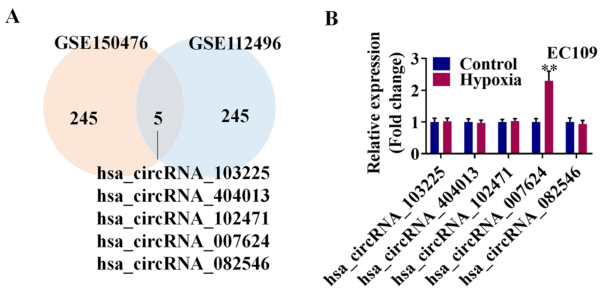
图1. 基于GSE150476和GSE112496数据库的食道癌CircRNA差异表达基因
荧光素酶测定结果表明,CircBCAR3对野生型miR-27a-3p具有负调控作用,与突变体miR-27A-3p没有结合关系。RIP实验表明,作为miRNA机制的效应分子,Ago2能与miR-27A-3p和CircBCAR3结合。RNA-FISH测定结果表明CircBCAR3和miR-27a-3p都位于细胞质中(图3)。
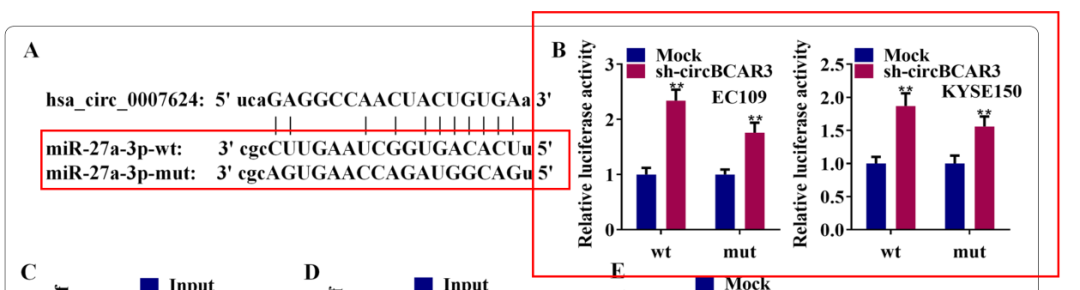
图2.荧光素酶报告实验揭示miR-27a-3p与CircBCAR3的结合
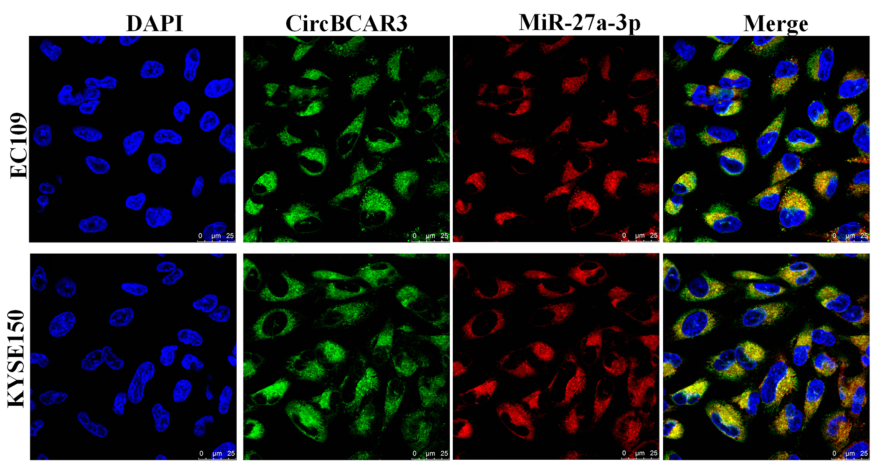
图3. miR-27a-3p和CircBCAR3在EC109和KYSE150细胞中的表达和亚细胞定位
由于目前尚不清楚低氧是如何诱导CircBCAR3表达上调的。作者进行了RNA-seq来筛选异常基因,在其中发现E2F7在食道癌中显著高表达(图4)。PCR分析证实,缺氧处理后EC109细胞中E2F7和QKI均上调。由于E2F7是一个转录激活因子,推测E2F7可能调节QKI的转录。检索GEPIA数据库,检查这些关键分子在182例食管癌组织中的表达相关性,结果显示E2F7与QKI、E2F7与TNPO1、QKI与TNPO1呈正相关。
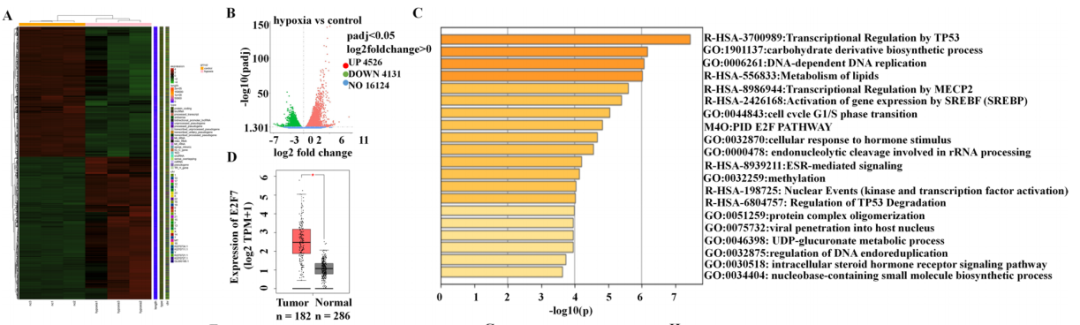 图4.热图与火山图显示低氧处理对食道癌细胞基因表达的影响与GO富集分析
图4.热图与火山图显示低氧处理对食道癌细胞基因表达的影响与GO富集分析
综上所述,低氧诱导的E2F7转录激活剪接因子QKI,通过与BCAR3形成外显子两侧的内含子QKI反应元件结合,促进CircBCAR3的生物发生。CircBCAR3与miR-27A-3p相互作用上调TNPO1,从而在食道癌细胞中发挥其生物学功能(图5)。这些数据表明,CircBCAR3可作为食道癌研究和治疗的潜在标记物。
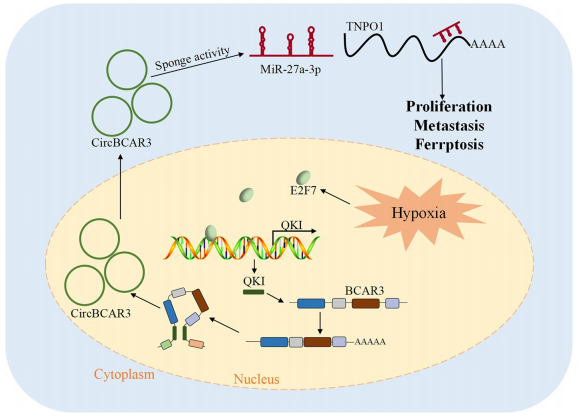
图5.CircBCAR3在食道癌中的作用机制
原文链接:
https://molecular-cancer.biomedcentral.com/articles/10.1186/s12943-022-01615-8
参考文献:
[1] F.L. Huang, S.J. Yu, Esophageal cancer: Risk factors, genetic association, and treatment, Asian journal of surgery 41(3) (2018) 210-215.
[2] C. Huang, G. Shan, What happens at or after transcription: Insights into circRNA biogenesis and function, Transcription 6(4) (2015) 61-4.
转载请联系邮箱授权:circRNA@163.com
.png)
