01 一代测序
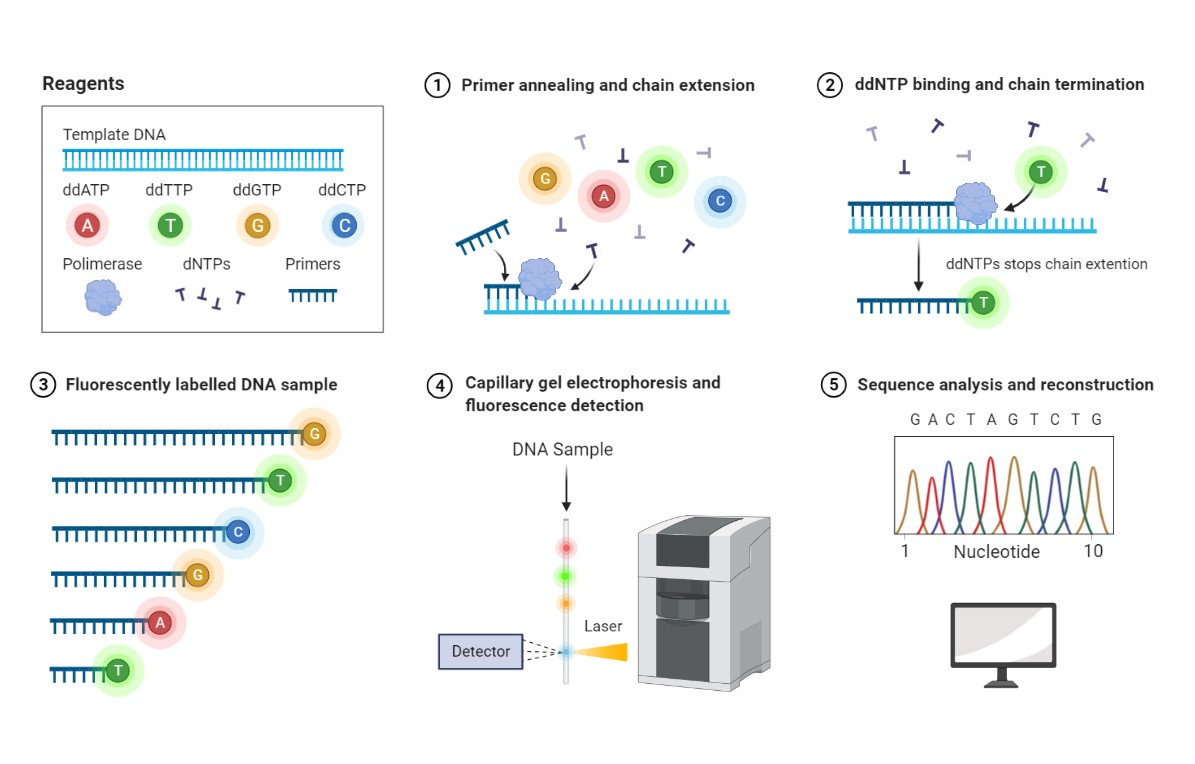
Sanger Method又称为双脱氧链终止法,其名字直观地阐述了该测序方法的核心:根据模板在引物上逐个添加ddNTP。
在circRNA研究中,sanger测序主要搭配RT-PCR对目标circRNA进行验证,主要包括以下几种方式:
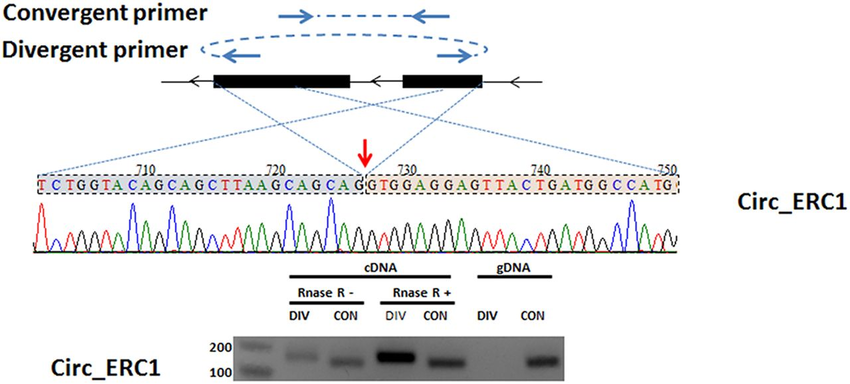
1-1 Divergent Primer检测BSJ
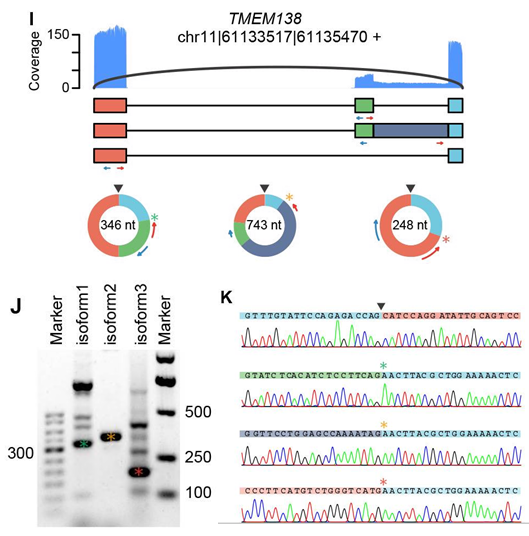
1-2 不同策略鉴定circRNA全长
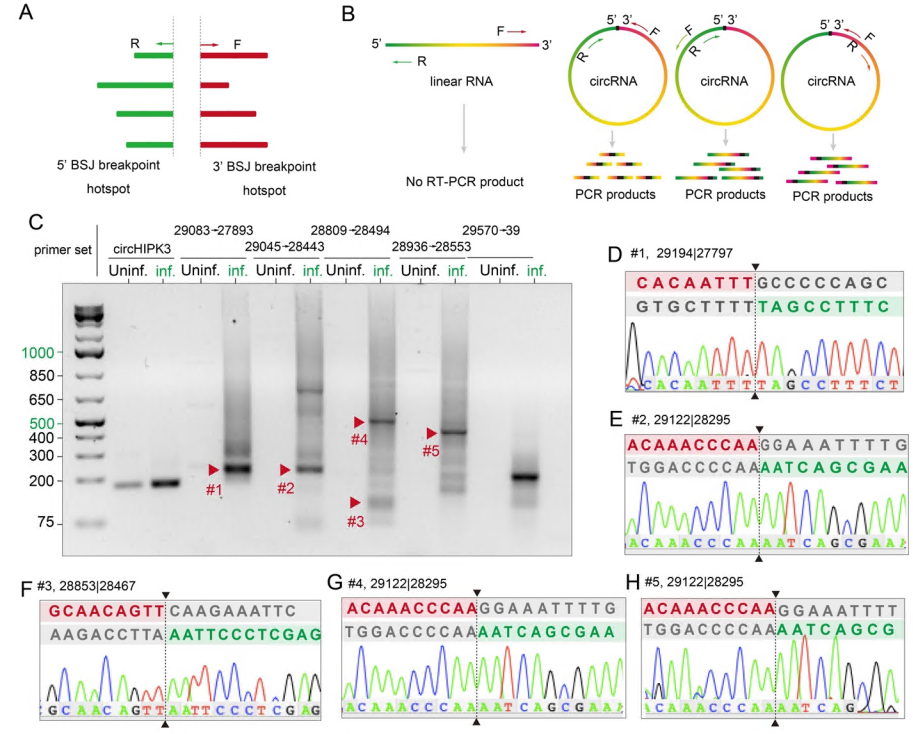
②拼接多组primers

③一步到位的滚环PCR
在我们对环状RNA验证时,一般只对“环状RNA特定标识BSJ”进行验证;然而大量证据已表明环状RNA BSJ内部存在着复杂的剪切结构,因此,虽然有相同BSJ,但具体是哪一个isoform发挥功能,仍需要对isoform的全长进行验证。
02 二代测序
随着人类基因组计划的完成,二代测序技术也迎来了它的辉煌时代:2005年Roche 454测序系统发布标志着测序技术正式跨入高通量测序时代。早期二代测序技术手段可谓百花齐放,但被大家熟知的测序仪包括Roche 454、Illumina Solexa以及ABI SOLID;然而短短几年,二代测序市场就基本被Illumina测序仪一家独大,国内市场早已被Illumina测序产品主导;直到2015年,我国终于迎来自主研发的首款测序仪BGISEQ-500,实现了国产测序“从0到1”的突破。
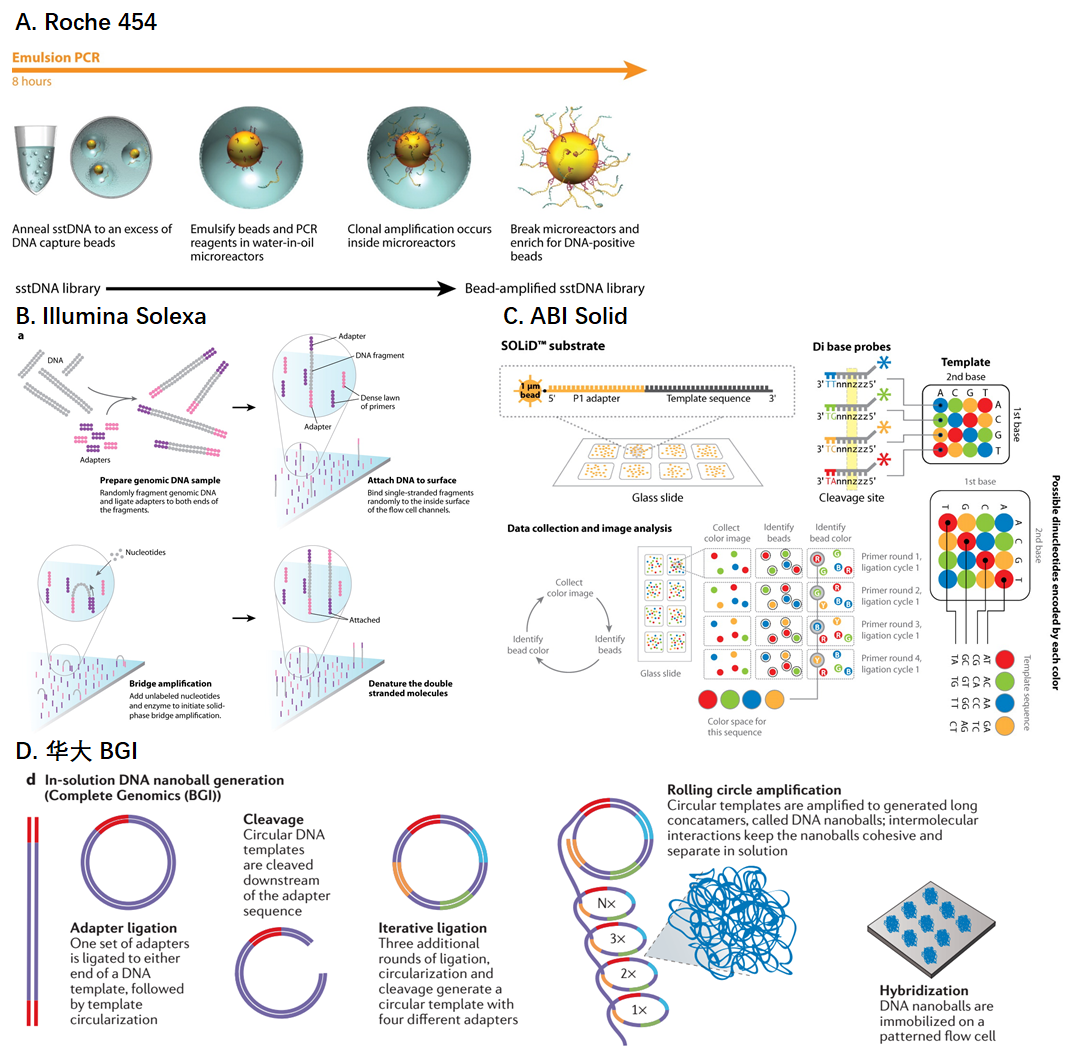
Roche 454乳液PCR技术、Illumina Solid桥式 PCR、ABI SOLID双碱基四色荧光以及华大BGI的DNB技术
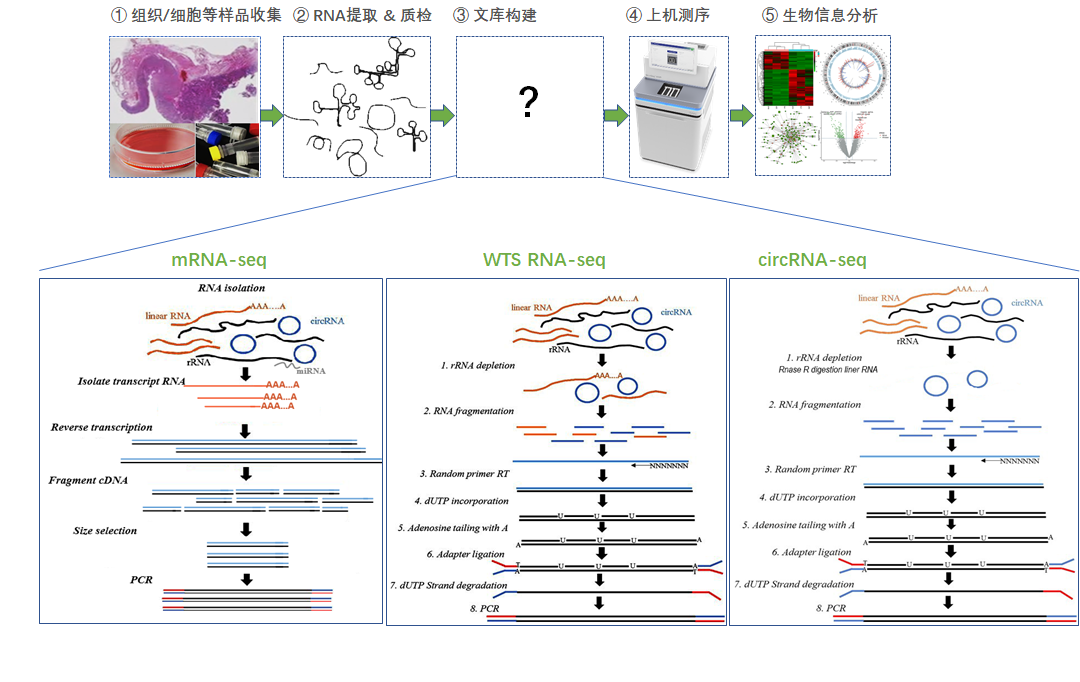
mRNA-seq、全转录组RNA-seq以及circRNA-seq的不同建库方式
2-1 环状RNA的检测
• rRNA-deleted library
能够同时检测mRNA,lncRNA等线性RNA以及circRNA。
• rRNA-deleted, RNase R + library
虽然只能检测circRNAs,但通常能够检测到更多circRNA,特别是一些表达丰度较低的circRNA。如果想同时研究circRNA与mRNA的关联,可以对同一批起始RNA进行mRNA-seq。
为了使circRNA更富集,我们也可以先通过加A的方式让线性RNA的3′ 末端更突出(RNase R只能结合到3’单链末端至少包含7个碱基的分子),从而让线性RNA被RNase R消化得更充分。
测序后我们可以开展一系列与circRNA相关的生物信息学分析,而这一切都要源于对back-spliced junction(BSJ)——RNA是否是环状的唯一标识——的理解和识别。
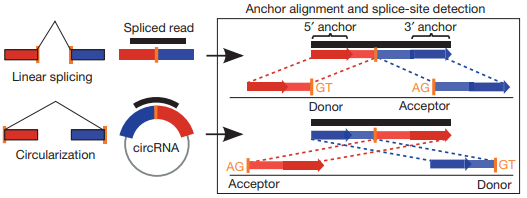
forward splicing(即Linear splicing) 与 back splicing(即circularization)
识别BSJ是生信软件鉴定环状RNA的核心,然后不同软件会包含一些其他的设置来保证环状RNA的真实性,例如reads在back-spliced site前后最小长度、BSJ侧翼序列是否存在剪切位点、BSJ内部序列长度是否超过某个阈值等。
从2012年到现在,已经有许多经典的鉴定与定量软件被我们熟知,例如find_circ、CIRI、DCC、CIRCexplorer等,这些软件各有各的优点但也有不足(主要是假阳性与敏感性的权衡),因此研究者往往通过联合多款软件对环状RNA进行识别与表达定量。现在虽然已经过了环状RNA鉴定方法开发的喷涌期,但由于不同的实验和技术存在偏倚性,直到现在仍有一些新的软件涌现,例如CARP(Li et.al, 2022)、CiLiQuant(Celine et.al, 2022)。
2-2 环状RNA的多组学研究
下面简单阐述了不同的组学技术对环状 RNA 的研究。
虽说翻译调控(translation regulation)所包含的程序超过了其他调控的总和(Schwanhausser et al., 2011),但我们仍困在哪些环状RNA能够翻译,环状RNA翻译的研究真的是“路漫漫其修远兮”。
当然,现在的项目研究所用到的技术都不是孤立的,我们往往需要整合多组学的技术联合对所观察到的现象进行剖析,例如circRNA m6A研究往往都需要结合RNA-seq,scRNA-seq往往需bulk RNA-seq。只有掌握了这些测序技术的基本原理,我们才能在研究中更游刃有余。
2-3 二代测序的痛点
虽然听起来让人诧异——不是已经有这么多软件能够分析circRNA了吗——这些环状RNA识别与定量也仅仅是通过算法推测,假阳性率甚至达到了45%(Dodbele et.al, 2021)。
为什么这么说呢?主要原因有两点:
• 二代测序固有的缺陷
二代测序由于DNA聚合酶活性等问题,只能检测较短的片段(例如最常用的PE150),因此需要事先对RNA分子进行片段化处理,这一措施很可能导致“circRNA唯一标识BSJ”丢失。
• 环状RNA自身的特征
绝大部分环状RNA表达丰度是极低,因此,在碎片化的RNA文库中circRNA的信号很容易就被掩盖了。
因此,二代测序对环状RNA进行检测一般都需要比mRNA更高的RNA建库起始量,从而保证BSJ分子能够被保留;同时,生信算法也需要足够敏感,但同时又要尽可能地排除假阳性。
二代测序研究环状RNA另一个缺陷是难以检测到环状RNA的中间序列,而大量研究都表明环状RNA存在着复杂的中间结构。为了缓解这一状况,许多根据RNA-seq数据预测环状RNA中间结构的算法被提出,例如CIRCexplorer2、CIRI-full、CircAST等等。
然而,尽管研究人员通过计算方法一定程度提高了二代测序检测环状RNA的准确性,但二代测序固有的局限性以及环状RNA结构的复杂性让我们需要更适合的技术对环状RNA更进一步的刻画。
03 三代测序
在2011年和2014年PacBio以及Oxford Nanopore分别发布了基于聚合酶和基于电信号的商用测序仪。随着技术的成熟和测序价格的下降,在2015年的时候You等研究者就开始使用PacBio检测环状RNA,虽然当时仅仅是12个二代测序来源的环状RNA进行了验证;在2017年Hirsch等人通过Nanopore对circNPM1的剪接异构体的序列组成进行解析;直到2019年,Rahimi等人才通过Nanopore对环状RNA进行了大规模的测序(发表在bioRxiv预印本),不过它采用片段化试剂盒“破环”的方式仍然可能导致环状RNA序列的不完整。
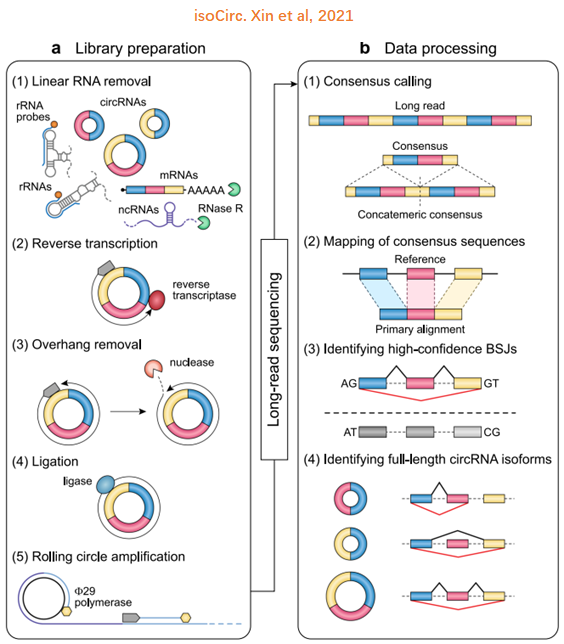 isoCirc通过rRNA-deleted、RNase R对circRNA进行富集,随后用随机引物对circRNA进行反转录,如果反转录后的产物超出了环的长度就用酶进行消化,随后反转录产物被连接成环,最后进行滚环扩增(RCA)。
isoCirc通过rRNA-deleted、RNase R对circRNA进行富集,随后用随机引物对circRNA进行反转录,如果反转录后的产物超出了环的长度就用酶进行消化,随后反转录产物被连接成环,最后进行滚环扩增(RCA)。
随后将RCA产物进行Nanopore测序。生信分析鉴别circRNA主要包括滚环一致序列(rolling consesus sequence)以及BSJ识别两个步骤。
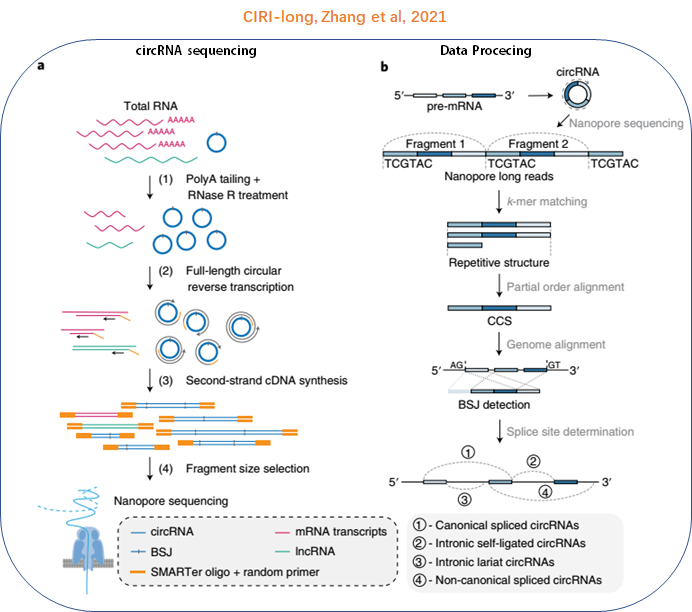 CIRI-long通过rRNA-deleted、poly(A) tailing和RNase R充分去除线性分子从而富集circRNA,随后用随机引物进行滚环反转录,再合成2nd链cDNA后进行了最优测序片段的筛选。
CIRI-long通过rRNA-deleted、poly(A) tailing和RNase R充分去除线性分子从而富集circRNA,随后用随机引物进行滚环反转录,再合成2nd链cDNA后进行了最优测序片段的筛选。
Nanopore测序后,用CIRI-long分析策略进行数据分析,分析的关键步骤包括滚环一致序列(cyclic consesus sequence)以及BSJ识别两个步骤。
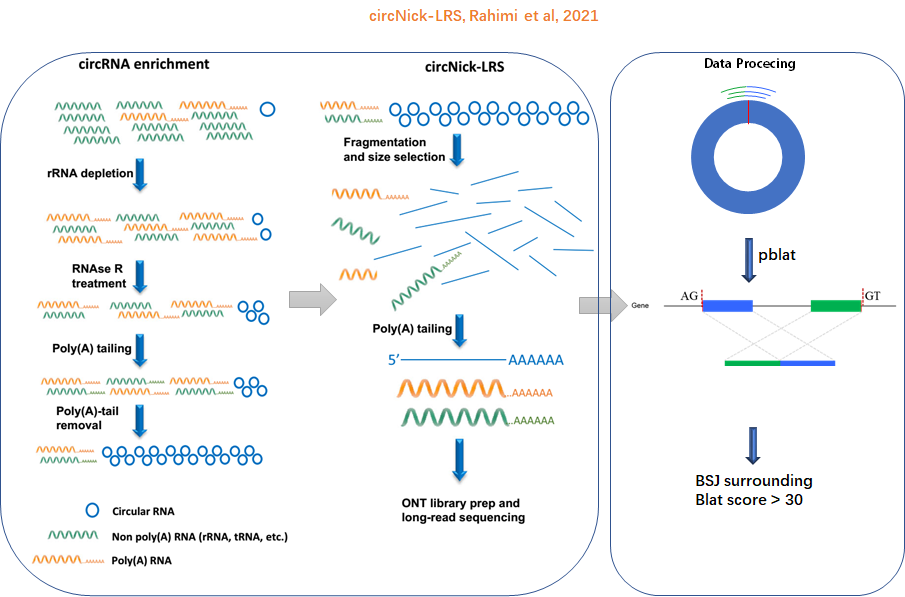 circNick-LRS通过rRNA-deleted、poly(A) tailing和RNase R充分富集环状分子,随后通过“破环”将环状RNA线性化并添加 poly(A) 尾。
circNick-LRS通过rRNA-deleted、poly(A) tailing和RNase R充分富集环状分子,随后通过“破环”将环状RNA线性化并添加 poly(A) 尾。
Nanopore 测序后通过pblat查看测得的序列是否包含BSJ位点。
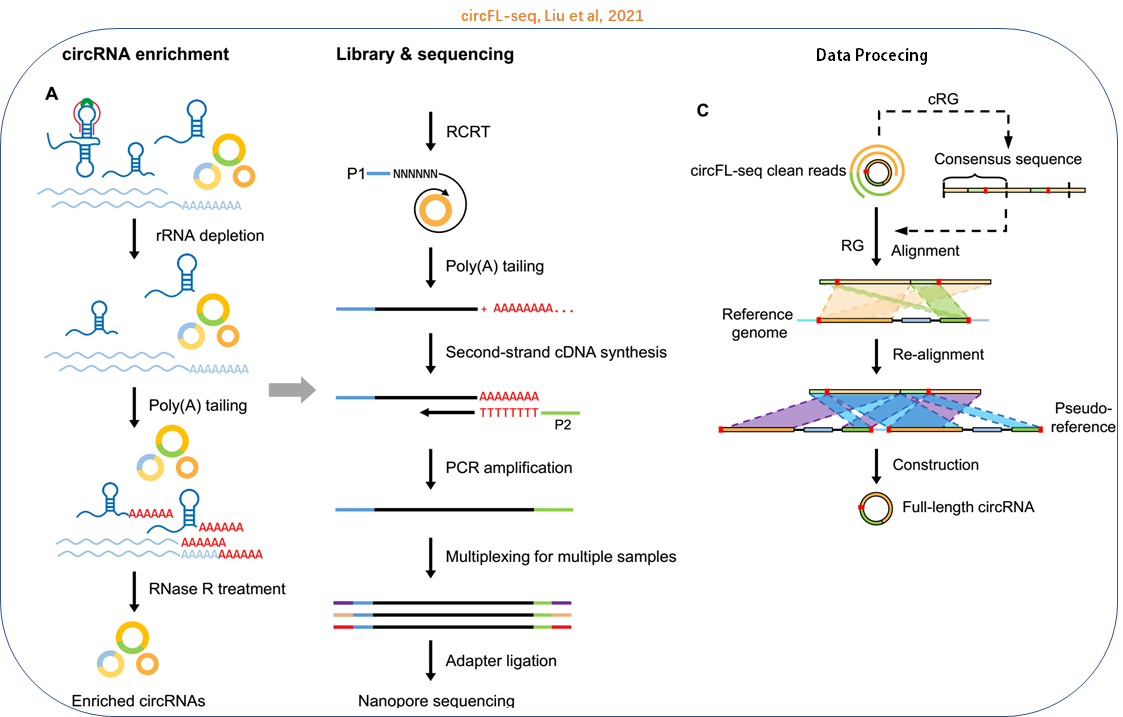 circFL-seq采用的仍然是rRNA-deleted、poly(A) tailing和RNase R富集环状分子的方法,随后用滚环反转录将环状RNA线性化建库。
circFL-seq采用的仍然是rRNA-deleted、poly(A) tailing和RNase R富集环状分子的方法,随后用滚环反转录将环状RNA线性化建库。
测序后生信分析方面,circFL-seq提供了包括序列直接进行参考基因组比对(RG)以及通过CCS进行参考基因组比对(cRG)两种方式寻找潜在circRNA。
Illumina在二代测序市场虽然占有绝对的统治地位,但面对测序市场向长读长测序的急速转变,它急需在该领域做出突破。另一方面,Illumina的一些核心专利(例如“修饰核苷酸”和“改良核苷酸”)到期或即将到期,早已被虎视眈眈的二代测序市场将被瓜分(例如华大制造已经发布了多款DNBSEQ产品在美上市的计划),这也迫使Illumina需要在新的领域有所作为。
因此,Illumina在2018年收购三代测序界的翘楚PacBio无果后,在今年1月推出了长读长技术Infinity。9月29日,首届Illumina基因组学论坛宣称Illumina三代测序产品Complete Long-Reads(曾用名Infinity)目前正处于抢先体验阶段,将于2023年正式推出。
当然,“尺有所短,寸有所长”,无论是一代、二代还是三代,它们都有着各自的优势领域,不同的科研问题用不同的技术,而我们只需要按需所取就可以了!
.png)
